目录
一、摘要
我们开发了一种序列对序列的方法seq2rel,它可以学习DocRE(实体提取、共同引用解析和关系提取)的端到端子任务,取代了任务特定组件的管道。我们使用一种称为实体暗示的简单策略,将我们的方法与几种流行的生物医学数据集上现有的基于管道的方法进行比较,在某些情况下性能超过了它们。
二、引言
1、PubMed数据集
它的任务是在一些文本中识别参与语义关系的实体组
2、贡献
- 我们提出了一个新的线性化模式,可以处理以前的seq2seq方法忽略的复杂性,比如coreferent提到和n-ary关系(§3.1)。
- 使用这种线性化模式,我们证明了seq2seq方法能够共同学习DocRE的子任务(实体提取、共同参考解析和关系提取),并报告了几个流行的生物医学数据集上的第一个端到端结果(§5.1)。
- 我们设计了一个简单的策略,称为“实体暗示”(§3.3),以比较我们的模型与现有的基于管道的方法,在某些情况下超过它们的性能(§5.1)。
三、任务定义
在本文中,我们主要关注的情况是,E没有给出,必须由一个模型预测,我们将其称为“端到端”。
四、我们提出来的模型: seq2rel
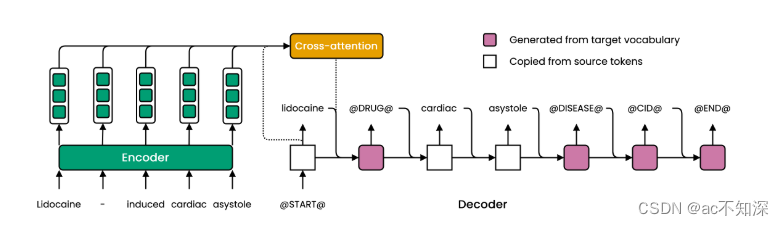
4.1 线性层
一个分号分隔共同引用提及,实体以一个表示其类型的特殊标记结束(例如@GENE@)。类似地,关系以表示其类型的特殊标记(例如@GDA@)终止。在特殊关系令牌之前可以包含两个或多个实体,以支持n元提取。如果实体充当关系的头或尾的特定角色,则可以对它们进行排序。
4.2 模型
4.2.1 模型简介
编码器将输入中的每个标记映射到上下文嵌入。自回归解码器生成一个一个标记的输出,关注编码器在每个时间步的输出(图2)。解码一直进行,直到生成一个特殊的“序列结束”标记(@END@),或生成了最大数量的标记。
形式上,X是长度为S的源序列,它是一些我们想要从中提取关系的文本。Y是对应的长度为T的目标序列,是源中包含的关系的线性化。
4.2.2 主要问题
- 通过生成源文本中没有出现的实体提及,模型可能会产生“幻觉”。
- 它可能生成一个不遵循线性化模式的目标字符串,因此不能被解析。
4.2.3 解决方法
a)解决1
为了解决1),我们使用了两种修改:受限的目标词汇(§3.2.1)和复制机制(§3.2.2)。
限制目标词汇
为了防止模型产生“幻觉”(生成源文本中没有出现的实体提及),目标词汇表被限制为建模实体和关系所需的一组特殊令牌(例如;和@DRUG@)。所有其他令牌必须使用复制机制从输入中复制(参见§3.2.2)。这些特殊标记的嵌入是随机初始化的,并与模型的其余参数一起学习。
复制机制
为了在解码期间启用输入标记的复制,我们使用了复制机制(Gu等人,2016a)。该机制通过使用源序列X中的标记有效地扩展目标词汇表,允许模型将这些标记“复制”到输出序列Y中。我们对复制机制的使用类似于以前基于关系抽取的seq2seq方法
b)解决2
为了解决2),我们在解码过程中使用了几个约束(§3.2.3)。
限制decoder
我们在测试期间对解码器应用了一些约束,以减少生成语法无效的目标字符串(不遵循线性化模式的字符串)的可能性。通过在每个时间步将无效标记的预测概率设置为一个极小的值来应用这些约束。在实践中,我们发现一个训练过的模型很少会产生无效的目标字符串,所以这些约束对最终性能的影响很小(见§5.3)。我们决定在剩下的实验中不应用它们。
c)解决3
为了解决3),我们根据源文本中出现的顺序对关系进行排序(§3.2.4)。
关系排序
从给定文档中提取的关系本质上是无序的。然而,序列交叉熵损失(式2)相对于预测的标记是排列敏感的。在训练过程中,这强制了一个不必要的解码顺序,并可能使模型容易在训练集中过拟合频繁标记组合(Vinyalset al., 2016;Yang等人,2019)。为了部分缓解这一问题,我们根据源文本中出现的顺序对目标字符串中的关系进行排序,从而为模型提供一致的解码顺序。关系的位置由第一次出现的对其头部实体的提及决定。提及的位置由其开始和结束字符偏移量的总和决定。对于联结,我们将根据第一次提到的尾部实体进行排序(对于n-ary关系也是如此)。
4.3 实体暗示
还提供实体作为输入,使用一种简单的策略,我们将其称为“实体暗示”。这涉及到将出现在目标字符串中的实体添加到源文本的前面。其中特殊的@SEP@标记划定了实体提示的结尾。
注:一些预先训练的模型有自己的分隔符,可以用来代替@SEP@,例如BERT使用[SEP]。
五、实验设置
5.1 数据集
CDR
BioCreative V CDR任务语料库手动注释化学品、疾病和化学品诱发疾病(CID)关系。它包含1500篇PubMed文章的标题和摘要,并被分成同等大小的训练、验证和测试集。鉴于训练集的规模相对较小,我们跟随Christopoulou等人(2019)和其他人,首先在验证集上调整模型,然后在测试集上进行评估之前,对训练集和验证集的组合进行训练。与之前的工作类似,我们过滤与疾病实体的负关系,这些疾病实体是同一摘要中对应的真实关系疾病实体的上位关系
GDA
基因-疾病关联语料库包含PubMed文章的30192个标题和摘要,这些文章通过远程监控自动标记出基因、疾病和基因-疾病关联。测试集由1000个这样的示例组成。在Christopoulou等人(2019)和其他人之后,我们随机拿出剩余20%的摘要作为验证集,并将其余的用于训练。
DGM
药物-基因突变语料库包含4606篇PubMed文章,这些文章通过远程监督自动标记药物、基因、突变以及药物-基因突变三元关系。数据集有三种变体:句子、段落和文档长度文本。我们根据段落长度的输入来训练和评估我们的模型。因为测试集不包含段落级别的关系注释,所以我们报告验证集的结果。我们随机拿出20%的训练示例,形成用于调优的新验证集
DOCRED
DocRED包括超过5000个来自维基百科的人类注释文档。共有6个实体和96种关系类型,其中超过句子边界的关系占40%。我们使用与以前的端到端方法(Eberts和Ulges, 2021)相同的分割方式,在训练集中有3,008个文档,在验证集中有300个文档,在测试集中有700个文档。
六、结果
6.1与现有方法比较
6.1.1 现存的base方法
当在GDA上使用实体提示时,它的性能优于现有的基于管道的方法。
当在CDR语料库上使用实体提示时,我们的方法与现有的方法相比具有竞争力,但最终不如最先进的结果。
在所有情况下,除了具有实体提示的GDA,性能都随数据集的大小单调增加。即使使用全部500个训练例,CDR也没有明显的平台。总之,这些结果表明,当有足够的训练示例时,我们基于seq2seq的方法可以优于现有的基于管道的方法,但在低数据状态下,相对于现有的方法,其性能要差一些。
6.1.2 n维关系抽取
我们在DGM语料库上的结果表明,我们的线性化模式有效地模拟了n元关系,而不需要改变模型架构或训练过程。
6.1.3 端到端的方法
DocRED与JEREX比较,前者性能略低,主要是由于召回。我们推测,这其中的一个原因是每个文档有大量的关系,这导致了更长的目标字符串,因此需要更多的解码步骤。
a)DocRED得分略低的原因
DocRED中目标字符串的中位数长度是110,而在GDA中第二大长度是19。
解决方法:提高解码器处理长序列的能力,例如将LSTM转换为变压器或修改线性化模式以产生更短的目标字符串,可以提高召回率并缩小与现有方法的差距。
6.2 现有方法VS端到端
- 证明了联合学习实体和关系提取的好处证明了联合学习实体和关系提取的好处
- 使用黄金实体提示明显优于所有其他设置。因为黄金标准实体注释是由领域专家生成的高质量标签。使用银色提示会显著降低性能。
6.3 消融实验
有意地对每个目标字符串中的关系排序(-排序关系,见§3.2.4)有很大的积极影响,将性能提高5.6%14.7%。
有意地对每个目标字符串中的关系排序(-排序关系,见§3.2.4)有很大的积极影响,将性能提高5.6%14.7%。
七、一些讨论
1、未来展望
- 增加模型的输入长度限制(默认长度为512)
- decoder一般是从头开始训练的,可以考虑对decoder部分进行预训练























 378
378











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








