2024年10月31日更新wrl转stl和xyz
【wrl2stl】WRL文件转STL文件-Python-CSDN博客
Wrl文件转XYZ文件-Python_python 打开wrl三维模型-CSDN博客
颗粒CT扫描数据使用Avizo重建后,需要将每个颗粒单独保存为一个文件,再进行后续的分析。前几部都可参考以下视频。
【Avizo Amira培训练习】 孔隙(或颗粒)分析 Colorize by Measure_哔哩哔哩_bilibili
每个颗粒单独分割后,软件内不支持批量将分割结果导出,只能通过python脚本将每个颗粒导出。因为python调用的是左侧project,因此只能导出VRWL文件,再通过python转为stl\obj格式。
需要先按照如下路径分割颗粒

python脚本自动化操作的是上述红框内的部分,起点是pmma1-483(3).label,通过修改Interactive Thresholding(2)中的intensityRange来选择要保存的颗粒。
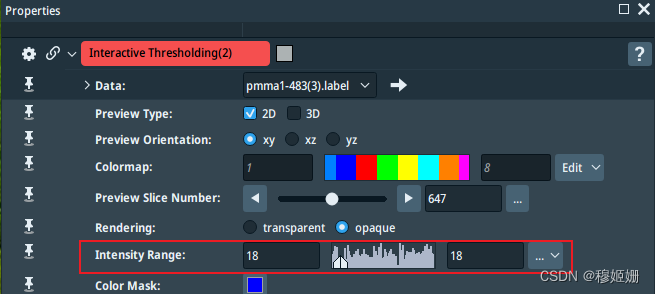
要注意的是Generate Surface、Smooth Surface都要勾选auto-refresh,可以保证Interactive Thresholding(2)修改后自动更新。Interactive Thresholding(2)和Export to VRML不需要勾选auto-refresh,因为已经在代码中分别加入了thresh.execute()和export.execute()执行“Apply”,二者选其一即可。
特别提醒:在运行python程序前一定要保存!!!程序一旦开始无法暂停!!!只能强制退出!!!可以修改range的范围进行调试!!!确认代码没问题后再执行!!!
num = int(hx_project.get('pmma1-483(3).label').range[1])
for i in range(1, num + 1):
thresh = hx_project.get('Interactive Thresholding(2)')
thresh.ports.intensityRange.range=(i,i)
thresh.execute()
export = hx_project.get('Export to VRML')
export.ports.filename.filenames = f'D:/fooling_around/PMMAbubble/pmma1_wrl/{str(i).zfill(3)}.wrl'
export.execute()如果弹出警告框,需要手动点击Continue,如果颗粒很多可以使用鼠标连点器……这很工程。

如果出现需要覆盖的情况:
- 首先检查文件夹内是否已存在同名文件,把这些文件删掉或转移走;
- 其次检查“Export to VRML”模块勾选了“auto-refresh”的同时,代码中也加入了export.execute(),二者选其一即可。

























 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








