前言
由于大三狗最近决定跟老师做项目,而老师给的第一个任务就是完成这道题,为了避免拖延症,决定翻译这玩意督促自己,肯定多有错漏之处,万望海涵。
本文的诞生要感谢我小学到大学的所有英语老师,谷歌翻译以及声时刻汉化的Graz datasetB,有许多地方感觉不通顺的都会看看声时刻大佬的翻译再润色一下。
附上文档链接:
http://bbci.de/competition/iv/desc_2a.pdf
声时刻大佬的datasetB 翻译帖子:
https://blog.csdn.net/shenshikexmu/article/details/51251769
正文

实验模型
这个数据集包括对9位实验者进行试验所得到的EEG(脑电)数据。每个带有提示的BCI(脑机接口)测试例子都包括对应四类不同的想象任务的运动图像,即左手运动图像(类1),右手(类2),双脚(类3),还有舌头(类4)。每个受试者都参加了两次在不同日期进行的session。每个session都由6次run组成,每次run都由短暂的休息间隔开。一次run包括48次测试(每个类测试12次),每个session包括288次测试。
每次session开始的时候,都会进行播放大约5分钟的影像,以评估EOG(眼电)数据的对EEG(脑电)信号的影响,影像被分成3部分:(1)睁眼两分钟(注视屏幕上固定的十字)(2)闭眼一分钟以及(3)进行一分钟的眼球运动。每次session的时序图如Figure 1所示,需要注意的是由于技术问题,4号受试者(A04T)的EOG(眼电)数据块更短,并且只包含眼球运动的测试条件(每位受试者列表请见Table 1)
测试时每个受试者都坐在一个舒适的扶椅上,面前是电脑屏幕。测试开始时(t=0s),一个固定的十字会出现在黑色的屏幕上。此外还有简短的声音提示音。两秒过后(t=2s),一个作为提示指向左、右、下或者上(对应于四个类别左手运动、右手运动,双脚运动以及舌头运动)的箭头会出现在屏幕上约1.25s。这促使受试者想象与图片对应的运动,没有提供任何反馈。每个受试者要求完成这些想象任务直到屏幕上的十字消失(t=6s)。短暂休息知道屏幕再次变黑。该测试例子详见Figure 2

数据记录
22个Ag/AgCl电极(电极间距离为3.5cm)被用于记录EEG(脑电)信号;Montage图展示在Figure 3左侧,所有信号均用电极记录,左边电极作为参考量,右边电极作为地。信号通过250Hz采样后通过0.5Hz到100Hz范围的带通滤波器。放大器灵敏度设置为100uV,一个50Hz的陷波滤波器被用于滤除噪声。

相对于22个EEG采样电极,3个EOG采样电极用于记录并进行250Hz采样,(见Figure 3右图)。这些信号经过0.5Hz到100Hz带通滤波器(同样开启了50Hz的陷波滤波器),放大器灵敏度设置为1mV。EOG采样是为了想象任务之后进行伪迹处理操作,没有必要进行分类。
每个数据集都由专家进行了视觉检查,并标记了包含伪迹的试验。8/9个数据集在[2,3]中被分析。
数据文件描述
所有数据集都用生物医学的通用数据格式存储(GDF),一个文件对应一个受试者的一次session。但是只有一个session包含了受试者试验的标签,另一个session被用于测试分类器并用于评估效果。所有文件列在Table 1中。注意评估用的数据集将在比赛截止日期后提供(作为示例的受试者A01的数据集除外)。可以使用开源的BioSig工具箱加载GDF文件,该工具箱可以从http://biosig.sourceforge.net/获得。提供的版本包括Octave/FreeMat/Matlab以及用于C/C++的库

GDF文件在Octave/FreeMat/Matlab中可以通过下面的命令加载(对于C/C++,对应的函数 HDRTYPE* sopen 和 size_t sread 必须被声明)

注意每次runs之间通过100缺省值分开,默认为NAN。 或者可以把缺省值存储为负最大值,通过以下语句实现:
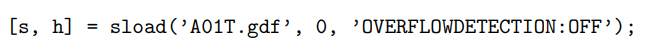
函数返回包含两个变量,信号s以及表头结构h,信号s的值来自25个通道(前面22个EEG通道以及后面3个EOG通道),表头结构包含了事件的相关信息,用于描述数据的结构。以下几个字段提供了这个数据集的重要评估信息

通过h.EVENT.POS可以得到样本中事件的位置,相对应的事件类型可以在h.EVENT.TYP找到,特定的时间区间存储在h.EVENT.DUR中。Table 2描述了数据集中使用的数据类型(十六进制数字,十进制数字标注在括号里)。注意分类标签(如1,2,3,4对应事件类型769,770,771,772)仅提供给训练数据集,不提供给测试数据集

其中包含伪迹的试验的事件类型被专家标注为1023,此外h.ArtifactSelection包含所有试验的目录。0表示没有伪迹的试验,1表示包含伪迹的试验。
为了观察GDF文件,可以使用软件SigViewer v0.2或其更高版本(包含于BioSig中)
评估
参与者要对每一个样本以4类标签的形式提供连续的分类器输出,包括被标记的试验以及被标记为伪迹的试验。对于每个时间点,不存在伪迹的试验可以建立一个混淆矩阵。从这些混淆矩阵得到时间准确度以及Kappa系数。BioSig会提供评估算法,最终Kappa值最大的算法将会获胜。
由于评估数据集直到比赛结束才会下发,提交的程序必须兼容EEG数据(必须使用与训练集中相同的数据结构),并将该EEG数据作为输入,产生前面提到的类标签向量。
因为数据集包括3个EOG通道,需要在后续处理数据前利用伪迹处理技术,如高通滤波或线性回归,移除EOG伪迹。我们选择最大透明度措施并提供给EOG通道,以保证其他校正方法可以正确应用;同时我们要求伪迹不能影响分类效果。
所有的算法都要符合因果性,意味着在k时刻的分类结果,只与当下和过去采样x_k ; x_ k-1,…, x_0有关。为了检测提交算法是否符合因果性和符合伪迹处理要求。所有参赛作品必须开源,包括使用的库,编译器,编程语言,等等(如Octave/FreeMat, C++, Python, . . . )。注意同样可以使用闭源环境MATLAB编写算法并提交,只要代码可以在Octave上运行。类似的,C++程序也可以在Microsoft或Intel环境下编译,但是代码必须可以在g++环境下编译。


附录(Matlab安装BioSig工具箱)
一开始我安装Matlab版本的BioSig工具箱时安装失败,发现Matlab无法通过图形界面SetPath,以为是这个问题,最后发现是因为轮子年久失修,很多函数不兼容的问题,这里提一下修改方案。
BioSig工具箱下载之后会得到下图的目录结构:

需要注意的是biosig文件夹里面有一个install.m文件,和biosig_installer.m文件一模一样,任选一个修改即可。
安装时把NaN文件夹和tsa文件夹放进biosig文件夹中,install.m(或biosig_install.m)放进biosig文件夹,打开install.m文件,进行以下修改。
首先是在大约40行那里,源代码为:
subdirs={'doc','t200_FileAccess','t210_Events','t250_ArtifactPreProcessingQualityControl','t300_FeatureExtraction','t400_Classification','t330_StimFit','t450_MultipleTestStatistic','t490_EvaluationCriteria','t500_Visualization','t501_VisualizeCoupling'};
addpath(sprintf(fullfile(BIOSIG_MATLAB_PATH,'%s:'),subdirs{:}))
修改为以下代码:
subdirs={'\doc','\t200_FileAccess','\t210_Events','\t250_ArtifactPreProcessingQualityControl','\t300_FeatureExtraction','\t400_Classification','\t330_StimFit','\t450_MultipleTestStatistic','\t490_EvaluationCriteria','\t500_Visualization','\t501_VisualizeCoupling'};
for i = 1:length(subdirs)
path([BIOSIG_MATLAB_PATH,subdirs{i}],path);
end
以及未修改前118行处:
try
x = mod(1:10,3)'-1;
[Pxx,f] = periodogram(x, [], 10, 100);
[b,a] = butter(5, [.08,.096]);
catch
path(path,[BIOSIG_MATLAB_PATH,'/freetb4matlab/signal'],'-end'); % Octave-Forge signal processing toolbox converted with freetb4matlab
disp('signal processing toolbox (butter,periodogram) from freetb4matlab added');
end;
将其修改为:
try
x = mod(1:10,3)'-1;
[Pxx,f] = periodogram(x, [], 10, 100);
[b,a] = butter(5, [.08,.096]);
catch
path(path,[BIOSIG_MATLAB_PATH,'/freetb4matlab/signal']); % Octave-Forge signal processing toolbox converted with freetb4matlab
disp('signal processing toolbox (butter,periodogram) from freetb4matlab added');
end;
然后运行即可,运行完在调用savepath命令,就能保存本次执行结果,以后不用再运行install.m文件(可以在install.m文件最后一行添一行savepath)






















 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








