有个表达谱基因数太多了,我用seurat找了500个高可变基因,
![]()
然后,我这个不太会R语言的人,把基因列表输出到文本文件,再用python找这500个基因对应的表达谱。先用的dataframe的切片操作,切出来一直只有492个基因,感觉很奇怪,要不是我检查了一遍,可能就少了这8个基因了。
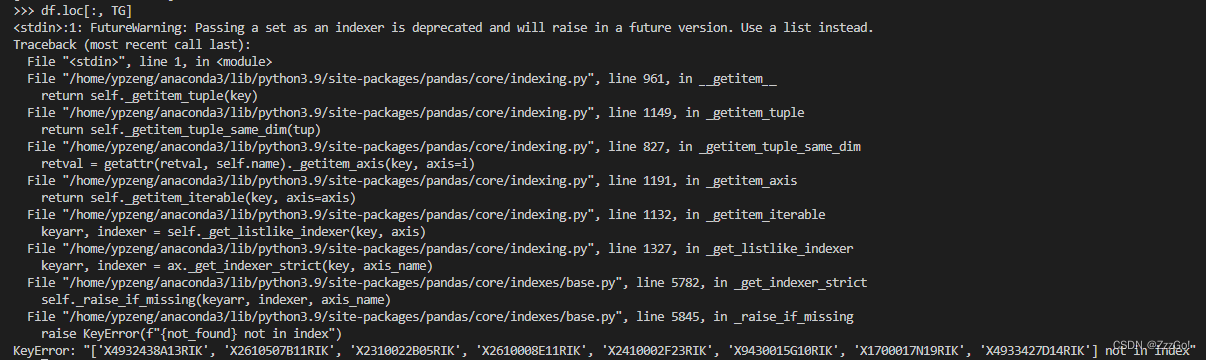
后来调用了loc函数,找到了那8个基因。这几个基因在原来的表达谱里的名字都没有X打头,查了一下才知道是因为R语言read.csv函数读入数据时会在数字开头的列名前自动加上“X”(冤大头就是我)。。。
解决办法:
调用函数时在参数里加check.names=FALSE
data<- read.csv(file, header=TRUE, check.names=FALSE)
ps:如果我是个会R语言数据切片的人,我可能会用 data[top500,] 这样取高可变基因的表达谱,这样切出来的维度是对的。但是我就错失了一次改bug学习的机会,另外,这八个基因就会像消失了一样。。(因为名字跟别的对不上。。。)
![]()
(感慨一下,无知有时候也挺好哈哈)



























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








