写在前面: 之前写过一篇论文笔记,讲的是MoleculeNet,同样的,这篇论文也是一篇起到benchmark作用的论文,但针对的是不同指纹在分子相似性搜索方面的基准和综合。
目前获取分子2D药效团的算法主要可以分为4类:
(1)基于字典的;
(2)基于拓扑或路径的;
(3)圆形指纹;
(4)药效团指纹。
目前一些常用虚拟筛选(VS)的评价指标:
(1)接收机工作特征(ROC)曲线下面积(AUC);(AUC对早期识别不够敏感)
(2)在数据集的给定分数χ处的富集因子(EF);(EF依赖于active与inactive的比例和χ的选择)
(3)robust initial enhancement (RIE);(使用一个连续递减的指数权重作为秩的函数,因此对早期识别很敏感,但同样依赖可调参数,不适合在不同数据集之间进行比较)
(4)玻尔兹曼增强鉴别(BEDROC)。(强迫RIE在0到1之间,避免了对active/inactive的依赖)
以上指标,本文全用。
标准数据库:
(1)MDDR(11个目标取自MDL药物数据报告)商用,不开放。
(2)DUD(the directory of useful decoys)
(3)MUV(the maximum unbiased validation)
(4)CHEMBL
分析讨论:
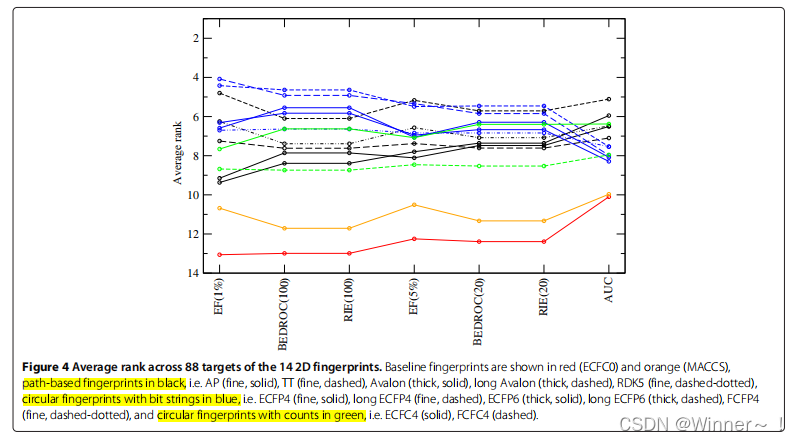
关于指纹,很多研究发现,二维方法通常优于3D方法。以上四种指纹中,圆形指纹的整体性能较好。也有其他研究标明,较长的位字符串表现得更好,但是,请注意,这些出版物都使用了不同的指纹集、不同的参考数据集和不同的评价标准,这使得不可能直接比较它们的结果和结论。
正因为难以比较,所以在这里,通过提供可用于比较的数据集和源代码,来实现真正意义上的可重复性和可比性。于是提供了三个数据集:MUV,DUD和ChEMBL,和14个指纹(ECFP0和MACCS作为baseline指纹)。并且指出,一般来说,给定目标的指纹之间的差异要小于给定指纹的目标之间的差异。在评估跨目标的指纹性能时,记住这一点是很重要的。也就是说,相同target,不同指纹间的差距可能不大,但是相同指纹作用在不同target上,性能表现天差地别。
后面,文章又对比了,不同评价指标之间的相关性和不同指纹之间的相关性。(本人不是太关心,所以略过)
Ranking of fingerprints
在50次VS实验中,每14个指纹的表现进行排序。这些等级可以在重复和目标中进行平均。因为平均排序的标准偏差很大,一个全局的Friedman检验被第一步计算来评价指纹性能的优越性。
scaffold diversity 分析
VS中的结构多样性分析,能够识别结构多样但功能相似的分子,称为“scaffold hopping”,被认为是相似性方法的理想特性。虽然二维指纹是一种简单的相似性方法,但一些已经被发现具有显著的scaffold hopping潜力,但关于scaffold的定义很模糊并且存在很多定义形式,其中,分子框架的Bemis-Murcko scaffold(BMS)被广泛采用。
BMS/actives过低(<50%)或过高(80-100%)都会使scaffold-hopping潜力的评估变得无意义。
scaffold-hopping潜力的评估可以用scaffold EF来测量,其计算方法与EF类似。
从下面两图可以看出,scaffold EF与指纹的一般性能密切相关。这种强相关性,也使得scaffold-hopping潜力的评估是无效的。


模拟虚拟筛选:
(1)Scoring : 计算相似性,.对于每个测试分子,只考虑最高的相似性值。
相似性测量指标:Dice,Tanimoto, Cosine, Russel, Kulczynski, McConnaughey, Manhattan and Rogot-Goldberg.前两个效果差不多。
(2)Validation:加载上一步中的排序列表,并使用不同的评价方法计算每个指纹的性能。目前支持以下评价方法:AUC、RIE、BEDROC和EF。
(3)从每个指纹和评价方法的50个排名列表中,计算出每个目标的平均性能。此外,根据每种方法计算每个指纹在所有数据集上的平均rank。对于每种评价方法,采用全局弗里德曼检验来检测指纹平均rank之间的统计学显著差异。
代码在论文的附件4中

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








