第16章 聚类分析
本章需要的包
library(flexclust)
library(rattle)
library(cluster)
library(NbClust)
library(fMultivar)
library(ggplot2)
本章使用的相关数据集:
flexclust包中的nutrient数据集
rattle包中的wine数据集
data(nutrient,package="flexclust")
data(wine,package="rattle")
16.1 聚类分析的一般步骤
选择合适的变量
缩放数据(最常用的方法是将每个变量标准化为均值为0标准差为1的变量 scale)
寻找异常点(outliers包 mvoutlier包)
计算距离(最常用欧几里得距离,还有曼哈顿距离、兰氏距离、非对称二元距离、最大距离等)
选择聚类算法(层次聚类、划分聚类)
获得一种或多种聚类方法
确定类的数目(NbCluster包中NbCluster()函数可以协助判断)
获得最终的聚类解决方案
结果可视化(层次聚类一般是树状图,划分聚类一般是双变量聚类图)
解读类(1个类的观测值有什么相似之处,不同类的观测值之间有何不同)
验证结果(是否确实给出了一个某种程度上有实际意义的结果)
16.2 计算欧氏距离
d <- dist(nutrient) #返回的是下三角矩阵
as.matrix(d) #将输出格式进行转化 用as.matrix()可使用标准括号符号得到距离
as.matrix(d)[1:4,1:4] #若只想看前四个数据
#观测值之间距离越大 异质性越大
#cluster::daisy() 函数可以获取任意二元 名义 有序 连续属性组合的相异矩阵
#cluster::agnes() 函数提供了层次聚类 cluster::pam()函数提供了围绕中心点的划分方法
16.3 层次聚类分析
通过hclust()函数实现 格式为hclust(d,method=)
d是通过dist()函数产生的距离矩阵
层次聚类方法包括单联动single 全联动complete 平均联动average 质心centroid ward法
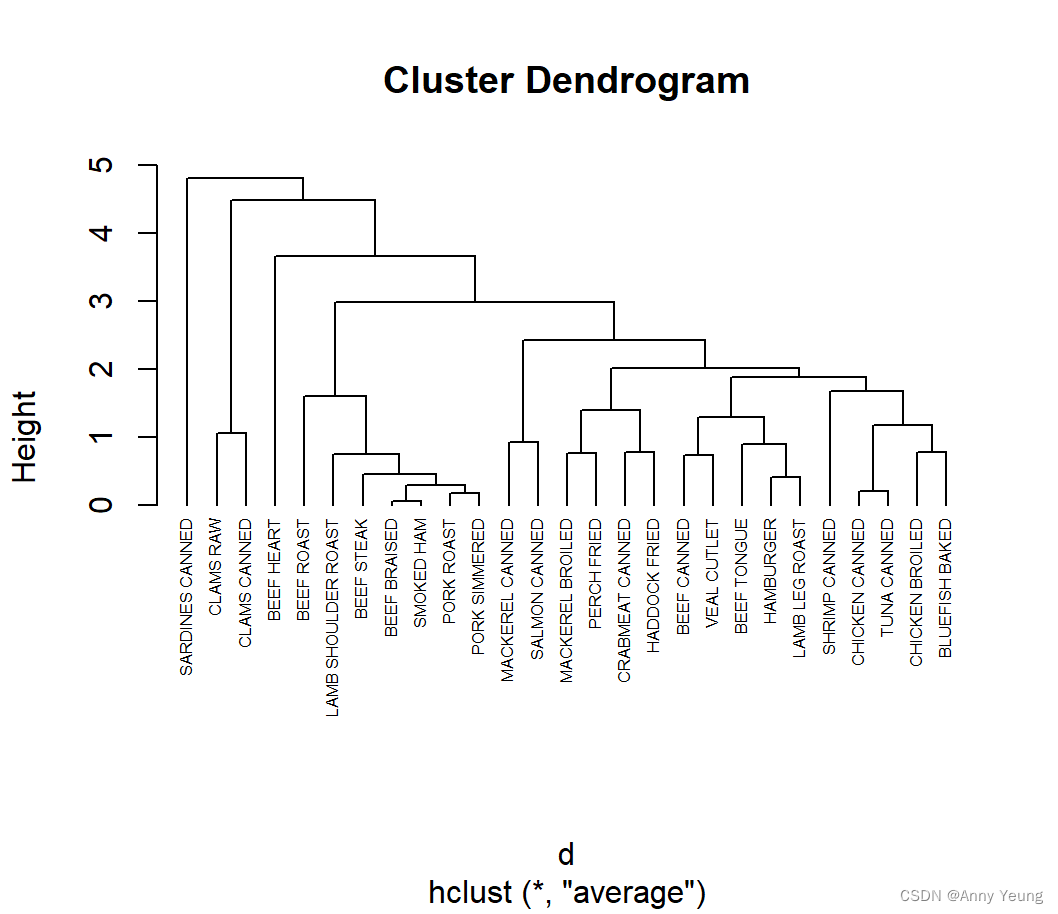
采用平均联动进行层次聚类:以营养数据为例
data("nutrient",package="flexclust")
nutrient.scale <- scale(nutrient) #标准化
d <- dist(nutrient.scale) #算距离矩阵
fit.average <- hclust(d,method="average") #进行层次聚类
plot(fit.average,hang=-1,cex=0.5) #hang的作用是让它们都挂在0下面
#选择聚类个数
library(NbClust)
devAskNewPage(ask = TRUE)
# This function can be used to control (for the current device)
# whether the user is prompted before starting a new page of output.
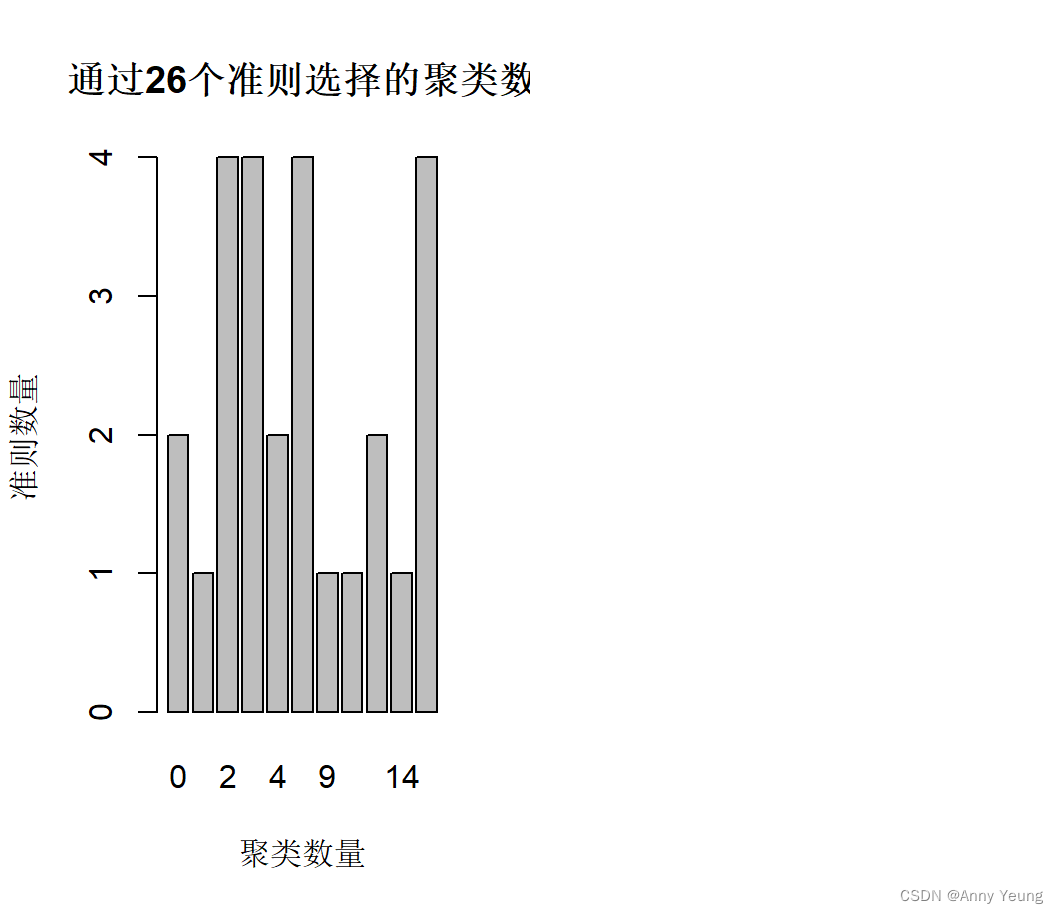
nc <- NbClust(nutrient.scale,distance = "euclidean",
min.nc = 2,max.nc = 15,method = "average")
nc
Return
table(nc$Best.n[1,])
barplot(table(nc$Best.n[1,]),
xlab="聚类数量",ylab="准则数量",
main="通过26个准则选择的聚类数" )
Return
#结果可见 最高4个评判准则赞同聚类个数为2 3 5 15个聚类个数 可以选择其中一个
#使得解释最有意义
#获取最终的聚类方案
clusters <- cutree(fit.average,k=5)
table(clusters) #每个类中的观测值数量
aggregate(nutrient,by=list(cluster=clusters),median) #获取原始度量中位数
aggregate(as.data.frame(nutrient.scale),
by=list(cluster=clusters),median) #获取标准度量中位数
plot(fit.average,hang=-1,cex=0.8,
main="五类解决方案得到营养数据的平均联动聚类")
rect.hclust(fit.average,k=5) #能显示五个分类的具体情况
16.4 划分聚类分析(k均值和基于中心点的划分)
16.4.1 k均值聚类
使用欧几里得距离(能处理比层次聚类更大的数据集 变量必须是联系的 且受异常值影响)
# 构建代码 类中总的平方值对聚类数量的曲线 选择适当的类的数量
wssplot <- function(data,nc=15,seed=1234){ #nc是要考虑的最大聚类个数
wss <- (nrow(data)-1)*sum(apply(data,2,var))
for (i in 2:nc) {
set.seed(seed) #设置中子数 保证结果可复制
wss[i] <- sum(kmeans(data,centers = i)$withiness)}
plot(1:nc,wss,type="b",xlab="聚类数量",
ylab="within groups sum of squares")}
k均值聚类:以葡萄酒数据为例
因为第一个变量是葡萄酒的分类 我们可以用这个检验最后的结果
data(wine,package="rattle")
head(wine)
df <- scale(wine[-1]) #对除类型外的数据进行标准化#决定聚类的个数 方法1
wssplot(df)
Return
#从1类到3类下降的很快 之后下降的很慢 建议选用聚类数为3#决定聚类的个数 方法2
library(NbClust)
set.seed(1234)
devAskNewPage(ask=TRUE)
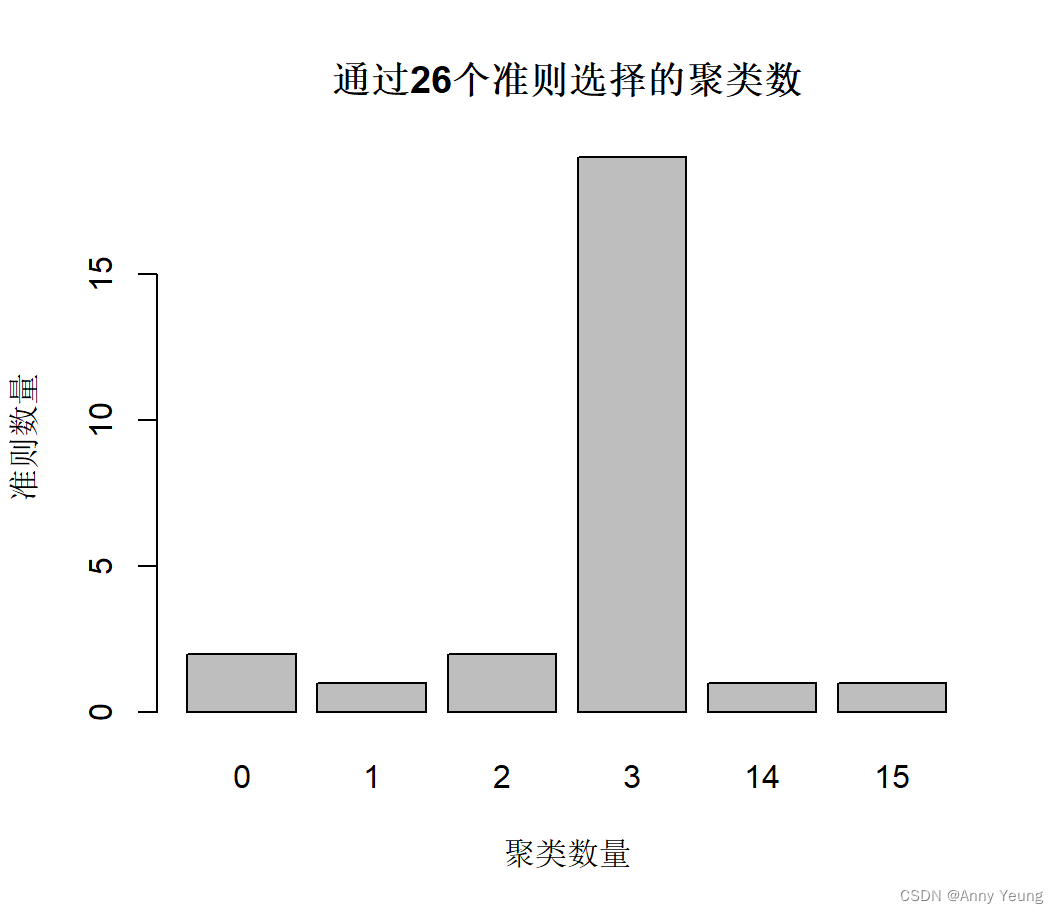
nc <- NbClust(df,min.nc = 2,max.nc = 15,method = "kmean")
table(nc$Best.n[1,])
barplot(table(nc$Best.n[1,]),
xlab="聚类数量",ylab="准则数量",
main="通过26个准则选择的聚类数")
return
#最佳聚类数为3
进行k均值聚类分析
set.seed(123)
fit.km <- kmeans(df,3,nstart = 25)
fit.km$sizefit.km$centers
aggregate(wine[-1],by=list(cluster=fit.km$cluster),mean)用数据集中原有类型检验本次聚类结果
ct.km <- table(wine$Type,fit.km$cluster)
ct.km
library(flexclust)
flexclust::randIndex(ct.km)
#结果范围在[-1,1] -1不同意 1同意
16.4.2 围绕中心点的划分(PAM 使用任意的距离来计算)
基于质心的划分方法:以葡萄酒数据为例
library(cluster)
set.seed(1234)#聚类数据的标准化 stand=TRUE
fit.pam <- cluster::pam(wine[-1],k=3,stand = TRUE)#输出中心点
fit.pam$medoids
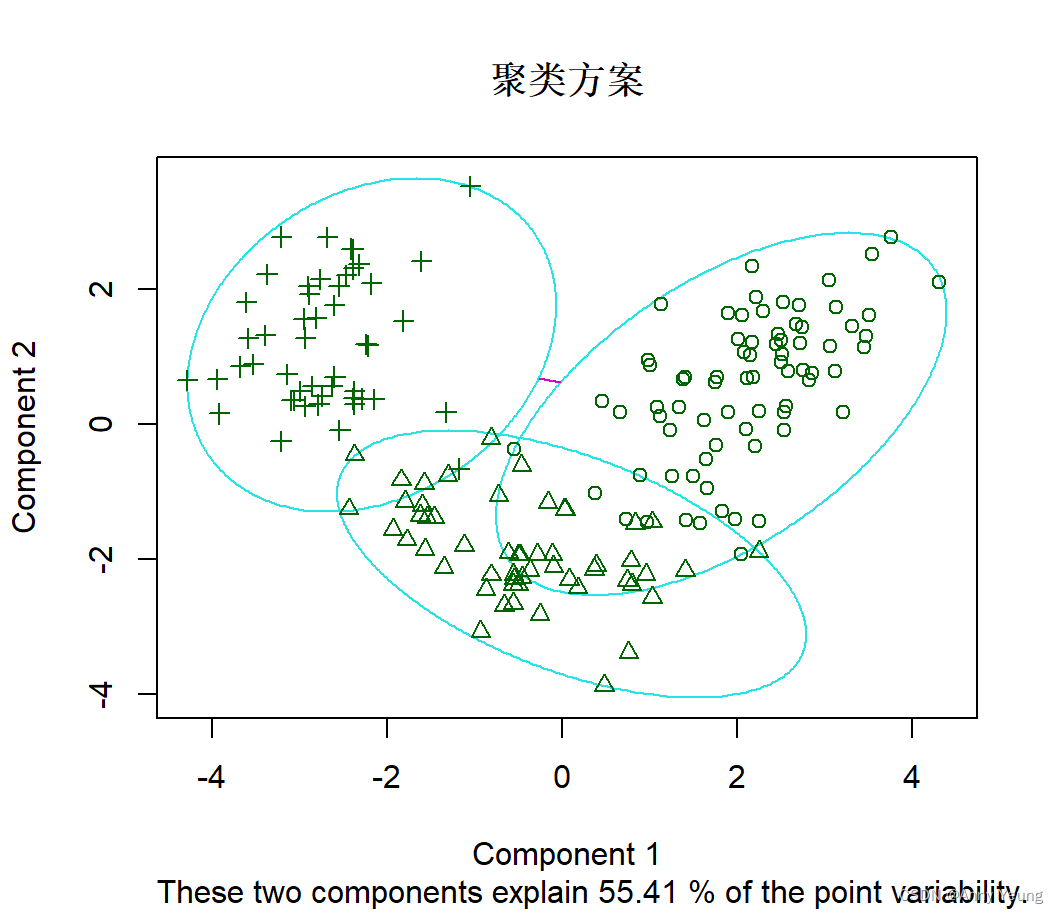
clusplot(fit.pam,
main="聚类方案")
return #从13个测定变量上得到的前两个主成分绘制每个观测的坐标来创建二元图#ps 可以按分类画出散点图
wine$cluster <- factor(fit.km$cluster)#k均值聚类结果赋值给原数据wine后进行绘图
ggplot(data = wine,aes(x=Alcalinity ,y=Magnesium ,color=cluster,shape=cluster))+
geom_point()
return
#用数据集中原有类型检验本次聚类结果
ct.pam <- table(wine$Type,fit.pam$clustering)
ct.pam
library(flexclust)
flexclust::randIndex(ct.pam)
#调整的懒得指数从(k均值的)0.9下降到0.7 说明有关葡萄酒数据PAM的表现不如k均值
16.5 避免不存在的类
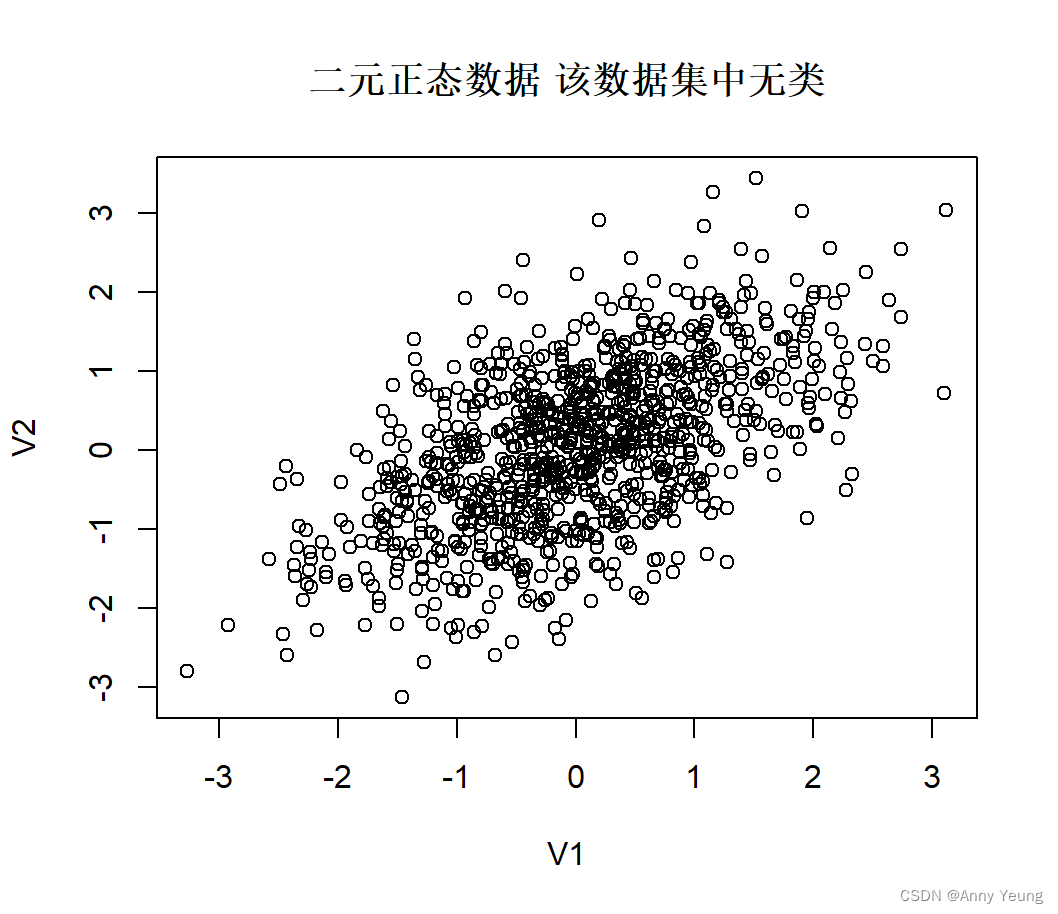
#用fMultivar包中的rnorm2d函数从相关系数为0.5的二元正态分布中抽取1000个观测值 用于本节讨论
library(fMultivar)
set.seed(123)
testdata <- fMultivar::rnorm2d(1000,rho=0.5)
testdata <- as.data.frame(testdata)
plot(testdata,
main="二元正态数据 该数据集中无类")
#使用wssplot和NbClust函数确定当前聚类个数
wssplot(testdata)return
library(NbClust)
nc <- NbClust(testdata,min.nc = 2,max.nc = 15,method = "kmeans")barplot(table(nc$Best.n[1,]),xlab="聚类数量",ylab="准则数量",main="通过26个准则选择的聚类数")
#wssplot建议个数是3个 NbClust多数支持2 3类
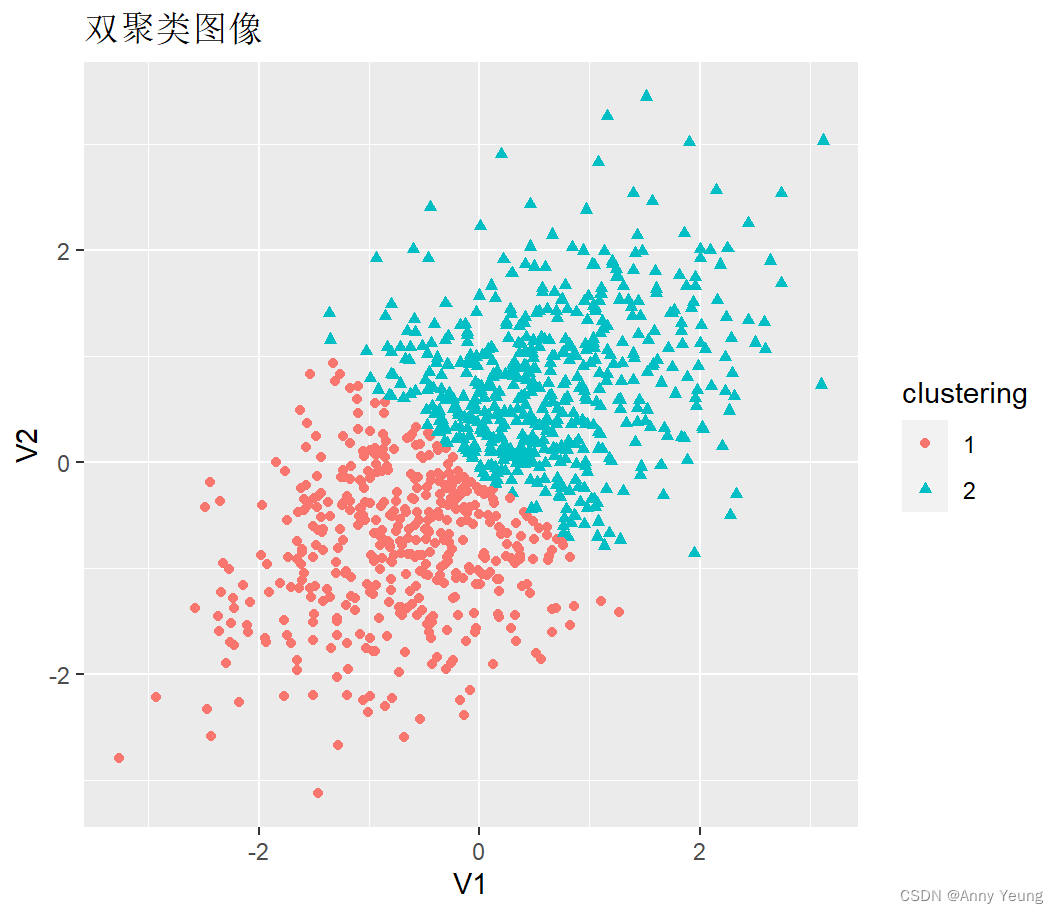
对数据进行聚类分析 利用PAM法进行双聚类分析(构建的数据实际上不存在类 这里只是为引出 避免不存在的类 这一问题)
library(ggplot2)
fit <- cluster::pam(testdata,k=2)
testdata$clustering <- factor(fit$clustering)
ggplot(data = testdata,aes(x=V1,y=V2,color=clustering,shape=clustering))+
geom_point()+ggtitle("双聚类图像")
#很明显这是人为划分的 实际上并不存在这样的真实分类
#利用NbClust包中的立方聚类规则 可以帮我们揭示不存在的结构
plot(nc$All.index[,4],type="o",ylab="CCC",xlab="聚类数量",col="blue")
#当CCC的值为负并且对于两类或是更多的类递减时 就是典型的单峰分布























 13万+
13万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








