hello,大家好,今天给大家分享一个新的内容,基因调控网络分析,文章在Modeling gene regulatory networks using neural network architectures,文章中提出的DeepSEM,可以针对单细胞转录组数据,进行基因调控网络推断,数据可视化和聚类,以及模拟数据生成,非常好的方法,值得大家借鉴和学习。
不同细胞类型中,基因调控网络存在差异,通过单细胞测序,可以找出这些差异。但由于单细胞数据中,不同细胞类型间的噪声大,传统的方法需要复杂校正,而基于深度学习的方法,往往模型是一个黑箱,对其内部运行的细节知之甚少。而DeepSEM,能够只基于单细胞转录组数据,不使用转录因子或单细胞 ATAC数据,就能完成基因调控网络的推断。

DeepSEM的算法架构是一个beta变分自编码器,其中编码器在经过多层感知机降维之后,通过优化编码器的模拟数据和真实数据的误差,训练模型中的参数,经由编码器,可得出基因调控网络,经由对神经网络权重的UMAP。可得出单细胞数据的聚类及可视化;而通过编码器,可生产类似给定训练数据的模拟数据集,生成的模拟数据,可在数据集有限的情况下,通过结合模拟数据,提升细胞类型分类模型的准确性,增强差异基因识别的准确性。
在来自人类和小鼠的五个细胞系中,选取不同大小的基因调控网络中,在已知调控网络时,使用不同的标准集,DeepSEM的表现,都优于其它方法,而当真集变为细胞特异的chip-seq数据时,其相比非特异性chip-seq数据为真集时,效果更好
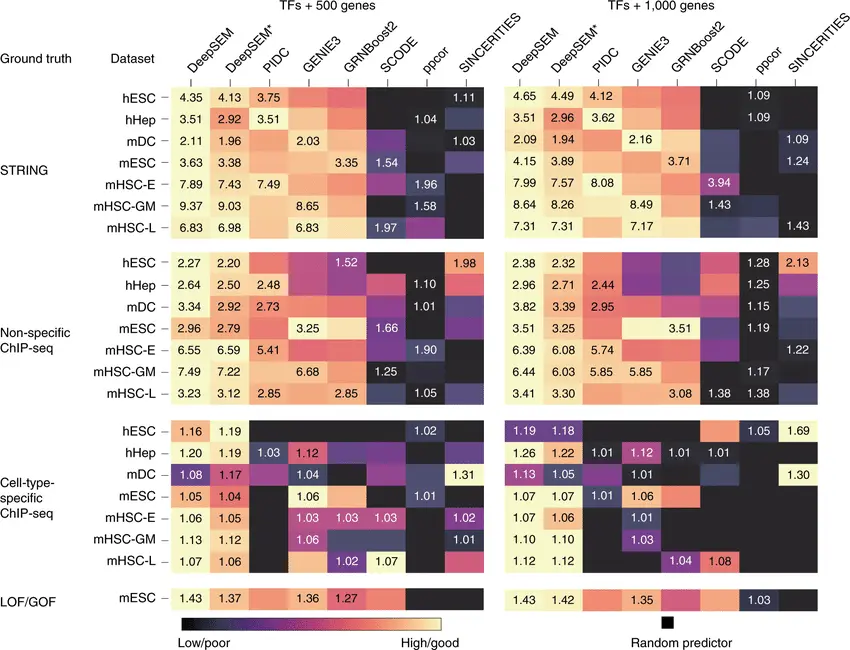
- 注:使用不同数据,将随机预测得出的情况作为最差预测,通过颜色判断推断出的转录网络好坏,图中的数字越小,模型预测的调控网络的边和实际情况越接近。
使用表观数据来验证,发现6个细胞的聚类分开,且不同细胞中,基因Rotb和Syt6预测的调控基因,其表达量都相对较高,这进一步说明了该方法预测的基因调控网络的准确性。

之后,该文论证了使用DeepSEM得出的低维度表征的有效性,相比其它方法得出的低维度表征,在由小鼠大脑,胚胎及外周血组成的数据集中,使用DeepDEM得出的表征,在可视化后细胞间分的更开,且ARI和MNI两个量化指标得分也更高。

由于DeepSEM的运行时间,会随着转录组中基因数目的变化而增大,因此作者推荐输入数据应先筛选出高可变基因,而不是以全部转录组作为输入。考虑到基因调控网络在各类细胞中是相同的,该方法构建的调控网络,可以作为隐空间,用于单细胞数据的批次校正。同时,类似的方法,未来还可能应用于更多类型的数据,例如转录组或染色质开放性的测序数据上。
示例代码链接
数据聚类
网络调控推断
数据模拟
生活很好,有你更好







 本文介绍了一种名为DeepSEM的深度学习方法,它能有效处理单细胞转录组数据,推断基因调控网络并进行数据可视化和聚类。实验结果显示,DeepSEM在多个细胞系和数据集上的表现优于传统方法。此外,文章还探讨了其在细胞批次校正和潜在空间应用的可能性。
本文介绍了一种名为DeepSEM的深度学习方法,它能有效处理单细胞转录组数据,推断基因调控网络并进行数据可视化和聚类。实验结果显示,DeepSEM在多个细胞系和数据集上的表现优于传统方法。此外,文章还探讨了其在细胞批次校正和潜在空间应用的可能性。

















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








