作者,Evil Genius
今天早上研究生一个宿舍的同学发来消息,自己发一篇nature正刊,这下子以后的路会很顺畅了,但是像我这样工作的人,发文章的机会应该很渺茫了。
今天我们又要完成一项工程,昨天有位粉丝问我,想多样本分析空间niche,自己拿到的是Xenium的样本,我们今天就把各种精度的niche分析全部总结一样,包括多样本的比较niche分析,niche分析在很多文章里也描述成了细胞的community。
首先我们必须要承认,10X可以说是生物科研方向一个“伟大”公司,单细胞不必说,10X将单细胞通量极大提升并且带到了全世界,这让我想起了当初英国的瓦特改良了蒸汽机,从而引发了第一次工业革命,由于10X利润很高,所以单细胞平台也在不断更新,最新已经到了GEM-X,我写过一篇介绍,文章在The neXt generation of single cell RNA-seq(GEM-X)。文章有个比喻很有意思,把单细胞技术比喻成了哈勃望远镜,对一个被低估的宇宙有了前所未有的认识。
空间角度来看,10X也几乎整合所有精度、各种技术的平台技术。而Xenium是2023年下半年推出的,这里也要说一下兰卫医学也是一家实力强劲的上市公司,引进了10X所有的平台技术,Xenium一推出就引进,同时配备了C9的博士们下场做一线分析,我也参与了其中,这配置,男人看了会沉默,女人看了会流泪!
说到这里,我们就需要来总结一下空间的技术分类,分类大约分为两类,大家应该都明白,一类是NGS-based空间技术,一类是image-based 空间技术。
我们首先来说NGS-based 空间技术,这一类技术典型的就是Visium,HD,华大的Stereo-seq、以及像寻因、百迈客的空间平台等。
这一类平台典型就是通量高,几乎是测全转录组,信息充足;缺点就是精度低,无论是Visium、HD,还是华大平台等其他,均无法真正实现单细胞级别的空间精度,其中寻因采用了空间核测序的策略实现了空间的单细胞精度,但是嫁接的10X单细胞核技术,细胞丢失率在20%以上。

我们再来看image-based 空间技术,这一类技术典型的就是Xenium,对标的技术还有CODEX等技术,如下图

image-based 空间技术优点就是精度高,经过详细的细胞边界划分(细胞分割)达到了真正的单细胞级别,但是缺点是通量低,测到的基因数目前还停留在几百,10X据说要达到5000.
同时我们也要明白,NGS-based空间技术和image-based 空间技术并不是相互独立的,有时也会融合在一起,比如我之前分享的文章空间单细胞图谱揭示脊椎动物咽部器官的演化起源就结合了这两种技术。

现在兰卫医学科研团队的努力下,分析都已经很成熟了。
10X Xenium的平台我相信接触的人不多,一个样本要9万,现在估计便宜一点了。



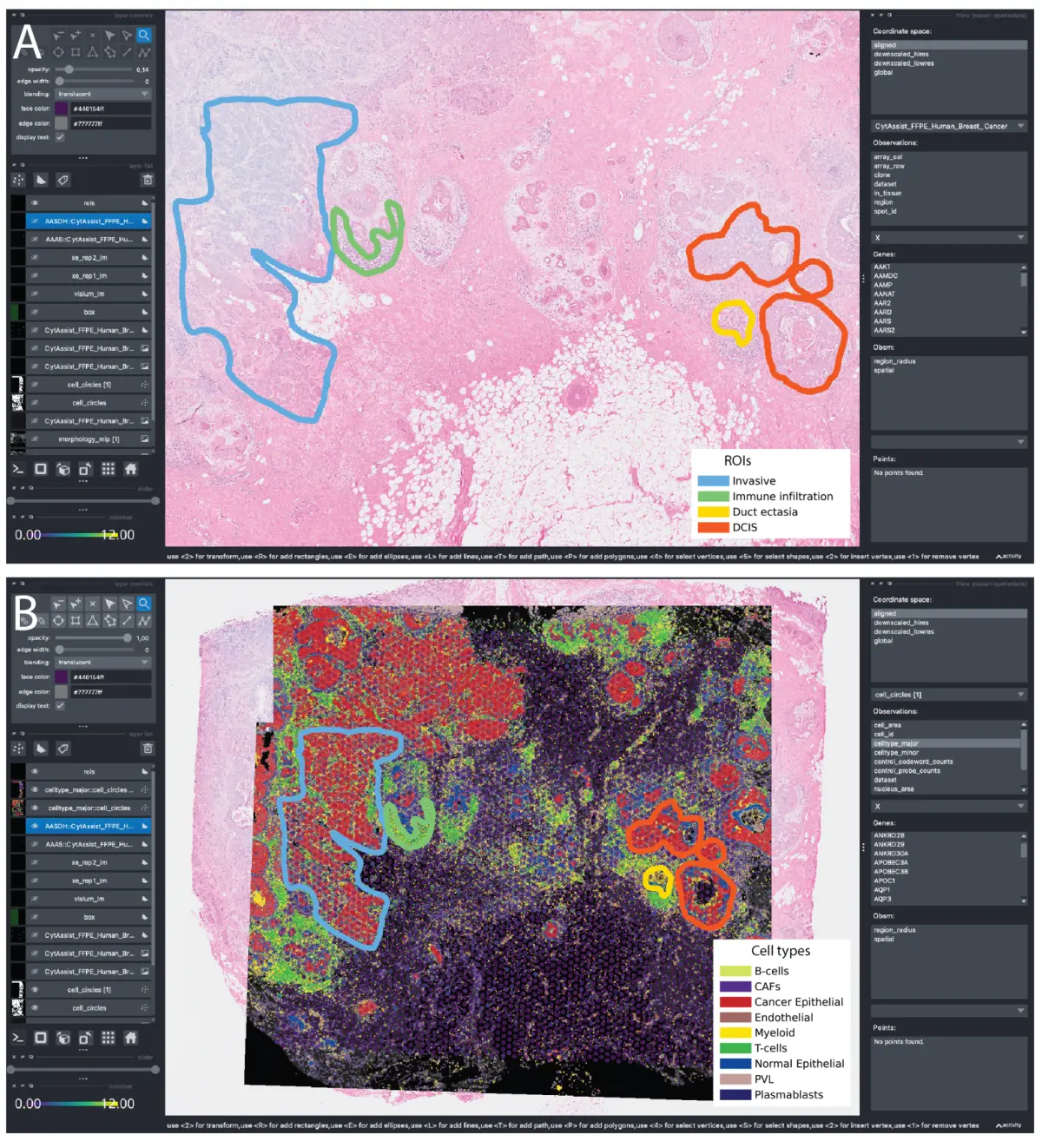

好了,讲了课这么多,我们要进行我们今天的主题工程了。首先我们来分析非单细胞精度的niche(Visium为主,华大、HD也包括),其实这个分析早就分享过了,参考的文章在Spatial multi-omic map of human myocardial infarction(nature)。
首先我们要知道niche的定义,niche有两种,在非单细胞精度的空间中,第一种将细胞组成相似的spot定义为niche,以表征具体的微环境,这里不以某种细胞类型为核心;另外一种niche的定义是以某种细胞类型为核心,比如肿瘤细胞,分析其周围的细胞类型的组成,从而进一步分析微环境,这种niche的定义通常需要单细胞级别的平台,文章大多数的非单细胞级别的空间平台niche的分析角度是前者。
我们先来看第一种niche的分析,这种分析首先我们需要做空间聚类,当然空间聚类也很复杂,专门有一篇文章介绍过,在空间转录组数据分析之CNV burden和CNV聚类(python版本),主流还是分子聚类和细胞聚类,如文章所示。



这种情况下表达相似或者细胞组成相似的spot聚成了一类,每一个cluster定义为一个niche,然后就可以分析niche的细胞组成等内容,进一步研究微环境的改变。

这些方法全部都分享过,空间系列课程上全部讲过,大家拿到资料要好好学习啊。
第二种niche,以某种细胞类型为核心,参考文章在Identification of HSC/MPP expansion units in fetal liver by single-cell spatiotemporal transcriptomics,如下图:

这种核心细胞需要人为指定,就是感兴趣的细胞类型

指定细胞类型后,研究其周围细胞类型的变化,这个分析难度也不低,我之前也分享过方法。
接下来看image-based的niche分析。这种方法采用细胞分割技术,和HE图片的契合度相当高了,下面是团队的一些结果,C9博士的水平果然是不一样的。





由于image-based的通量低,所以一般不能套用NGS的那套方法直接降维聚类等等,好在其精度很高,所以直接就是单细胞级别的空转,所以分析起来相对好一点。


常规的分析方法总结如下

这里大家就可以看出来,单细胞级别的平台主要采用的是核心细胞类型的niche分析方法



这时候niche分析首推squidpy,文章在Squidpy: a scalable framework for spatial omics analysis(nature methods),方法分享过,在文章空间细胞共定位与空间邻域通讯分数脚本更新(python版本)
其次是BANKSY,文章在空间组学邻域分析方法更新之BANKSY,空间转录组数据分析之生态位聚类(Banksy)这个方法也是分析邻域的,也就是niche的。


还有就是hoodscanR,文章在高精度空间平台
更多的还可以借鉴一下CODEX平台的分析方法,我自己稍作改动的方法,文章在空转数据分析之细胞“社区”。

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








