今天准备弄双高斯拟合,看到一个信号峰拟合的MATLAB版本的程序,大体看了一下,很不错,先MARK一下,以后再详细研究。
http://terpconnect.umd.edu/~toh/spectrum/CurveFittingC.html
http://terpconnect.umd.edu/~toh/spectrum/InteractivePeakFitter.htm
下面是其CODE。感叹别人的代码写的那个结构可扩展性,牛人啊!先来看他的一个双高斯拟合示例:
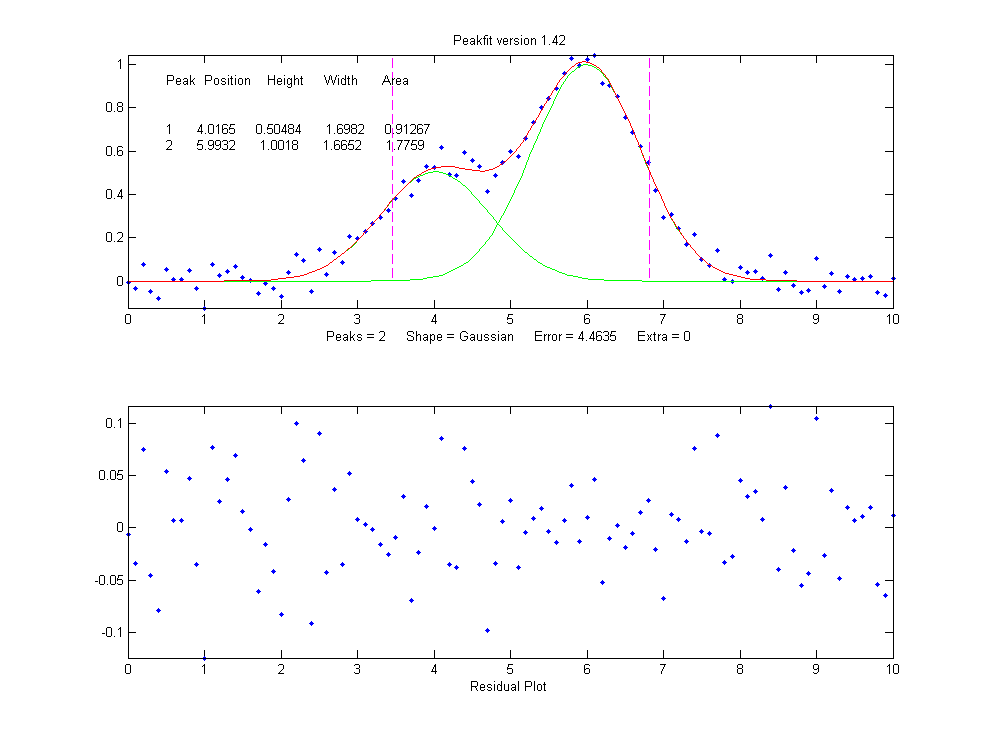
下面是MATLAB峰拟合函数:
- function [FitResults,LowestError,BestStart,xi,yi]=peakfit(signal,center,window,NumPeaks,peakshape,extra,NumTrials,start,AUTOZERO)
- % Version 2.2: October, 2011. Adds exponential pulse and sigmoid models
- % A command-line peak fitting program for time-series signals,
- % written as a self-contained Matlab function in a single m-file.
- % Uses an non-linear optimization algorithm to decompose a complex,
- % overlapping-peak signal into its component parts. The objective
- % is to determine whether your signal can be represented as the sum of
- % fundamental underlying peaks shapes. Accepts signals of any length,
- % including those with non-integer and non-uniform x-values. Fits
- % Gaussian, equal-width Gaussians, exponentially-broadened Gaussian,
- % Lorentzian, equal-width Lorentzians, Pearson, Logistic, exponential
- % pulse, abd sigmoid shapes (expandable to other shapes). This is a command
- % line version, usable from a remote terminal. It is capable of making
- % multiple trial fits with sightly different starting values and taking
- % the one with the lowest mean fit error. Version 2.2: Sept, 2011.
- %
- % PEAKFIT(signal);
- % Performs an iterative least-squares fit of a single Gaussian
- % peak to the data matrix "signal", which has x values
- % in column 1 and Y values in column 2 (e.g. [x y])
- %
- % PEAKFIT(signal,center,window);
- % Fits a single Gaussian peak to a portion of the
- % matrix "signal". The portion is centered on the
- % x-value "center" and has width "window" (in x units).
- %
- % PEAKFIT(signal,center,window,NumPeaks);
- % "NumPeaks" = number of peaks in the model (default is 1 if not
- % specified).
- %
- % PEAKFIT(signal,center,window,NumPeaks,peakshape);
- % Specifies the peak shape of the model: "peakshape" = 1-5.
- % (1=Gaussian (default), 2=Lorentzian, 3=logistic, 4=Pearson,
- % 5=exponentionally broadened Gaussian; 6=equal-width Gaussians;
- % 7=Equal-width Lorentzians; 8=exponentionally broadened equal-width
- % Gaussian, 9=exponential pulse, 10=sigmoid).
- %
- % PEAKFIT(signal,center,window,NumPeaks,peakshape,extra)
- % Specifies the value of 'extra', used in the Pearson and the
- % exponentionally broadened Gaussian shapes to fine-tune the peak shape.
- %
- % PEAKFIT(signal,center,window,NumPeaks,peakshape,extra,NumTrials);
- % Performs "NumTrials" trial fits and selects the best one (with lowest
- % fitting error). NumTrials can be any positive integer (default is 1).
- %
- % PEAKFIT(signal,center,window,NumPeaks,peakshape,extra,NumTrials,start)
- % Specifies the first guesses vector "firstguess" for the peak positions
- % and widths, e.g. start=[position1 width1 position2 width2 ...]
- %
- % [FitResults,MeanFitError]=PEAKFIT(signal,center,window...)
- % Returns the FitResults vector in the order peak number, peak
- % position, peak height, peak width, and peak area), and the MeanFitError
- % (the percent RMS difference between the data and the model in the
- % selected segment of that data) of the best fit.
- %
- % Optional output parameters
- % 1. FitResults: a table of model peak parameters, one row for each peak,
- % listing Peak number, Peak position, Height, Width, and Peak area.
- % 2. LowestError: The rms fitting error of the best trial fit.
- % 3. BestStart: the starting guesses that gave the best fit.
- % 4. xi: vector containing 100 interploated x-values for the model peaks.
- % 5. yi: matrix containing the y values of each model peak at each xi.
- % Type plot(xi,yi(1,:)) to plot peak 1 or plot(xi,yi) to plot all peaks
- %
- % T. C. O'Haver (toh@umd.edu). Version 2.2
- %
- % Example 1:
- % >> x=[0:.1:10]';y=exp(-(x-5).^2);peakfit([x y])
- % Fits exp(-x)^2 with a single Gaussian peak model.
- %
- % Peak number Peak position Height Width Peak area
- % 1 5 1 1.665 1.7725
- %
- % Example 2:
- % x=[0:.1:10]';y=exp(-(x-5).^2)+.1*randn(size(x));peakfit([x y])
- % Like Example 1, except that random noise is added to the y data.
- % ans =
- % 1 5.0279 0.9272 1.7948 1.7716
- %
- % Example 3:
- % x=[0:.1:10];y=exp(-(x-5).^2)+.5*exp(-(x-3).^2)+.1*randn(size(x));
- % peakfit([x' y'],5,19,2,1,0,1)
- % Fits a noisy two-peak signal with a double Gaussian model (NumPeaks=2).
- % ans =
- % 1 3.0001 0.49489 1.642 0.86504
- % 2 4.9927 1.0016 1.6597 1.7696
- %
- % Example 4:
- % >> y=lorentzian(1:100,50,2);peakfit(y,50,100,1,2)
- % Create and fit Lorentzian located at x=50, height=1, width=2.
- % ans =
- % 1 50 0.99974 1.9971 3.1079
- % Example 5:
- % >> x=[0:.005:1];y=humps(x);peakfit([x' y'],.3,.7,1,4,3);
- % Fits a portion of the humps function, 0.7 units wide and centered on
- % x=0.3, with a single (NumPeaks=1) Pearson function (peakshape=4)
- % with extra=3 (controls shape of Pearson function).
- %
- % Example 6:
- % >> x=[0:.005:1];y=(humps(x)+humps(x-.13)).^3;smatrix=[x' y'];
- % >> [FitResults,MeanFitError]=peakfit(smatrix,.4,.7,2,1,0,10)
- % Creates a data matrix 'smatrix', fits a portion to a two-peak Gaussian
- % model, takes the best of 10 trials. Returns FitResults and MeanFitError.
- % FitResults =
- % 1 0.31056 2.0125e+006 0.11057 2.3689e+005
- % 2 0.41529 2.2403e+006 0.12033 2.8696e+005
- % MeanFitError =
- % 1.1899
- %
- % Example 7:
- % >> peakfit([x' y'],.4,.7,2,1,0,10,[.3 .1 .5 .1]);
- % As above, but specifies the first-guess position and width of the two
- % peaks, in the order [position1 width1 position2 width2]
- %
- % Example 8:
- % >> peakfit([x' y'],.4,.7,2,1,0,10,[.3 .1 .5 .1],0);
- % As above, but sets AUTOZERO mode in the last argument.
- % AUROZERO=0 does not subtract baseline from data segment.
- % AUROZERO=1 (default) subtracts linear baseline from data segment.
- % AUROZERO=2, subtracts quadratic baseline from data segment.
- %
- % For more details, see
- % http://terpconnect.umd.edu/~toh/spectrum/CurveFittingC.html and
- % http://terpconnect.umd.edu/~toh/spectrum/InteractivePeakFitter.htm
- %
- global AA xxx PEAKHEIGHTS
- format short g
- format compact
- warning off all
- datasize=size(signal);
- if datasize(1)<datasize(2),signal=signal';end
- datasize=size(signal);
- if datasize(2)==1, % Must be isignal(Y-vector)
- X=1:length(signal); % Create an independent variable vector
- Y=signal;
- else
- % Must be isignal(DataMatrix)
- X=signal(:,1); % Split matrix argument
- Y=signal(:,2);
- end
- X=reshape(X,1,length(X)); % Adjust X and Y vector shape to 1 x n (rather than n x 1)
- Y=reshape(Y,1,length(Y));
- % If necessary, flip the data vectors so that X increases
- if X(1)>X(length(X)),
- disp('X-axis flipped.')
- X=fliplr(X);
- Y=fliplr(Y);
- end
- % Y=Y-min(Y); % Remove excess offset from data
- % Isolate desired segment from data set for curve fitting
- if nargin==1 || nargin==2,center=(max(X)-min(X))/2;window=max(X)-min(X);end
- xoffset=center-window/2;
- n1=val2ind(X,center-window/2);
- n2=val2ind(X,center+window/2);
- if window==0,n1=1;n2=length(X);end
- xx=X(n1:n2)-xoffset;
- yy=Y(n1:n2);
- ShapeString='Gaussian';
- % Define values of any missing arguments
- switch nargin
- case 1
- NumPeaks=1;
- peakshape=1;
- extra=0;
- NumTrials=1;
- xx=X;yy=Y;
- start=calcstart(xx,NumPeaks,xoffset);
- AUTOZERO=1;
- case 2
- NumPeaks=1;
- peakshape=1;
- extra=0;
- NumTrials=1;
- xx=signal;yy=center;
- start=calcstart(xx,NumPeaks,xoffset);
- AUTOZERO=1;
- case 3
- NumPeaks=1;
- peakshape=1;
- extra=0;
- NumTrials=1;
- start=calcstart(xx,NumPeaks,xoffset);
- AUTOZERO=1;
- case 4
- peakshape=1;
- extra=0;
- NumTrials=1;
- start=calcstart(xx,NumPeaks,xoffset);
- AUTOZERO=1;
- case 5
- extra=0;
- NumTrials=1;
- start=calcstart(xx,NumPeaks,xoffset);
- AUTOZERO=1;
- case 6
- NumTrials=1;
- start=calcstart(xx,NumPeaks,xoffset);
- AUTOZERO=1;
- case 7
- start=calcstart(xx,NumPeaks,xoffset);
- AUTOZERO=1;
- case 8
- AUTOZERO=1;
- otherwise
- end % switch nargin
- % Remove baseline from data segment (alternative code)
- % lxx=length(xx);
- % bkgsize=10;
- % if AUTOZERO==1, % linear autozero operation
- % XX1=xx(1:round(lxx/bkgsize));
- % XX2=xx((lxx-round(lxx/bkgsize)):lxx);
- % Y1=yy(1:round(length(xx)/bkgsize));
- % Y2=yy((lxx-round(lxx/bkgsize)):lxx);
- % bkgcoef=polyfit([XX1,XX2],[Y1,Y2],1); % Fit straight line to sub-group of points
- % bkg=polyval(bkgcoef,xx);
- % yy=yy-bkg;
- % end % if
- % Remove baseline from data segment
- X1=min(xx);X2=max(xx);Y1=min(Y);Y2=max(Y);
- if AUTOZERO==1, % linear autozero operation
- Y1=mean(yy(1:length(xx)/20));
- Y2=mean(yy((length(xx)-length(xx)/20):length(xx)));
- yy=yy-((Y2-Y1)/(X2-X1)*(xx-X1)+Y1);
- end % if
- if AUTOZERO==2, % Quadratic autozero operation
- XX1=xx(1:round(lxx/bkgsize));
- XX2=xx((lxx-round(lxx/bkgsize)):lxx);
- Y1=yy(1:round(length(xx)/bkgsize));
- Y2=yy((lxx-round(lxx/bkgsize)):lxx);
- bkgcoef=polyfit([XX1,XX2],[Y1,Y2],2); % Fit parabola to sub-group of points
- bkg=polyval(bkgcoef,xx);
- yy=yy-bkg;
- end % if autozero
- PEAKHEIGHTS=zeros(1,NumPeaks);
- n=length(xx);
- newstart=start;
- switch NumPeaks
- case 1
- newstart(1)=start(1)-xoffset;
- case 2
- newstart(1)=start(1)-xoffset;
- newstart(3)=start(3)-xoffset;
- case 3
- newstart(1)=start(1)-xoffset;
- newstart(3)=start(3)-xoffset;
- newstart(5)=start(5)-xoffset;
- case 4
- newstart(1)=start(1)-xoffset;
- newstart(3)=start(3)-xoffset;
- newstart(5)=start(5)-xoffset;
- newstart(7)=start(7)-xoffset;
- case 5
- newstart(1)=start(1)-xoffset;
- newstart(3)=start(3)-xoffset;
- newstart(5)=start(5)-xoffset;
- newstart(7)=start(7)-xoffset;
- newstart(9)=start(9)-xoffset;
- case 6
- newstart(1)=start(1)-xoffset;
- newstart(3)=start(3)-xoffset;
- newstart(5)=start(5)-xoffset;
- newstart(7)=start(7)-xoffset;
- newstart(9)=start(9)-xoffset;
- newstart(11)=start(11)-xoffset;
- otherwise
- end % switch NumPeaks
- % Perform peak fitting for selected peak shape using fminsearch function
- options = optimset('TolX',.00001,'Display','off' );
- LowestError=1000; % or any big number greater than largest error expected
- FitParameters=zeros(1,NumPeaks.*2);
- BestStart=zeros(1,NumPeaks.*2);
- height=zeros(1,NumPeaks);
- bestmodel=zeros(size(yy));
- for k=1:NumTrials,
- % disp(['Trial number ' num2str(k) ] ) % optionally prints the current trial number as progress indicator
- switch peakshape
- case 1
- TrialParameters=fminsearch(@fitgaussian,newstart,options,xx,yy);
- ShapeString='Gaussian';
- case 2
- TrialParameters=fminsearch(@fitlorentzian,newstart,options,xx,yy);
- ShapeString='Lorentzian';
- case 3
- TrialParameters=fminsearch(@fitlogistic,newstart,options,xx,yy);
- ShapeString='Logistic';
- case 4
- TrialParameters=fminsearch(@fitpearson,newstart,options,xx,yy,extra);
- ShapeString='Pearson';
- case 5
- TrialParameters=fminsearch(@fitexpgaussian,newstart,options,xx,yy,-extra);
- ShapeString='ExpGaussian';
- case 6
- cwnewstart(1)=newstart(1);
- for pc=2:NumPeaks,
- cwnewstart(pc)=newstart(2.*pc-1);
- end
- cwnewstart(NumPeaks+1)=(max(xx)-min(xx))/5;
- TrialParameters=fminsearch(@fitewgaussian,cwnewstart,options,xx,yy);
- ShapeString='Equal width Gaussians';
- case 7
- cwnewstart(1)=newstart(1);
- for pc=2:NumPeaks,
- cwnewstart(pc)=newstart(2.*pc-1);
- end
- cwnewstart(NumPeaks+1)=(max(xx)-min(xx))/5;
- TrialParameters=fminsearch(@fitlorentziancw,cwnewstart,options,xx,yy);
- ShapeString='Equal width Lorentzians';
- case 8
- cwnewstart(1)=newstart(1);
- for pc=2:NumPeaks,
- cwnewstart(pc)=newstart(2.*pc-1);
- end
- cwnewstart(NumPeaks+1)=(max(xx)-min(xx))/5;
- TrialParameters=fminsearch(@fitexpewgaussian,cwnewstart,options,xx,yy,-extra);
- ShapeString='Exp. equal width Gaussians';
- case 9
- TrialParameters=fminsearch(@fitexppulse,newstart,options,xx,yy);
- ShapeString='Exponential Pulse';
- case 10
- TrialParameters=fminsearch(@fitsigmoid,newstart,options,xx,yy);
- ShapeString='Sigmoid';
- otherwise
- end % switch peakshape
- % Construct model from Trial parameters
- A=zeros(NumPeaks,n);
- for m=1:NumPeaks,
- switch peakshape
- case 1
- A(m,:)=gaussian(xx,TrialParameters(2*m-1),TrialParameters(2*m));
- case 2
- A(m,:)=lorentzian(xx,TrialParameters(2*m-1),TrialParameters(2*m));
- case 3
- A(m,:)=logistic(xx,TrialParameters(2*m-1),TrialParameters(2*m));
- case 4
- A(m,:)=pearson(xx,TrialParameters(2*m-1),TrialParameters(2*m),extra);
- case 5
- A(m,:)=expgaussian(xx,TrialParameters(2*m-1),TrialParameters(2*m),-extra)';
- case 6
- A(m,:)=gaussian(xx,TrialParameters(m),TrialParameters(NumPeaks+1));
- case 7
- A(m,:)=lorentzian(xx,TrialParameters(m),TrialParameters(NumPeaks+1));
- case 8
- A(m,:)=expgaussian(xx,TrialParameters(m),TrialParameters(NumPeaks+1),-extra)';
- case 9
- A(m,:)=exppulse(xx,TrialParameters(2*m-1),TrialParameters(2*m));
- case 10
- A(m,:)=sigmoid(xx,TrialParameters(2*m-1),TrialParameters(2*m));
- otherwise
- end % switch
- switch NumPeaks % adds random variation to non-linear parameters
- case 1
- newstart=[newstart(1)*(1+randn/50) newstart(2)*(1+randn/10)];
- case 2
- newstart=[newstart(1)*(1+randn/50) newstart(2)*(1+randn/10) newstart(3)*(1+randn/50) newstart(4)*(1+randn/10)];
- case 3
- newstart=[newstart(1)*(1+randn/50) newstart(2)*(1+randn/10) newstart(3)*(1+randn/50) newstart(4)*(1+randn/10) newstart(5)*(1+randn/50) newstart(6)*(1+randn/10)];
- case 4
- newstart=[newstart(1)*(1+randn/50) newstart(2)*(1+randn/10) newstart(3)*(1+randn/50) newstart(4)*(1+randn/10) newstart(5)*(1+randn/50) newstart(6)*(1+randn/10) newstart(7)*(1+randn/50) newstart(8)*(1+randn/10)];
- case 5
- newstart=[newstart(1)*(1+randn/50) newstart(2)*(1+randn/10) newstart(3)*(1+randn/50) newstart(4)*(1+randn/10) newstart(5)*(1+randn/50) newstart(6)*(1+randn/10) newstart(7)*(1+randn/50) newstart(8)*(1+randn/10) newstart(9)*(1+randn/50) newstart(10)*(1+randn/10)];
- otherwise
- end % switch NumPeaks
- end % for
- % Multiplies each row by the corresponding amplitude and adds them up
- model=PEAKHEIGHTS'*A;
- % Compare trial model to data segment and compute the fit error
- MeanFitError=100*norm(yy-model)./(sqrt(n)*max(yy));
- % Take only the single fit that has the lowest MeanFitError
- if MeanFitError<LowestError,
- if min(PEAKHEIGHTS)>0, % Consider only fits with positive peak heights
- LowestError=MeanFitError; % Assign LowestError to the lowest MeanFitError
- FitParameters=TrialParameters; % Assign FitParameters to the fit with the lowest MeanFitError
- BestStart=newstart; % Assign BestStart to the start with the lowest MeanFitError
- height=PEAKHEIGHTS; % Assign height to the PEAKHEIGHTS with the lowest MeanFitError
- bestmodel=model; % Assign bestmodel to the model with the lowest MeanFitError
- end % if min(PEAKHEIGHTS)>0
- end % if MeanFitError<LowestError
- end % for k (NumTrials)
- %
- % Construct model from best-fit parameters
- AA=zeros(NumPeaks,100);
- xxx=linspace(min(xx),max(xx));
- for m=1:NumPeaks,
- switch peakshape
- case 1
- AA(m,:)=gaussian(xxx,FitParameters(2*m-1),FitParameters(2*m));
- case 2
- AA(m,:)=lorentzian(xxx,FitParameters(2*m-1),FitParameters(2*m));
- case 3
- AA(m,:)=logistic(xxx,FitParameters(2*m-1),FitParameters(2*m));
- case 4
- AA(m,:)=pearson(xxx,FitParameters(2*m-1),FitParameters(2*m),extra);
- case 5
- AA(m,:)=expgaussian(xxx,FitParameters(2*m-1),FitParameters(2*m),-extra*length(xxx)./length(xx))';
- case 6
- AA(m,:)=gaussian(xxx,FitParameters(m),FitParameters(NumPeaks+1));
- case 7
- AA(m,:)=lorentzian(xxx,FitParameters(m),FitParameters(NumPeaks+1));
- case 8
- AA(m,:)=expgaussian(xxx,FitParameters(m),FitParameters(NumPeaks+1),-extra*length(xxx)./length(xx))';
- case 9
- AA(m,:)=exppulse(xxx,FitParameters(2*m-1),FitParameters(2*m));
- case 10
- AA(m,:)=sigmoid(xxx,FitParameters(2*m-1),FitParameters(2*m));
- otherwise
- end % switch
- end % for
- % Multiplies each row by the corresponding amplitude and adds them up
- heightsize=size(height');
- AAsize=size(AA);
- if heightsize(2)==AAsize(1),
- mmodel=height'*AA;
- else
- mmodel=height*AA;
- end
- % Top half of the figure shows original signal and the fitted model.
- subplot(2,1,1);plot(xx+xoffset,yy,'b.'); % Plot the original signal in blue dots
- hold on
- for m=1:NumPeaks,
- plot(xxx+xoffset,height(m)*AA(m,:),'g') % Plot the individual component peaks in green lines
- area(m)=trapz(xxx+xoffset,height(m)*AA(m,:)); % Compute the area of each component peak using trapezoidal method
- yi(m,:)=height(m)*AA(m,:); % (NEW) Place y values of individual model peaks into matrix yi
- end
- xi=xxx+xoffset; % (NEW) Place the x-values of the individual model peaks into xi
- % Mark starting peak positions with vertical dashed lines
- for marker=1:NumPeaks,
- markx=BestStart((2*marker)-1);
- subplot(2,1,1);plot([markx+xoffset markx+xoffset],[0 max(yy)],'m--')
- end % for
- plot(xxx+xoffset,mmodel,'r'); % Plot the total model (sum of component peaks) in red lines
- hold off;
- axis([min(xx)+xoffset max(xx)+xoffset min(yy) max(yy)]);
- switch AUTOZERO,
- case 0
- title('Peakfit 2.2. Autozero OFF.')
- case 1
- title('Peakfit 2.2. Linear autozero.')
- case 2
- title('Peakfit 2.2. Quadratic autozero.')
- end
- if peakshape==4||peakshape==5||peakshape==8, % Shapes with Extra factor
- xlabel(['Peaks = ' num2str(NumPeaks) ' Shape = ' ShapeString ' Error = ' num2str(round(100*LowestError)/100) '% Extra = ' num2str(extra) ] )
- else
- xlabel(['Peaks = ' num2str(NumPeaks) ' Shape = ' ShapeString ' Error = ' num2str(round(100*LowestError)/100) '%' ] )
- end
- % Bottom half of the figure shows the residuals and displays RMS error
- % between original signal and model
- residual=yy-bestmodel;
- subplot(2,1,2);plot(xx+xoffset,residual,'b.')
- axis([min(xx)+xoffset max(xx)+xoffset min(residual) max(residual)]);
- xlabel('Residual Plot')
- % Put results into a matrix, one row for each peak, showing peak index number,
- % position, amplitude, and width.
- for m=1:NumPeaks,
- if m==1,
- if peakshape==6||peakshape==7||peakshape==8, % equal-width peak models
- FitResults=[[round(m) FitParameters(m)+xoffset height(m) abs(FitParameters(NumPeaks+1)) area(m)]];
- else
- FitResults=[[round(m) FitParameters(2*m-1)+xoffset height(m) abs(FitParameters(2*m)) area(m)]];
- end % if peakshape
- else
- if peakshape==6||peakshape==7||peakshape==8, % equal-width peak models
- FitResults=[FitResults ; [round(m) FitParameters(m)+xoffset height(m) abs(FitParameters(NumPeaks+1)) area(m)]];
- else
- FitResults=[FitResults ; [round(m) FitParameters(2*m-1)+xoffset height(m) abs(FitParameters(2*m)) area(m)]];
- end % if peakshape
- end % m==1
- end % for m=1:NumPeaks
- % Display Fit Results on upper graph
- subplot(2,1,1);
- startx=min(xx)+xoffset+(max(xx)-min(xx))./20;
- dxx=(max(xx)-min(xx))./10;
- dyy=(max(yy)-min(yy))./10;
- starty=max(yy)-dyy;
- FigureSize=get(gcf,'Position');
- if peakshape==9||peakshape==10,
- text(startx,starty+dyy/2,['Peak # tau1 Height tau2 Area'] );
- else
- text(startx,starty+dyy/2,['Peak # Position Height Width Area'] );
- end
- % Display FitResults using sprintf
- for peaknumber=1:NumPeaks,
- for column=1:5,
- itemstring=sprintf('%0.4g',FitResults(peaknumber,column));
- xposition=startx+(1.7.*dxx.*(column-1).*(600./FigureSize(3)));
- yposition=starty-peaknumber.*dyy.*(400./FigureSize(4));
- text(xposition,yposition,itemstring);
- end
- end
- % ----------------------------------------------------------------------
- function start=calcstart(xx,NumPeaks,xoffset)
- n=max(xx)-min(xx);
- start=[];
- startpos=[n/(NumPeaks+1):n/(NumPeaks+1):n-(n/(NumPeaks+1))]+min(xx);
- for marker=1:NumPeaks,
- markx=startpos(marker)+ xoffset;
- start=[start markx n/5];
- end % for marker
- % ----------------------------------------------------------------------
- function [index,closestval]=val2ind(x,val)
- % Returns the index and the value of the element of vector x that is closest to val
- % If more than one element is equally close, returns vectors of indicies and values
- % Tom O'Haver (toh@umd.edu) October 2006
- % Examples: If x=[1 2 4 3 5 9 6 4 5 3 1], then val2ind(x,6)=7 and val2ind(x,5.1)=[5 9]
- % [indices values]=val2ind(x,3.3) returns indices = [4 10] and values = [3 3]
- dif=abs(x-val);
- index=find((dif-min(dif))==0);
- closestval=x(index);
- % ----------------------------------------------------------------------
- function err = fitgaussian(lambda,t,y)
- % Fitting function for a Gaussian band signal.
- global PEAKHEIGHTS
- numpeaks=round(length(lambda)/2);
- A = zeros(length(t),numpeaks);
- for j = 1:numpeaks,
- A(:,j) = gaussian(t,lambda(2*j-1),lambda(2*j))';
- end
- PEAKHEIGHTS = abs(A\y');
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function err = fitewgaussian(lambda,t,y)
- % Fitting function for a Gaussian band signal with equal peak widths.
- global PEAKHEIGHTS
- numpeaks=round(length(lambda)-1);
- A = zeros(length(t),numpeaks);
- for j = 1:numpeaks,
- A(:,j) = gaussian(t,lambda(j),lambda(numpeaks+1))';
- end
- PEAKHEIGHTS = abs(A\y');
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function err = fitgaussianfw(lambda,t,y)
- % Fitting function for a Gaussian band signal with fixed peak widths.
- global PEAKHEIGHTS
- numpeaks=round(length(lambda)-1);
- A = zeros(length(t),numpeaks);
- for j = 1:numpeaks,
- A(:,j) = gaussian(t,lambda(j),lambda(numpeaks+1))';
- end
- PEAKHEIGHTS = abs(A\y');
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function err = fitlorentziancw(lambda,t,y)
- % Fitting function for a Lorentzian band signal with equal peak widths.
- global PEAKHEIGHTS
- numpeaks=round(length(lambda)-1);
- A = zeros(length(t),numpeaks);
- for j = 1:numpeaks,
- A(:,j) = lorentzian(t,lambda(j),lambda(numpeaks+1))';
- end
- PEAKHEIGHTS = abs(A\y');
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function g = gaussian(x,pos,wid)
- % gaussian(X,pos,wid) = gaussian peak centered on pos, half-width=wid
- % X may be scalar, vector, or matrix, pos and wid both scalar
- % Examples: gaussian([0 1 2],1,2) gives result [0.5000 1.0000 0.5000]
- % plot(gaussian([1:100],50,20)) displays gaussian band centered at 50 with width 20.
- g = exp(-((x-pos)./(0.6005615.*wid)) .^2);
- % ----------------------------------------------------------------------
- function err = fitlorentzian(lambda,t,y)
- % Fitting function for single lorentzian, lambda(1)=position, lambda(2)=width
- % Fitgauss assumes a lorentzian function
- global PEAKHEIGHTS
- A = zeros(length(t),round(length(lambda)/2));
- for j = 1:length(lambda)/2,
- A(:,j) = lorentzian(t,lambda(2*j-1),lambda(2*j))';
- end
- PEAKHEIGHTS = A\y';
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function g = lorentzian(x,position,width)
- % lorentzian(x,position,width) Lorentzian function.
- % where x may be scalar, vector, or matrix
- % position and width scalar
- % T. C. O'Haver, 1988
- % Example: lorentzian([1 2 3],2,2) gives result [0.5 1 0.5]
- g=ones(size(x))./(1+((x-position)./(0.5.*width)).^2);
- % ----------------------------------------------------------------------
- function err = fitlogistic(lambda,t,y)
- % Fitting function for logistic, lambda(1)=position, lambda(2)=width
- % between the data and the values computed by the current
- % function of lambda. Fitlogistic assumes a logistic function
- % T. C. O'Haver, May 2006
- global PEAKHEIGHTS
- A = zeros(length(t),round(length(lambda)/2));
- for j = 1:length(lambda)/2,
- A(:,j) = logistic(t,lambda(2*j-1),lambda(2*j))';
- end
- PEAKHEIGHTS = A\y';
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function g = logistic(x,pos,wid)
- % logistic function. pos=position; wid=half-width (both scalar)
- % logistic(x,pos,wid), where x may be scalar, vector, or matrix
- % pos=position; wid=half-width (both scalar)
- % T. C. O'Haver, 1991
- n = exp(-((x-pos)/(.477.*wid)) .^2);
- g = (2.*n)./(1+n);
- % ----------------------------------------------------------------------
- function err = fitlognormal(lambda,t,y)
- % Fitting function for lognormal, lambda(1)=position, lambda(2)=width
- % between the data and the values computed by the current
- % function of lambda. Fitlognormal assumes a lognormal function
- % T. C. O'Haver, May 2006
- global PEAKHEIGHTS
- A = zeros(length(t),round(length(lambda)/2));
- for j = 1:length(lambda)/2,
- A(:,j) = lognormal(t,lambda(2*j-1),lambda(2*j))';
- end
- PEAKHEIGHTS = A\y';
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function g = lognormal(x,pos,wid)
- % lognormal function. pos=position; wid=half-width (both scalar)
- % lognormal(x,pos,wid), where x may be scalar, vector, or matrix
- % pos=position; wid=half-width (both scalar)
- % T. C. O'Haver, 1991
- g = exp(-(log(x/pos)/(0.01.*wid)) .^2);
- % ----------------------------------------------------------------------
- function err = fitpearson(lambda,t,y,shapeconstant)
- % Fitting functions for a Pearson 7 band signal.
- % T. C. O'Haver (toh@umd.edu), Version 1.3, October 23, 2006.
- global PEAKHEIGHTS
- A = zeros(length(t),round(length(lambda)/2));
- for j = 1:length(lambda)/2,
- A(:,j) = pearson(t,lambda(2*j-1),lambda(2*j),shapeconstant)';
- end
- PEAKHEIGHTS = A\y';
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function g = pearson(x,pos,wid,m)
- % Pearson VII function.
- % g = pearson7(x,pos,wid,m) where x may be scalar, vector, or matrix
- % pos=position; wid=half-width (both scalar)
- % m=some number
- % T. C. O'Haver, 1990
- g=ones(size(x))./(1+((x-pos)./((0.5.^(2/m)).*wid)).^2).^m;
- % ----------------------------------------------------------------------
- function err = fitexpgaussian(lambda,t,y,timeconstant)
- % Fitting functions for a exponentially-broadened Gaussian band signal.
- % T. C. O'Haver, October 23, 2006.
- global PEAKHEIGHTS
- A = zeros(length(t),round(length(lambda)/2));
- for j = 1:length(lambda)/2,
- A(:,j) = expgaussian(t,lambda(2*j-1),lambda(2*j),timeconstant);
- end
- PEAKHEIGHTS = A\y';
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function err = fitexpewgaussian(lambda,t,y,timeconstant)
- % Fitting function for exponentially-broadened Gaussian bands with equal peak widths.
- global PEAKHEIGHTS
- numpeaks=round(length(lambda)-1);
- A = zeros(length(t),numpeaks);
- for j = 1:numpeaks,
- A(:,j) = expgaussian(t,lambda(j),lambda(numpeaks+1),timeconstant);
- end
- PEAKHEIGHTS = abs(A\y');
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function g = expgaussian(x,pos,wid,timeconstant)
- % Exponentially-broadened gaussian(x,pos,wid) = gaussian peak centered on pos, half-width=wid
- % x may be scalar, vector, or matrix, pos and wid both scalar
- % T. C. O'Haver, 2006
- g = exp(-((x-pos)./(0.6005615.*wid)) .^2);
- g = ExpBroaden(g',timeconstant);
- % ----------------------------------------------------------------------
- function yb = ExpBroaden(y,t)
- % ExpBroaden(y,t) convolutes y by an exponential decay of time constant t
- % by multiplying Fourier transforms and inverse transforming the result.
- fy=fft(y);
- a=exp(-[1:length(y)]./t);
- fa=fft(a);
- fy1=fy.*fa';
- yb=real(ifft(fy1))./sum(a);
- % ----------------------------------------------------------------------
- function err = fitexppulse(tau,x,y)
- % Iterative fit of the sum of exponental pulses
- % of the form Height.*exp(-tau1.*x).*(1-exp(-tau2.*x)))
- global PEAKHEIGHTS
- A = zeros(length(x),round(length(tau)/2));
- for j = 1:length(tau)/2,
- A(:,j) = exppulse(x,tau(2*j-1),tau(2*j));
- end
- PEAKHEIGHTS =abs(A\y');
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function g = exppulse(x,t1,t2)
- % Exponential pulse of the form
- % Height.*exp(-tau1.*x).*(1-exp(-tau2.*x)))
- e=(x-t1)./t2;
- p = 4*exp(-e).*(1-exp(-e));
- p=p .* [p>0];
- g = p';
- % ----------------------------------------------------------------------
- function err = fitsigmoid(tau,x,y)
- % Fitting function for iterative fit to the sum of
- % sigmiods of the form Height./(1 + exp((t1 - t)/t2))
- global PEAKHEIGHTS
- A = zeros(length(x),round(length(tau)/2));
- for j = 1:length(tau)/2,
- A(:,j) = sigmoid(x,tau(2*j-1),tau(2*j));
- end
- PEAKHEIGHTS = A\y';
- z = A*PEAKHEIGHTS;
- err = norm(z-y');
- % ----------------------------------------------------------------------
- function g=sigmoid(x,t1,t2)
- g=1./(1 + exp((t1 - x)./t2))';






















 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








