第三章无监督学习与预处理
第二章监督学习中介绍了不少训练模型,这些模型都需要喂数据,调试参数以达到预想结果,
由于模型理解相对较复杂,因此使用独立章节学习总结.
而第三章内容衔接比较紧凑因此合并学习.
第二章内容属于监督学习部分,都需要人工参与分析选择模型.
第三章内容属于无监督学习部分,核心是围绕着如何自动分析数据展开.
第三章共分3大部分
- 数据缩放(属于通用的数据处理方式)
第二章在学习线性模型的时候有接触过,主要是讲解如何将差异很大的数据特征都调校到同一水平.
这部分的内容可以同时应用在训练及测试数据上. - 数据变换(解构数据)
主要内容讲解如何降低数据纬度,如何提取数据成分,如何将高维数据可视化.
用于算法的独特性,这部分内容只能应用在训练数据上. - 数据凝聚(聚类)
(2)在提取数据的过程会删掉不重要的数据特征,而(3)会保留数据特征
主要内容是如何将数据按分类分组(簇).
同样的这部分的算法也这能用在训练数据之上.
开始学习之前,我们在脑海里先回顾一下第二章学习的那些模型,
都是一个通用模式: 类实例化 -> fit(训练数据) -> transform(训练数据/测试数据)
通俗来说就是构建模型,配置调校模型,应用模型.
书上给出一种官方的说法:
这些模型统一称作:估计器(estimator)
- 所有的估计器中都包含算法,同时也保存了利用算法从数据中学到的模型.
- 都有的估计器都有fit方法, 第一参数永远是x数据(NumPy 数组或SciPy 稀疏矩阵)
每一行代表一个数据点. 还需要一个目标参数y, - 应用模型主要有两种方法:
- 想要创建一个新的y, 比如predict
- 想要创建一个新的x的表示 比如transform
- 所有的模型都有source(x,y),可以评估模型.
01 无监督学习的类型
- 变换(dimensionality reduction)
降维数据,从复杂数据中筛选出数据的主要部分 - 聚类(clustering algorithm)
按照某些相似特征将数据进行分组
02 数据预处理 数据缩放
以直观的方式观察缩放后的数据表现.
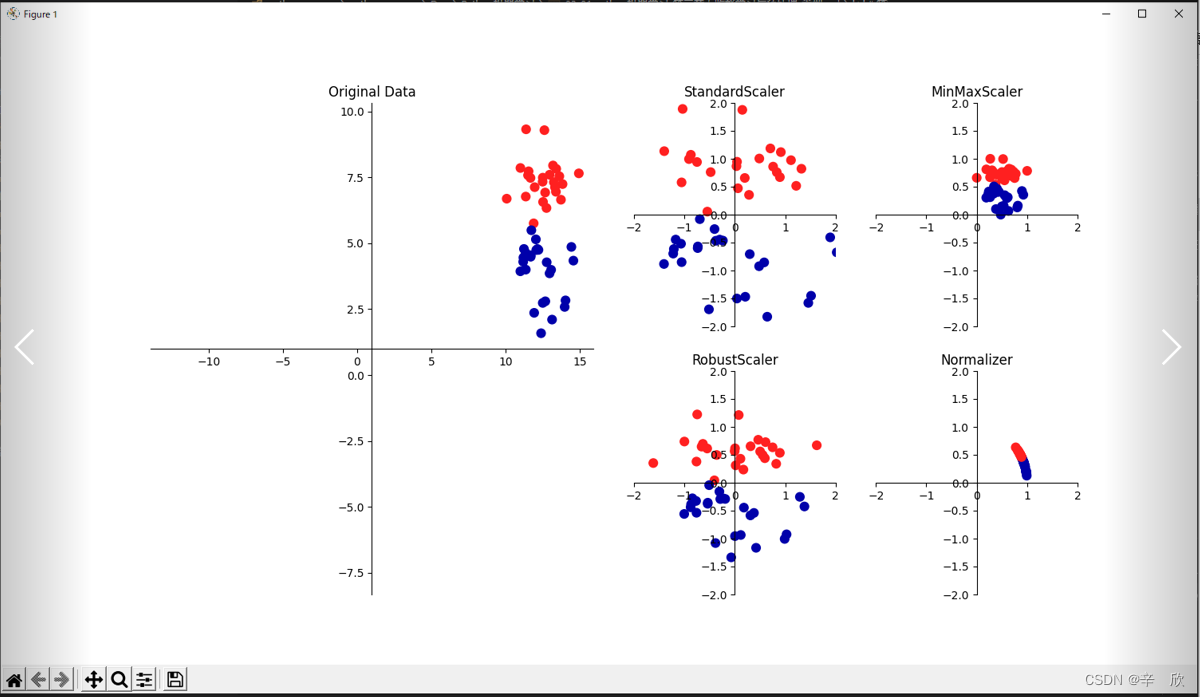
# 数据预处理_数据缩放种类快速演示
# 1. 左图为原始数据样本 数据范围 X: 0~15, Y: 0~10.
# 2. 右图4张为缩放后样本
# 1. 上左(StandardScaler): 每个特征的平均值为0,方差调整为1.由于强调的是整体,因此对于最大最小值可能不会太友好.
# 2. 上右(MinMaxScaler): 将特征都移动到X,Y都是0~1的矩阵范围内(想象一下离远点看数据,或将XY轴的测量单位扩大几倍).
# 3. 下左(RobustSclaer): 跟1差不多, 但是它用的是 中位数或四分位数.
# 4. 下右(Normalizer): 调整欧式长度为1(想象一下半径为1的球体)
# 中位数: 指有那么一个数可以让整体的数据一半比它大一半比它小.
# 四分位数: 跟中位数类似, 不过它不是一半而是1/4.
import mglearn
import matplotlib.pyplot as plt
mglearn.plots.plot_scaling()
plt.show()
02_01 MinMaxScaler缩放原理
函数帮助中介绍的原理:
X_std = (X - X.min(axis=0)) / (X.max(axis=0) - X.min(axis=0))
X_scaled = X_std * (max - min) + min
# 数据预处理_MinMaxScaler缩放原理
# 数据样本源
from re import M
from sklearn.datasets import load_breast_cancer
# 数据拆分
from sklearn.model_selection import train_test_split
# 用于将数据缩放到 X,Y 为1的矩阵中
from sklearn.preprocessing import MinMaxScaler
# 数据准备
cancer = load_breast_cancer()
X_train, X_test, y_train, y_test = train_test_split(
cancer.data, cancer.target, random_state=1)
# 缩放类准备
# 原理:
# The transformation is given by::
# X_std = (X - X.min(axis=0)) / (X.max(axis=0) - X.min(axis=0))
# X_scaled = X_std * (max - min) + min
scaler = MinMaxScaler()
# 不同与模型的fit函数, 缩放函数之填充训练数据就行.
# 因为缩放只关心源,不关心目标类别
scaler.fit(X_train)
# 这个有点奇怪,直接实例化然后变换就行了呗,为啥中间还要喂一遍数据?
# 看函数帮助也是这么用的.
# fit函数可能的作用是先预读数据样本,确定样本中一些内容,不如个数,最大,最小值等等,
# 以此来确定数据的轮廓,在转换时会参照这个轮廓来变换数据.理论上喂的数据不用跟变换数据必须一致.
X_train_scaler = scaler.transform(X_train)
# 缩放不会改变结构,只会改变大小
# In [19]: X_train_scaler.shape
# Out[19]: (426, 30)
# In [20]: X_train.shape
# Out[20]: (426, 30)
# In [41]: X_train.min(axis=0)
# Out[41]:
# array([6.981e+00, 9.710e+00, 4.379e+01, 1.435e+02, 5.263e-02, 1.938e-02,
# 0.000e+00, 0.000e+00, 1.060e-01, 5.024e-02, 1.153e-01, 3.602e-01,
# 7.570e-01, 6.802e+00, 1.713e-03, 2.252e-03, 0.000e+00, 0.000e+00,
# 9.539e-03, 8.948e-04, 7.930e+00, 1.202e+01, 5.041e+01, 1.852e+02,
# 7.117e-02, 2.729e-02, 0.000e+00, 0.000e+00, 1.566e-01, 5.521e-02])
# In [42]: X_train_scaler.min(axis=0)
# Out[42]:
# array([0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.,
# 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0., 0.])
# In [43]: X_train.max(axis=0)
# Out[43]:
# array([2.811e+01, 3.928e+01, 1.885e+02, 2.501e+03, 1.634e-01, 2.867e-01,
# 4.268e-01, 2.012e-01, 3.040e-01, 9.575e-02, 2.873e+00, 4.885e+00,
# 2.198e+01, 5.422e+02, 3.113e-02, 1.354e-01, 3.960e-01, 5.279e-02,
# 6.146e-02, 2.984e-02, 3.604e+01, 4.954e+01, 2.512e+02, 4.254e+03,
# 2.226e-01, 9.379e-01, 1.170e+00, 2.910e-01, 5.774e-01, 1.486e-01])
# In [44]: X_train_scaler.max(axis=0)
# Out[44]:
# array([1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1.,
# 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1., 1.])
X_test_scaler = scaler.transform(X_test)
# 对于测试数据达不到完全缩放. 结果可以看出有的小于0,大于1,不如训练数据缩放的结果完美.
# In [46]: X_test_scaler.min(axis=0)
# Out[46]:
# array([ 0.0336031 , 0.0226581 , 0.03144219, 0.01141039, 0.14128374,
# 0.04406704, 0. , 0. , 0.1540404 , -0.00615249,
# -0.00137796, 0.00594501, 0.00430665, 0.00079567, 0.03919502,
# 0.0112206 , 0. , 0. , -0.03191387, 0.00664013,
# 0.02660975, 0.05810235, 0.02031974, 0.00943767, 0.1094235 ,
# 0.02637792, 0. , 0. , -0.00023764, -0.00182032])
# In [47]: X_test_scaler.max(axis=0)
# Out[47]:
# array([0.9578778 , 0.81501522, 0.95577362, 0.89353128, 0.81132075,
# 1.21958701, 0.87956888, 0.9333996 , 0.93232323, 1.0371347 ,
# 0.42669616, 0.49765736, 0.44117231, 0.28371044, 0.48703131,
# 0.73863671, 0.76717172, 0.62928585, 1.33685792, 0.39057253,
# 0.89612238, 0.79317697, 0.84859804, 0.74488793, 0.9154725 ,
# 1.13188961, 1.07008547, 0.92371134, 1.20532319, 1.63068851])
02_02 MinMaxScaler缩放前后对比
# 数据预处理_MinMaxScaler缩放前后对比
import mglearn
import matplotlib.pyplot as plt
# 生成聚类数据
from sklearn.datasets import make_blobs
from sklearn.model_selection import train_test_split
# 缩放类
from sklearn.preprocessing import MinMaxScaler
# 讲解数据缩放并不关心种类目标,因此没有y
# make_blobs 用于生成聚类数据
# n_samples 创建的样本个数
# centers 目标种类个数 本例将生成5各类别的数据
# random_state 随机种子用于打乱数据
# cluster_std 标准偏差 默认是1
X, _ = make_blobs(n_samples=50, centers=5, random_state=4, cluster_std=2)
X_train, X_test = train_test_split(X, random_state=5, test_size=.1)
#######################
# 第一个画布,使用原始数据绘制散点图
#######################
# 绘制训练集和测试集
fig, axes = plt.subplots(1, 3, figsize=(13, 4))
axes[0].scatter(X_train[:, 0], X_train[:, 1],
c=mglearn.cm2(0), label="Training set", s=60)
axes[0].scatter(X_test[:, 0], X_test[:, 1], marker='^',
c=mglearn.cm2(1), label="Test set", s=60)
axes[0].legend(loc='upper left')
axes[0].set_title("Original Data")
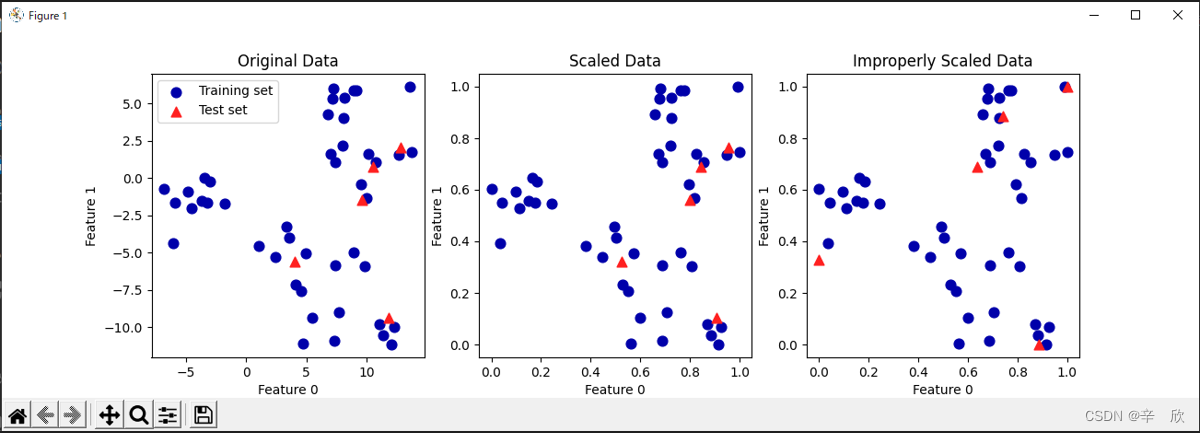
#######################
# 第二个画布,同时缩放训练数据和测试数据
#######################
# 因为使用了同时缩放,因此即使标尺改变也不影响预测结果
# 从图中可以看出,散点图与原始图接近
# 利用MinMaxScaler缩放数据
scaler = MinMaxScaler()
# 缩放都是先fit在transform,其实有个方法是fit_transform可以整合这俩个方法,而且运行效率较高
# 例如: scaler.fit_transform(X_train) 来简化步骤
scaler.fit(X_train)
X_train_scaled = scaler.transform(X_train)
X_test_scaled = scaler.transform(X_test)
# 将正确缩放的数据可视化
axes[1].scatter(X_train_scaled[:, 0], X_train_scaled[:, 1],
c=mglearn.cm2(0), label="Training set", s=60)
axes[1].scatter(X_test_scaled[:, 0], X_test_scaled[:, 1], marker='^',
c=mglearn.cm2(1), label="Test set", s=60)
axes[1].set_title("Scaled Data")
#######################
# 第三个画布,只对测试数据进行缩放
#######################
# 要么就全缩放,要么就全不缩放,只缩放一部分相当于改变了衡量标准,
# 因此模型会认为其中一部分数据有重大偏差进而影响预测结果.
test_scaler = MinMaxScaler()
test_scaler.fit(X_test)
X_test_scaled_badly = test_scaler.transform(X_test)
# 将错误缩放的数据可视化
axes[2].scatter(X_train_scaled[:, 0], X_train_scaled[:, 1],
c=mglearn.cm2(0), label="training set", s=60)
axes[2].scatter(X_test_scaled_badly[:, 0], X_test_scaled_badly[:, 1],
marker='^', c=mglearn.cm2(1), label="test set", s=60)
axes[2].set_title("Improperly Scaled Data")
for ax in axes:
ax.set_xlabel("Feature 0")
ax.set_ylabel("Feature 1")
plt.show()
02_03 数据缩放对模型的影响
为了便于较明显的说明效果,我们使用对缩放比较敏感的核支持向量机模型来进行实验.
# 数据变换_缩放对模型的影响
from sklearn.model_selection import train_test_split
from sklearn.datasets import load_breast_cancer
from sklearn.svm import SVC
# 将数据缩放到 x,y为1的矩阵中
from sklearn.preprocessing import MinMaxScaler
# 特征平均为0, 方差为1
from sklearn.preprocessing import StandardScaler
# 数据准备
cancer = load_breast_cancer()
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target,
random_state=0)
svm = SVC(C=100)
svm.fit(X_train, y_train)
print("Test set accuracy: {:.2f}".format(svm.score(X_test, y_test)))
# Test set accuracy: 0.94
#########################
# 使用MinMaxScaler缩放模型
#########################
scaler = MinMaxScaler()
scaler.fit(X_train)
X_train_scaled = scaler.transform(X_train)
X_test_scaled = scaler.transform(X_test)
svm.fit(X_train_scaled, y_train)
print("Scaled test set accuracy: {:.2f}".format(
svm.score(X_test_scaled, y_test)))
# Scaled test set accuracy: 0.97
#########################
# 使用StandardScaler缩放模型
#########################
scaler = StandardScaler()
scaler.fit(X_train)
X_train_scaled = scaler.transform(X_train)
X_test_scaled = scaler.transform(X_test)
# 在缩放后的训练数据上学习SVM
svm.fit(X_train_scaled, y_train)
# 在缩放后的测试集上计算分数
print("SVM test accuracy: {:.2f}".format(svm.score(X_test_scaled, y_test)))
# SVM test accuracy: 0.96
03 数据变换_主成分分析PCA
变换数据的一种方式: PCA(principal components analysis)
特点:
- PCA重在找出主要成分
- PCA不关心数据方向
- PCA可以自己降维数据提取主成分
- 越重要的成分排名越靠前
结合以上特点,我觉的PCA适合模糊匹配(因为不关心方向,因此有偏移没关系),并且能找到最像的结果.
顺便说一下,书中的例子是用PCA来找人脸.
为了好理解,举个例子:
训练数据使用人脸正面靠镜框左面的图片,测试数据使用靠右面的图片,PCA就能很容易的识别出来.
# 数据变换_PCA 特征提取快速演示
# 特征提取的代码虽然很少但是信息量很大
# 特征提取用的最多的就是 主成分分析(principal component analysis 简称PCA)
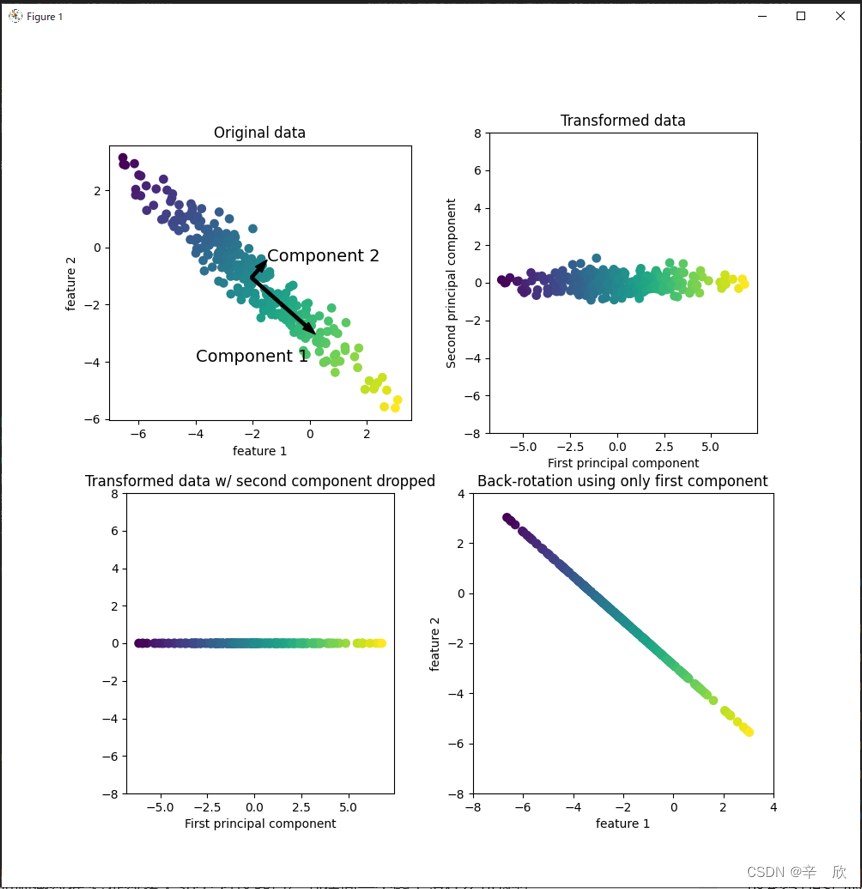
# PCA算法的步骤是:
# 1. 寻找主要数据,标记主数据的方向.
# 同过计算方差,找到方差最大的方向,将其标记成主数据.
# 主数据的方向应当是包含数据最多且特征之间最为相关.
# 2. 沿着主数据的垂直方向寻找信息最多的方向.
# 二维空间对于x轴的垂直方向就是y轴,但是在空间上与x轴垂直的方向可是有很多.
# 模型负责在这些方向上找出信息最多的那个.
# 3. 旋转主数据方向,使主数据与x轴重合
# 我猜测模型的预测计算等都是使用标准化的z轴为基础的,因此需要将数据像标准化.
# 找到主数据方向后,整体旋转数据,使主数据方向平行与x轴重合.
# 重合后的主数据,其平局值可能与x轴平行也可能与X轴重合,为了不影响模型判断因此先减去这部分平均值
# 最终得到右上图的效果,主数据方向延x轴延申,且全部以0(Y轴)为中心.
# 4. 降维
# 仅保留一部分主数据,去掉其它非主要数据来达到降维的目的.
# 得到坐下图中一条直线. (是不是线性模型就是这么找方向的呢?)
# 5. 还原数据方向
# 再次旋转数据,使整理后的数据方向与元数据主数据的方向一致.
# 将第三步中临时去掉的平均值再加回来,保持主数据的完整性.最终得到右下图
# 以上使PCA算法的思路,虽然会损失一部分的数据特征(噪点),但是可以找到真正有用的主数据方向
# 需要注意的是主数据不是主特征.主数据表示最能代表数据含义的一组或多组数据特征集合.
import mglearn
from matplotlib import pyplot as plt
mglearn.plots.plot_pca_illustration()
plt.show()
03_01 不使用数据缩放的多特征数据如何表示
首先多特征数据不能用散点图来表示,不是实现不了而是实现了无法直观的看到结果.
例如肿瘤数据,特征数多大30个,将这些点绘制在同一个图上没什么可读性.
在为应用PCA之前,有一种折中解决方案,虽然也很乱,但是至少具备了可读性.
肿瘤数据虽然特征数众多,但是类别只有良性跟恶性两种.
因此可以画出每一个特征的良性和恶性结果,图像表示上选用直方图(较散点图直观),
查找重合部分最少的那个特征(重合少代表类别区分明显),即为PCA中的主成分.
# 数据变换_多特征&无缩放如何表示
import mglearn
import matplotlib.pyplot as plt
import numpy as np
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
fig, axes = plt.subplots(15, 2, figsize=(10, 20))
malignant = cancer.data[cancer.target == 0]
benign = cancer.data[cancer.target == 1]
# 将绘图结构变成线性结构
ax = axes.ravel()
for i in range(30):
# histogram 在numpy下用于表示数据的直方图
# 函数帮助参照: https://www.cjavapy.com/article/1103/
# 其它的参数函数说明介绍的很清楚了,这里单独讲一下bins参数
# bins: 直方图中柱状图的个数
# 有三种入力模式, 数字,序列,字符串.这里只讲前两种
# 数字: 柱状图个数,首先会取得数组集合中的虽大值最小值作为边界,然后通过linspace来自动填充
# 使得均匀分布的柱状个数=bins值
# 序列: 跟上面的差不多,区别在于柱状图的范围完全由序列指定.
# 例子(数字):
# bin为数字时,个数小于类别个数的时候,会在最小,最大范围内,自己寻找均等位置
#
# In [21]: np.histogram([1, 2, 1], bins=1)
# Out[21]: (array([3], dtype=int64), array([1., 2.]))
#
# In [23]: np.histogram([1, 2, 1,8], bins=2)
# Out[23]: (array([3, 1], dtype=int64), array([1. , 4.5, 8. ]))
#
# bin的值在histogram中计算时是 bin+1.
# In [50]: np.linspace(1,8, 3)
# Out[50]: array([1. , 4.5, 8. ])
# 例子(序列):
# In [46]: np.histogram([1, 2, 1,8,4], bins=[2,3,6,8])
# Out[46]: (array([1, 1, 1], dtype=int64), array([2, 3, 6, 8]))
# 因为指定了返回值范围,因此只能返回范围内找到的值
# 范围2~3, 找到2, 返回个数1
# 范围3~6, 找到4, 返回个数1
# 范围6~8, 找到8, 返回个数1
# 因此返回的是array([1, 1, 1]
# 把范围最小值1设置上,看看结果
# In [51]: np.histogram([1, 2, 1,8,4], bins=[1,2,3,6,8])
# Out[51]: (array([2, 1, 1, 1], dtype=int64), array([1, 2, 3, 6, 8]))
# 这个值已经能够看懂了吧.
# 通过上面的介绍,基本了解了函数是做什么用的,接下来会到这个例子
# 这个函数的第一个返回值是 概率或者个数对接下来的画图没有用因此用"_"占位,直接舍弃
# 第二个参数返回的是柱状图每个柱的范围,是画图中需要用到的.
_, bins = np.histogram(cancer.data[:, i], bins=50)
# 上面np中的hitogram是为了造柱子的轮廓数据,绘图类ax中的hist是实际的画图
# 分别用良性/恶性样本中的每个特征数据来画柱状图
ax[i].hist(malignant[:, i], bins=bins, color=mglearn.cm3(0), alpha=.5)
ax[i].hist(benign[:, i], bins=bins, color=mglearn.cm3(2), alpha=.5)
ax[i].set_title(cancer.feature_names[i])
ax[i].set_yticks(())
ax[0].set_xlabel("Feature magnitude")
ax[0].set_ylabel("Frequency")
ax[0].legend(["malignant", "benign"], loc="best")
# 紧凑显示画布上的绘图
fig.tight_layout()
plt.show()
 上图中,柱状图交集越少,边界越清晰,越容易判断分类.比如: 结果中的"worst concave points”.
上图中,柱状图交集越少,边界越清晰,越容易判断分类.比如: 结果中的"worst concave points”.
03_02 使用PCA表示多特征数据
# 数据变换_PCA 分析多特征
import mglearn
import matplotlib.pyplot as plt
# 主成分分析库
from sklearn.decomposition import PCA
from sklearn.datasets import load_breast_cancer
from sklearn.preprocessing import StandardScaler
#####################
# 准备数据,并进行数据的缩放
#####################
cancer = load_breast_cancer()
scaler = StandardScaler()
scaler.fit(cancer.data)
X_scaled = scaler.transform(cancer.data)
#####################
# 实例化PCA,指定保留2个重要主成分
#####################
# 跟快速演示中的不太一样,看样子PCA只是用于提取,并没有缩放功能,缩放需要自己单独写
pca = PCA(n_components=2)
# 初始化数据分析轮廓
pca.fit(X_scaled)
# compents_ 属性中存放有序主成分,越重要的主成分越靠前
# In [4]: pca.components_.shape
# Out[4]: (2, 30)
# 提取主成分
X_pca = pca.transform(X_scaled)
print("Original shape: {}".format(str(X_scaled.shape)))
print("Reduced shape: {}".format(str(X_pca.shape)))
# Original shape: (569, 30)
# Reduced shape: (569, 2)
# 成功提取出来俩个特征
#####################
# 画图 主成分的散点图
#####################
plt.figure(figsize=(8, 8))
mglearn.discrete_scatter(X_pca[:, 0], X_pca[:, 1], cancer.target)
plt.legend(cancer.target_names, loc="best")
plt.gca().set_aspect("equal")
plt.xlabel("First principal component")
plt.ylabel("Second principal component"<







 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 1188
1188











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








