约束条件:
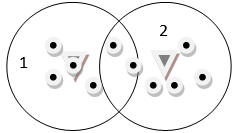
对于某一区域的点的任务点,要对该区域中的任务进行打包,首先算出中心点hi到其余点zj的距离aij;aij中i为该区域有i个打包中心点数,每个打包内除中心点外,还有个j其余点;s为其余点到打包中心点的距离上限;D为其余点与打包点的关系,如果在给定范围以内的取值为1,表示可以打包为一类,为0则不打包;A为一个打包内的所有点距之和。打包示意图如下:
4.3 免疫算法打包程序实现的步骤
Step1:初始化参数并产生初始打包中心点,即初始种群;
Step2:对每个打包内的点进行距离分析;
Step3:分析每个点对所有打包中心点的距离,每个点都可能处于多个打包范围内;
Step4:通过比对步骤2与3中的距离,对于每个任务点而言,优先选择距离最近的一个中心点,并记录;
Step5:判断是否每个点都给予了分类,并检验是否满足条件,是则结束继续步骤6;
Step6:产生新群体,基于步骤4的计算结果进行遗传方式产生新的打包群体;
Step7:比对前一个打包群体,对比各个打包内的A值,保留最小的群体,并循环步骤4-7;
Step8:输出结果。
4.4 Matlab代码
clear
clc
%% 位置坐标
city_coordinate=[1304,2312;3639,1315;4177,2244;3712,1399;3488,1535;3326,1556;3238,1229;4196,1044;4312,790;4386,570;
3007,1970;2562,1756;2788,1491;2381,1676;1332,695;3715,1678;3918,2179;4061,2370;3780,2212;3676,2578;
4029,2838;4263,2931;3429,1908;3507,2376;3394,2643;3439,3201;2935,3240;3140,3550;2545,2357;2778,2826;2370,2975];
n=length(city_coordinate);
%% step1 参数设置
sizepop=10; % 种群规模
overbest=10; % 记忆库容量大小
MAXGEN=100; % 迭代次数
pcross=0.5; % 免疫细胞遗传过程中的交叉概率
pmutation=0.4; % 免疫细胞遗传过程中的变异概率
s=3; %记录最优个体数
ps=0.95; % 多样性评价参数(一般设置为0.95)
len=10; % 最大打包数
jiange=1000; %打包点之间距离不低于1000m
z=3000; %其余点到中心点距离上限
M=sizepop+overbest;
%% step2 将种群信息定义为一个结构体,方便调用
individuals = struct(‘fitness’,zeros(1,M), ‘concentration’,zeros(1,M),‘excellence’,zeros(1,M),‘chrom’,[]);
%struct函数功能是生成一个具有指定字段名和相应数据的结构数组
%% step3 产生初始抗体种群
individuals.chrom = popinit(city_coordinate,M,len,jiange);%“.chrom”是调用individuals数组
trace=[]; %初始化矩阵,用于记录每代最个体优适应度和平均适应度
%% 迭代寻优
for iii=1:MAXGEN
%% step4 抗体群多样性评价
for i=1:M
individuals.fitness(i) = fitness(city_coordinate,individuals.chrom(i,:),z); % 每个个体适应度计算
individuals.concentration(i) = concentration(i,M,individuals); % 计算第i个个体的打包中心点与种群中每个个体的打包中心点间的相似度
end
% 综合亲和度和浓度评价抗体优秀程度,得出繁殖概率
individuals.excellence = excellence(individuals,M,ps);
% 记录当代最佳个体和种群平均适应度
[best,index] = min(individuals.fitness); % 找出当前种群中适应值最小的中心点类,及较优的打包中心点
bestchrom = individuals.chrom(index,:); % 找出最优个体,即min(individuals.fitness)对应的个体
average = mean(individuals.fitness); % 计算平均适应度
trace = [trace;best,average]; % 记录最有个体
%% step5 根据excellence,形成父代群,更新记忆库(加入精英保留策略,可由s控制)
bestindividuals = bestselect(individuals,M,overbest,s); % 更新记忆库
individuals = bestselect(individuals,M,sizepop,s); % 更新父代群
%% step6 选择,交叉,变异操作,再加入记忆库中抗体,产生新种群
individuals = Select(individuals,sizepop); % 选择
individuals.chrom = Cross(pcross,individuals.chrom,sizepop,len,city_coordinate,jiange); % 交叉
individuals.chrom = Mutation(pmutation,individuals.chrom,sizepop,len,n,city_coordinate,jiange); % 变异
individuals = incorporate(individuals,sizepop,bestindividuals,overbest); % 加入记忆库中抗体
end
%% 画出免疫算法收敛曲线
figure(1)
plot(trace(:,1),‘g–’);
hold on
plot(trace(:,2),‘-’);
legend(‘最优适应度值’,‘平均适应度值’)
title(‘免疫算法收敛曲线’,‘fontsize’,12)
xlabel(‘迭代次数’,‘fontsize’,12)
ylabel(‘适应度值’,‘fontsize’,12)
%% 画出打包中心选址图
%找出最近打包点
for i=1:n
distance(i,:)=dist(city_coordinate(i,:),city_coordinate(bestchrom,:)');
end
[a,b]=min(distance’);
index=cell(1,len);
for i=1:len
%记录各个打包中心点的位置
index{i}=find(b==i);
end
figure(2)
title(‘最优打包图’)
cargox=city_coordinate(bestchrom,1);
cargoy=city_coordinate(bestchrom,2);
plot(cargox,cargoy,‘rs’,‘LineWidth’,2,‘MarkerEdgeColor’,‘r’,‘MarkerFaceColor’,‘b’,‘MarkerSize’,20)
hold on
plot(city_coordinate(:,1),city_coordinate(:,2),‘o’,‘LineWidth’,2,‘MarkerEdgeColor’,‘k’,‘MarkerFaceColor’,‘g’,‘MarkerSize’,10)
for i=1:31
x=[city_coordinate(i,1),city_coordinate(bestchrom(b(i)),1)];
y=[city_coordinate(i,2),city_coordinate(bestchrom(b(i)),2)];
plot(x,y,‘c’);hold on
end
%% Step1:初始化参数并产生初始打包中心点,即初始种群;
function pop = popinit(city_coordinate,M,len,jiange)
%种群初始化函数(记忆库库为空,全部随机产生)
% M input 种群数量
% length input 抗体长度
% pop output 初始种群
n=length(city_coordinate);
for i=1:M
flag=0;
while flag==0
aaa=randperm(n);%产生随机序列
pop(i,:)=aaa(1:len);%pop矩阵提取的是b矩阵中的前length个元素
flag=test(city_coordinate,pop(i,:),jiange);%test为条件函数文件,如果满足条件,那么flag就=0,此时终止这次循环
end
end
%% Step2:对每个打包内的点进行距离分析;
function flag=test(city_coordinate,code,jiange)
% 检查个体是否满足距离约束
% code input 个体
% flag output 是否满足要求标志
flag=1;
a=dist( city_coordinate(code,:)');%dist是欧式距离计算函数
a(find(a==0))=[];
b=find(a<jiange);
if isempty(b)==1
flag=0;
end
end
%% Step3:分析每个点对所有打包中心点的距离,每个点都可能处于多个打包范围内;
function fit=fitness(city_coordinate,individual,z)
%计算个体适应度值
%individual input 个体
%fit output 适应度值
%计算出所有打包点到中心点的距离
for i=1:length(city_coordinate)
distance(i,:)=dist(city_coordinate(i,:),city_coordinate(individual,:)');
end
%% Step4:通过比对步骤2与3中的距离,对于每个任务点而言,优先选择距离最近的一个中心点,并记录;
%找出离中心点最近点,a记录最近距离,b记录最近的点
[a,b]=min(distance’);
%如果距离大于z,那么就要加上一个惩罚值:4.0e+4*length,这里自己定,惩罚值尽可能大些,但也别太大
fit=sum(a) + 4.0e+4*length(find(a>z));
end
function concentration = concentration(i,M,individuals)
%% (3)计算个体浓度值
% i input 第i个抗体
% M input 种群规模
% individuals input 个体
% concentration output 浓度值
concentration=0;
for j=1:M
xsd=similar(individuals.chrom(i,:),individuals.chrom(j,:)); % 第i个体与种群所有个体间的相似度
% 相似度大于阀值就说明相似
if xsd>0.7
concentration=concentration+1;
end
end
concentration=concentration/M;%计算相似度
end
%% (2)抗原与抗体之间的亲和力
function resemble = similar(individual1,individual2)
% 计算个体individual1和individual2的相似度
% individual1,individual2 input 两个个体
% resemble output 相似度
k=zeros(1,length(individual1));
for i=1:length(individual1)
if find(individual1(i)==individual2)
k(i)=1;
end
end
resemble=sum(k)/length(individual1);
end
function exc=excellence(individuals,M,ps)
%% (4)计算个体繁殖概率
% individuals input 种群
% M input 种群规模
% ps input 多样性评价参数
% exc output 繁殖概率
fit = 1./individuals.fitness; %Av
sumfit = sum(fit);
con = individuals.concentration; %Cv
sumcon = sum(con);
for i=1:M
exc(i) = fit(i)/sumfitps +con(i)/sumcon(1-ps); %exc为当前每个个体的一个评价值
end
end
%% 初始化记忆库,依据excellence,将群体中高适应度低相似度的overbest个个体存入记忆库
function rets=bestselect(individuals,m,n,s)
% m input 抗体数
% n input 记忆库个体数\父代群规模
% individuals input 抗体群
% bestindividuals output 记忆库\父代群
% 精英保留策略,将fitness最好的s个个体先存起来,避免因其浓度高而被淘汰
rets=struct(‘fitness’,zeros(1,n), ‘concentration’,zeros(1,n),‘excellence’,zeros(1,n),‘chrom’,[]);
[fitness,index] = sort(individuals.fitness);
for i=1:s %s记忆最优个体的数量
rets.fitness(i) = individuals.fitness(index(i));
rets.concentration(i) = individuals.concentration(index(i));
rets.excellence(i) = individuals.excellence(index(i));
rets.chrom(i,:) = individuals.chrom(index(i)😅;
end
% 剩余m-s个个体
leftindividuals=struct(‘fitness’,zeros(1,m-s), ‘concentration’,zeros(1,m-s),‘excellence’,zeros(1,m-s),‘chrom’,[]);
for k=1:m-s
leftindividuals.fitness(k) = individuals.fitness(index(k+s));
leftindividuals.concentration(k) = individuals.concentration(index(k+s));
leftindividuals.excellence(k) = individuals.excellence(index(k+s));
leftindividuals.chrom(k,:) = individuals.chrom(index(k+s)😅;
end
% 将剩余抗体按excellence值排序
[excellence,index]=sort(1./leftindividuals.excellence);
% 在剩余群体中再选n-s个最好的个体,也就是总共筛选出记忆库的最大含量个个体,然后按优劣从上往下排序
for i=s+1:n
rets.fitness(i) = leftindividuals.fitness(index(i-s));
rets.concentration(i) = leftindividuals.concentration(index(i-s));
rets.excellence(i) = leftindividuals.excellence(index(i-s));
rets.chrom(i,:) = leftindividuals.chrom(index(i-s)😅;
end
end
%% 轮盘赌选择
function ret=Select(individuals,sizepop)
% individuals input : 种群信息
% sizepop input : 种群规模
% ret output : 选择后得到的种群
excellence=individuals.excellence;
pselect=excellence./sum(excellence);
% 事实上 pselect = excellence
index=[];
for i=1:sizepop % 转sizepop次轮盘,sizepop为种群数
pick=rand;
for j=1:sizepop
pick=pick-pselect(j);
if pick<0
index=[index j];
break; % 寻找落入的区间,此次转轮盘选中了染色体j
end
end
end
% 注意:在转sizepop次轮盘的过程中,有可能会重复选择某些染色体
individuals.chrom=individuals.chrom(index,:);
individuals.fitness=individuals.fitness(index);
individuals.concentration=individuals.concentration(index);
individuals.excellence=individuals.excellence(index);
ret=individuals;
end
%% 交叉操作
function ret=Cross(pcross,chrom,sizepop,length,city_coordinate,jiange)
% pcorss input : 交叉概率
% chrom input : 抗体群
% sizepop input : 种群规模
% length input : 抗体长度
% ret output : 交叉得到的抗体群
% 每一轮for循环中,可能会进行一次交叉操作,随机选择染色体是和交叉位置,是否进行交叉操作则由交叉概率(continue)控制
for i=1:sizepop
% 随机选择两个染色体进行交叉
pick=rand;
while prod(pick)==0
pick=rand(1);
end
if pick>pcross
continue;
end
% 找出交叉个体
index(1)=unidrnd(sizepop);
index(2)=unidrnd(sizepop);
while index(2)==index(1)
index(2)=unidrnd(sizepop);
end
% 选择交叉位置
pos=ceil(length*rand);
while pos==1
pos=ceil(length*rand);
end
% 个体交叉
chrom1=chrom(index(1)😅;
chrom2=chrom(index(2)😅;
k=chrom1(pos:length);
chrom1(pos:length)=chrom2(pos:length);
chrom2(pos:length)=k;
% 满足约束条件赋予新种群
flag1=test(city_coordinate,chrom(index(1)😅,jiange);
flag2=test(city_coordinate,chrom(index(2)😅,jiange);
if flag1*flag2==1
chrom(index(1),:)=chrom1;
chrom(index(2),:)=chrom2;
end
end
ret=chrom;
end
%% 变异操作
function ret=Mutation(pmutation,chrom,sizepop,length1,n,city_coordinate,jiange)
% pmutation input : 变异概率
% chrom input : 抗体群
% sizepop input : 种群规模
% iii input : 进化代数
% MAXGEN input : 最大进化代数
% length1 input : 抗体长度
% ret output : 变异得到的抗体群
% 每一轮for循环中,可能会进行一次变异操作,染色体是随机选择的,变异位置也是随机选择的
for i=1:sizepop
% 变异概率
pick=rand;
while pick==0
pick=rand;
end
index=unidrnd(sizepop);
% 判断是否变异
if pick>pmutation
continue;
end
pos=unidrnd(length1);
while pos==1
pos=unidrnd(length1);
end
nchrom=chrom(index,:);
nchrom(pos)=unidrnd(n);
while length(unique(nchrom))==(length1-1)
nchrom(pos)=unidrnd(n);
end
自我介绍一下,小编13年上海交大毕业,曾经在小公司待过,也去过华为、OPPO等大厂,18年进入阿里一直到现在。
深知大多数Python工程师,想要提升技能,往往是自己摸索成长或者是报班学习,但对于培训机构动则几千的学费,着实压力不小。自己不成体系的自学效果低效又漫长,而且极易碰到天花板技术停滞不前!
因此收集整理了一份《2024年Python开发全套学习资料》,初衷也很简单,就是希望能够帮助到想自学提升又不知道该从何学起的朋友,同时减轻大家的负担。

既有适合小白学习的零基础资料,也有适合3年以上经验的小伙伴深入学习提升的进阶课程,基本涵盖了95%以上前端开发知识点,真正体系化!
由于文件比较大,这里只是将部分目录大纲截图出来,每个节点里面都包含大厂面经、学习笔记、源码讲义、实战项目、讲解视频,并且后续会持续更新
如果你觉得这些内容对你有帮助,可以扫码获取!!!(备注Python)
深知大多数Python工程师,想要提升技能,往往是自己摸索成长或者是报班学习,但对于培训机构动则几千的学费,着实压力不小。自己不成体系的自学效果低效又漫长,而且极易碰到天花板技术停滞不前!
因此收集整理了一份《2024年Python开发全套学习资料》,初衷也很简单,就是希望能够帮助到想自学提升又不知道该从何学起的朋友,同时减轻大家的负担。
[外链图片转存中…(img-K3d0N68q-1713230233691)]
[外链图片转存中…(img-c2QKldRE-1713230233692)]
[外链图片转存中…(img-AEy4jgcc-1713230233692)]
[外链图片转存中…(img-H8CS89VP-1713230233693)]


既有适合小白学习的零基础资料,也有适合3年以上经验的小伙伴深入学习提升的进阶课程,基本涵盖了95%以上前端开发知识点,真正体系化!
由于文件比较大,这里只是将部分目录大纲截图出来,每个节点里面都包含大厂面经、学习笔记、源码讲义、实战项目、讲解视频,并且后续会持续更新
如果你觉得这些内容对你有帮助,可以扫码获取!!!(备注Python)









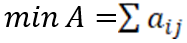
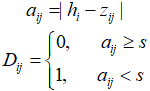














 392
392











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








