背景
分子对接定义:受体和配体之间通过能量匹配、空间匹配和化学性质匹配而相互识别形成分子复合物,并预测复合物结构的一种计算技术。也即,将配体分子放置到受体大分子的活性位点中,观察小分子与受体结合构象及预测作用能的过程。其目的是从小分子数据库中发现合适的化合物作为受体大分子的配体。
分子对接常用于研究药物和受体相互作用,一般过程包括确定受体的活性位点、定义活性口袋,根据受体活性位点与药物分子的性质和形状的互补性,调整受体活性位点柔性残基或药物的构象,计算对接时不同取向的药物与受体的相互作用能量来评估受体与配体的作用方式等。其他应用场景还包括:酶机理研究,先导化合物优化,先导分子鉴定/虚拟筛选等。
从对接对象角度可将分子对接分为五类:大分子受体蛋白质与小分子配体间的对接,蛋白质与蛋白质间的对接,蛋白质与DNA分子间的对接,蛋白质与RNA分子间的对接,以及蛋白质与多肽间的对接。
根据受体配体构象特征又可将分子对接分为三类:刚性对接,半柔性对接及柔性对接。刚性对接中受体和配体都看作是刚性的、不发生构象变化的。半柔性对接将受体看作是刚性的,而配体小分子在对接过程中通过平动、转动及可旋转二面角扭转等产生多种构象。柔性对接中则受体及配体均是柔性的。
01
现有软件性能评估
•过去的20年已涌现出了大量的分子对接软件,比如Auto Dock、GOLD、DOCK、Flex X、Glide等,其中既有商业软件也有学术软件,使用占比如右图所示。
•采样算法与打分函数是对接软件最核心的部分。前者决定了对接软件的采样性能;后者用于近似预测两个分子相互作用后的结合亲和力,其对复合物结合亲合力排序的预测精度为打分性能,用预测的结合亲和力(打分值)与实验值之间的皮尔逊相关系(rp)与史皮尔曼等级相关系数(rs)来评价。
•打分最佳结合构象通常不是实际最好的结合构象,之间差距巨大,这主要是因为打分函数存在缺陷。因此,有必要考察一致率以评估打分最佳与实际最佳的一致性。一致率用SRtsp/SRbp来定义,其中SRtsp与SRbp分别是打分最佳与实际最佳结合构象的成功率。
•打分性能的评估也表明,同一软件对不同蛋白家族的打分性能差异很大,因此不同蛋白的对接研究可能需要使用不同的软件。评价结果还发现,没有一个软件可以在采样性能与打分性能两样优于其它软件,因此最好的虚拟筛选解决方案是组合使用几种不同的对接软件。
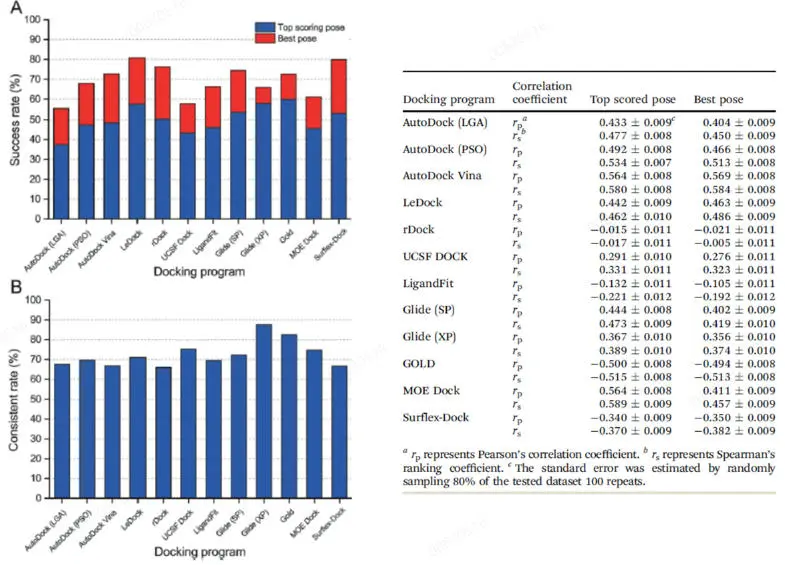
图1 十种对接软件用优化的构象做为初始构象对接计算的采样性能成功率(左)与一致率(右),打分性能测试结果
02
SOTA模型分析
1. KarmaDock:针对超大规模虚拟筛选的基于深度学习的分子对接方法(Nat. Comput. Sci. 2023)
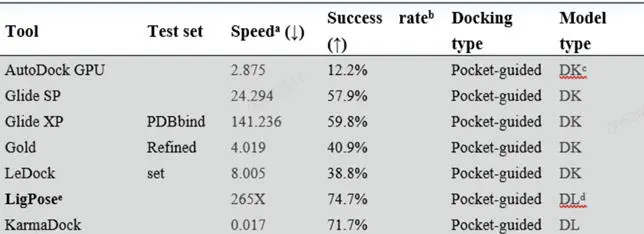
图2 KarmaDock与传统对接软件性能对比
目前,深度学习在保证对接速度和精度的平衡方面仍存在挑战。大多数模型将结合构象和结合强度的预测视为两个独立的任务,使得在预测蛋白配体结合时无法同时获取结合亲和力,这对于大规模虚拟筛选不够友好。KarmaDock利用深度学习技术,特别是几何深度学习,来预测蛋白质和配体间的结合姿势。该方法能够处理配体的灵活性,并考虑蛋白质口袋的复杂性以提高对接准确性。
KarmaDock的架构包括两个编码器(Graph Transformer (GT) 和 Geometric Vector Perceptrons (GVP))、一个混合密度网络(MDN)模块进行打分和一个EGNN模块进行对接。KarmaDock不仅要生成结合姿态,还要基于生成的结合姿态给出蛋白质和配体之间的结合强度打分,因而在对接能力和筛选能力方面均表现出强劲的性能。从精度上来看,KarmaDock的性能在三种数据集划分方法下均超过了传统的对接软件,至少提高了14.9%/22.3%的成功率;从速度的角度上看,KarmaDock在PDBbind测试集上较传统对接软件实现了至少163.06倍的加速。该方法还通过减少计算资源的需求,提高了对接过程的效率。
应用场景:药物设计、大规模虚拟筛选、生物分子研究等。
2. DiffBindFR:基于扩散模型的柔性分子对接方法(Chem. Sci. 2024)
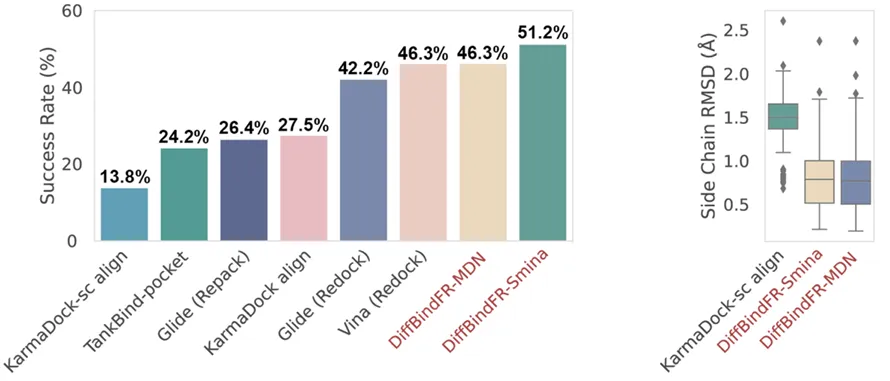
图3 DiffBindFR与传对接方法和深度学习方法性能对比
现有的柔性分子对接方法受限于计算复杂度,仅能考虑少量侧链构象变化。DiffBindFR 使用基于消息传递的 SE(3)-等变网络编码蛋白质口袋和配体分子之间全原子的复杂相互作用。整个模型将柔性对接定义为学习四个变量(配体旋转、平移、可旋转键扭转和口袋侧链扭转)在其切空间中的联合去噪过程的问题,可生成多个对接构象,利用confidence model挑选出排名第一的构象作为最终对接结果输出。
在PDBbind测试集上,DiffBindFR在分子对接成功率上超越了传统对接方法和深度学习方法,能够高精度预测蛋白配体结合口袋中残基的侧链构象,且该方法生成的口袋-配体复合物的全原子模型具有很好的物理合理性。此外,交叉对接是更能检验柔性对接方法应用潜力的任务,而该方法在交叉对接中也展现出了优异的性能(比如能够处理AlphaFold2预测的蛋白质结构中侧链构象阻挡配体分子进入正确结合位置的问题)。
应用场景:药物设计、虚拟筛选、结构基药物设计、交叉对接等。
3. RosettaVS:一种高精度基于结构的虚拟筛选方法(Nature Communications 2024)
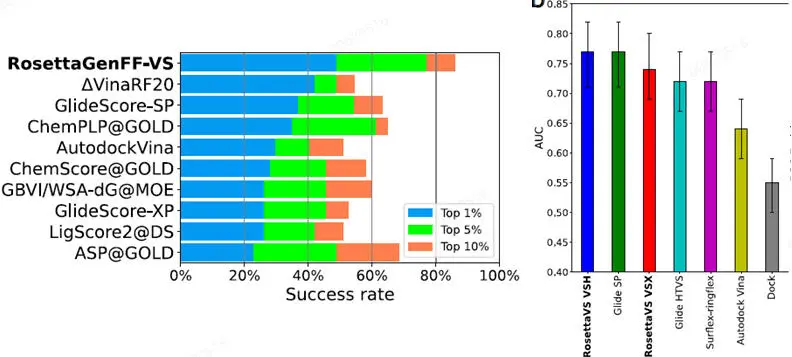
图4 RosettaVS与传统对接软件性能对比
为了能够针对超大化合物库进行筛选,研究人员采用了两种策略。首先,开发了一种改进的对接协议 RosettaVS,它实现了两种高速配体对接模式:虚拟筛选快速版 (VSX) 专为快速初步筛选而设计,虚拟筛选高精度版 (VSH) 是一种更准确的方法,用于对初始筛选中的最佳匹配进行最终排序。基于此,研究人员还开发了一个开源AI加速虚拟筛选平台 (OpenVS) ,该平台使用主动学习技术在对接计算过程中同时训练目标特定的神经网络,以高效地分类和选择最有希望的化合物进行昂贵的对接计算。
RosettaVS在多个基准测试集上表现出了优异的性能,包括CASF2016和DUD数据集。它在区分天然结合姿势和诱饵结构方面取得了领先的成绩,并且在预测配体结合亲和力方面也显示出了高准确性。利用OpenVS平台,研究人员成功从数十亿种化合物库中筛选出了具有个位数微摩尔结合亲和力的命中化合物,且筛选过程在不到七天内完成,显示了其高效的筛选能力。
应用场景:药物发现、先导化合物优化、蛋白质功能研究等。
03
启发和建议
实际应用中,在一个分子对接模型中采样性能与打分性能往往不能兼顾,最好的解决方案是几种不同模型组合使用,因此我们在模型迁移选择上应该重点关注模型的长板特征而非综合性能。
相对来讲,今年新出的DiffBindFR模型值得考虑迁移至MindSpore Science。不仅其性能达到了目前最优,其应用对象柔性分子本身就是目前分子对接算法中难度最大、挑战最大的一类,而且该方法在复杂的交叉对接场景下也可以有很好的表现,实属难得。
在药物发现应用领域,除了分子对接模型的开发以外,超大规模虚拟筛选的开源、可扩展平台的开发也至关重要。OpenVS开创了这一新方向,充分彰显了高通量优势和巨大应用潜力,有望推动学界业界更广泛地应用和优化这一技术,从而为海量先导化合物库的高效筛选提供理想解决方案。
参考文献
[1] Physical Chemistry Chemical Physics, 2016, 18(18): 12964-12975.
[2] Nature Computational Science, 2023, 3(9): 789-804.
[3] Chemical Science, 2024, 15(21): 7926-7942.
[4] Nature Communications, 2024, 15(1): 7761.
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








