BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。BLAST程序能迅速将核苷酸或蛋白质序列与公开数据库进行相似性序列比较,并计算匹配的统计显著性。进而可用于推断序列之间的功能和进化关系,并帮助识别基因家族成员。如果您想进一步了解BLAST算法,您可以参考NCBI的BLAST Course,该页有BLAST算法的介绍。找到序列之间的局部相似区域。该程序与序列数据库进行比较,详情请看官网链接BLAST: Basic Local Alignment Search Tool
-
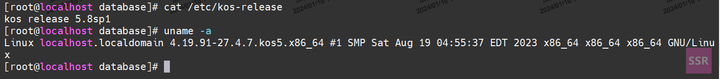
安装准备 2.1 操作系统环境 版本信息:KeyarchOS 5.8 硬件平台:X86_64
添加图片注释,不超过 140 字(可选)
2.2 BLAST版本 ncbi-blast-2.9.0+-x86-linux
-
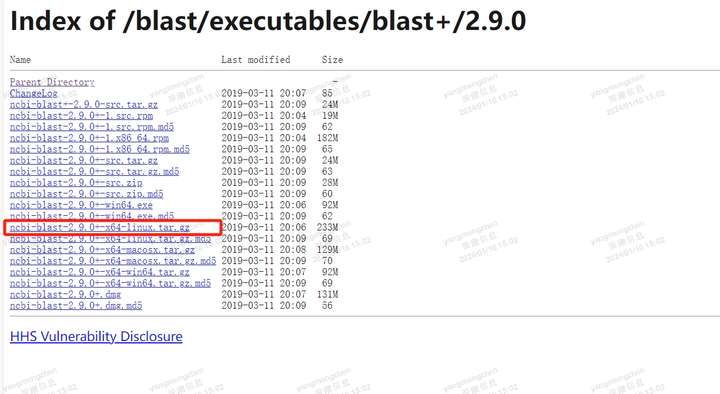
下载
添加图片注释,不超过 140 字(可选)

添加图片注释,不超过 140 字(可选)
选择合适的版本下载,这里我选择的是2.9.0+的x86架构,linux环境的压缩包
-
安装
-
安装并配置环境
-
在linux上创建文件夹,例如/home下创建soft文件夹,并将下载的压缩包放到文件夹下解压,命令如:tar -zxvf ncbi-blast-2.9.0+-x64-linux.tar.gz

添加图片注释,不超过 140 字(可选)
-
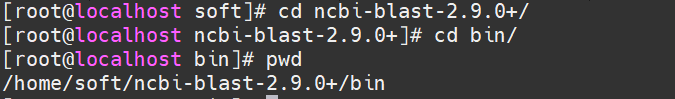
进入解压的文件夹下的bin目录,输入pwd,查看当前路径,复制路径
添加图片注释,不超过 140 字(可选)
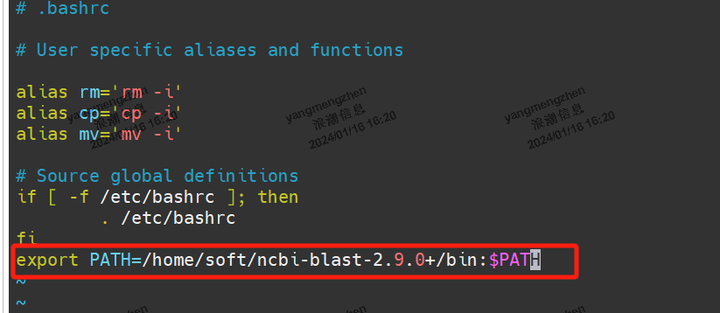
添加环境变量,使用 vim ~/.bashrc 命令,编辑文件,在最后一行添加:export PATH=刚刚复制的路径:$PATH ,然后点击esc,输入:wq!保存文件并退出。
添加图片注释,不超过 140 字(可选)
使用source ~/.bashrc 命令让其生效
-
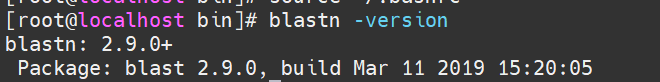
验证是否安装成功 输入blastn -version 命令,出现版本号则安装成功,若出错可能由于环境配置错误或blast依赖包未安装,可根据具体错误信息使用yum install 进行安装。
添加图片注释,不超过 140 字(可选)
-
使用 在使用之前需要准备一个fasta库,也就是所有的序列信息,还有要比对的源数据,也是一个fasta文件,这里以人基因组序列文件与人TP53基因序列将进行序列对比实例演示。
-
下载并解压人基因组序列文件 以GRCh38为例,下载路径为https://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/release_44/gencode.v44.transcripts.fa.gz
-
下载人TP53基因序列 >TP53 CTCAAAAGTCTAGAGCCACCGTCCAGGGAGCAGGTAGCTGCTGGGCTCCGGGGACACTTTGCGTTCGGGC TGGGAGCGTGCTTTCCACGACGGTGACACGCTTCCCTGGATTGGGTAAGCTCCTGACTGAACTTGATGAG TCCTCTCTGAGTCACGGGCTCTCGGCTCCGTGTATTTTCAGCTCGGGAAAATCGCTGGGGCTGGGGGTGG GGCAGTGGGGACTTAGCGAGTTTGGGGGTGAGTGGGATGGAAGCTTGGCTAGAGGGATCATCATAGGAGT TGCATTGTTGGGAGACCTGGGTGTAGATGATGGGGATGTTAGGACCATCCGAACTCAAAGTTGAACGCCT AGGCAGAGGAGTGGAGCTTTGGGGAACCTTGAGCCGGCCTAAAGCGTACTTCTTTGCACATCCACCCGGT GCTGGGCGTAGGGAATCCCTGAAATAAAAGATGCACAAAGCATTGAGGTCTGAGACTTTTGGATCTCGAA 以上是人TP53基因的部分序列,复制以上文本并保存到TP53.fa文件中。
-
创建数据库
-
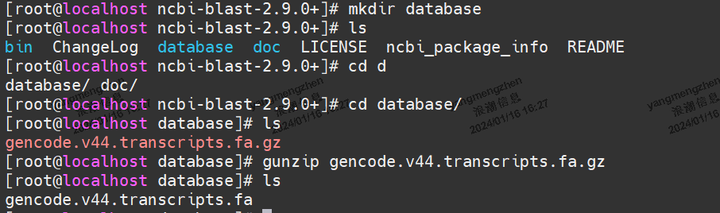
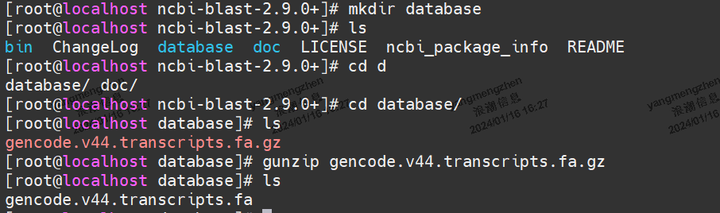
linux 解压的文件夹下创建database文件夹,将下载的gencode.v44.transcripts.fa.gz文件放在database文件夹下,并使用gunzip
添加图片注释,不超过 140 字(可选)
gencode.v44.transcripts.fa.gz解压压缩包
添加图片注释,不超过 140 字(可选)
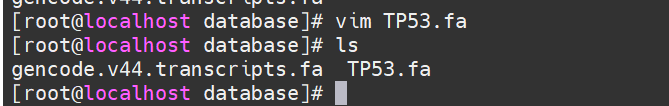
将TP53.fa也放在database文件夹下
添加图片注释,不超过 140 字(可选)
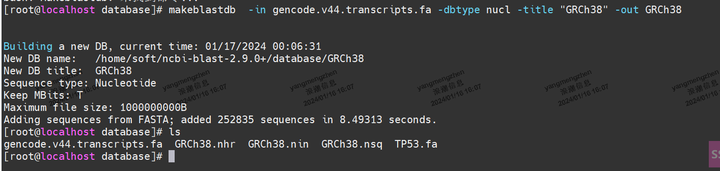
进入database目录下,执行建库命令:
makeblastdb -in gencode.v44.transcripts.fa -dbtype nucl -title "GRCh38" -out GRCh38
主要参数说明:
-in:待格式化的序列文件(即gencode.v44.transcripts.fa文件)
-dbtype:数据库类型,prot(蛋白质序列)或nucl(核酸序列)
-title:为数据库指定一个标题。
-out:数据库名
更多参数说明请执行:makeblastdb -help
添加图片注释,不超过 140 字(可选)
序列对比
-
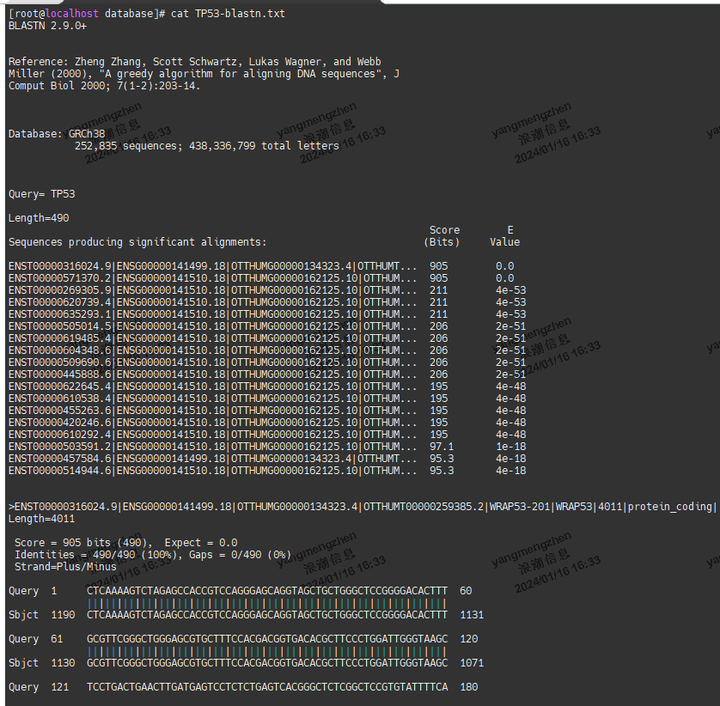
进入database目录下,执行命令:blastn -query TP53.fa -db "GRCh38" -out TP53-blastn.txt
主要参数说明:
blastn:使用的比对程序(此处为核酸序列和核酸序列的比对)
-query:表示自己想要比对的FASTA格式的核酸序列
-db:是所建立的比对数据库
-out:输出文件路径及文件名(此处为TP53-blastn.txt)
![]()
添加图片注释,不超过 140 字(可选)
添加图片注释,不超过 140 字(可选)
查看结果 cat TP53-blastn.txt





















 595
595

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








