线粒体氧化磷酸化(OXPHOS)为大脑活动提供能量,且线粒体缺陷与神经退行性及神经精神疾病相关。为了理解大脑活动和行为的基础,需要界定大脑的分子能量景观(molecular energetic landscape)。在此,为了弥合认知神经科学与细胞生物学之间的尺度差距,我们开发了一种物理体素化(physical voxelization)方法,将一个冷冻的人脑冠状半球切片分割成703个与神经成像分辨率相当(3×3×3毫米)的体素。在每个皮层和皮层下脑体素中,我们分析了线粒体表型,包括OXPHOS酶活性、线粒体DNA和体积密度,以及线粒体特异性呼吸能力。我们发现人脑包含多样的线粒体表型,这些表型受拓扑结构(topology)和细胞类型的共同驱动。与白质相比,灰质包含的线粒体多出50%以上。此外,灰质中的线粒体在生物化学上被优化以进行能量转换,尤其是在近期进化出的皮层脑区中。将这些数据扩展至全脑,我们创建了一个反向线性回归模型,该模型整合了多种神经成像模态,以生成线粒体分布与特化(specialization)的全脑图谱。该模型预测了来自同一捐赠者大脑的一个独立脑区中的线粒体特征。这种方法以及由此产生的线粒体表型MitoBrainMap(线粒体脑图谱)为探索支撑正常大脑功能的分子能量景观提供了一个基础。这项资源也与神经成像数据相关联,并为与神经精神和神经退行性疾病相关的区域化大脑过程界定了亚细胞基础。本文发表在Nature杂志。
正文
功能性神经成像技术能够捕捉动态的电、代谢和血流动力学大脑能量状态,但仅提供对潜在亚细胞生物能量过程的间接测量。所有基础和活动依赖性的大脑过程都依赖于细胞能量转换(或称生物能量学),这涉及数万亿个进行呼吸作用的线粒体通过氧化磷酸化(OXPHOS)合成ATP(三磷酸腺苷)。每个神经元和神经胶质细胞中含有数百到数千个这样的细胞器。线粒体在分子层面上特化以满足特定的细胞需求,指导亚细胞活动,并提供支持大脑活动所必需的能量。除了能量学之外,线粒体还参与其他关键功能,包括细胞间信号传导、神经元兴奋性的调节、神经递质释放以及炎症过程的调控。因此,线粒体在分布于大脑大尺度网络中的众多支持性功能中也扮演着至关重要的角色。近期的机制性研究已经强调了线粒体对认知和行为的影响。然而,线粒体通常在细胞生物学的亚微米尺度上进行研究,这与认知和系统神经科学之间存在一个主要的方法学和概念上的尺度差距。当使用常规场强的磁共振成像(MRI)对全脑进行成像时,该领域(指认知和系统神经科学)在毫米尺度上运作。因此,阻碍我们解析驱动和指导复杂人脑动态的能量力量的一个主要障碍是,能量转换线粒体在不同大脑结构中的空间分布。
神经成像技术对于大尺度绘制大脑新陈代谢图谱是无价的。正电子发射断层扫描(PET)提供了代谢过程的定量测量,包括使用[18F]-FDG测量葡萄糖代谢,但需要侵入性的放射性示踪剂且空间分辨率有限。血氧水平依赖(BOLD)功能性MRI(fMRI)能够以毫米和秒级的分辨率捕捉血红蛋白氧合的变化,但它仅间接反映了代谢(有氧和无氧)反应对脑组织活动的混合贡献。化学交换饱和转移(CEST)MRI通过检测可交换的生物分子质子来绘制代谢物图谱,具有相对较高的空间分辨率,但在量化绝对代谢物浓度方面面临限制。功能性磁共振波谱(fMRS)量化神经递质和代谢物浓度(如谷氨酸和乳酸),为体内大脑代谢提供见解。然而,低信噪比限制了其时间和空间分辨率,需要在采集过程中使用较大的体素尺寸。这些技术以及其他技术的整合(例如,测量脑氧代谢率或脑血容量的技术)提供了关于大脑代谢与功能关系的广阔视角。然而,它们的解释需要开发高分辨率、全脑范围的分子能量景观(molecular energetic landscape)图谱,这对于理解区域代谢特性和能量转换能力至关重要。这样的图谱将能够将线粒体生物能量学与宏观神经成像数据整合起来,从而通过将其与线粒体生物学联系起来,潜在地增强PET、BOLD fMRI、CEST MRI和fMRS代谢评估的特异性。
初步工作已开始将中尺度(mesoscale)神经成像与微观解剖学联系起来,这为人类神经成像发现的细胞成像解释提供了突破。这些进展旨在加深我们对大脑发育、认知、情绪以及各种神经病理学背后机制的理解。为了连接认知神经科学和细胞生物学,我们在此开发了一种物理体素化(physically voxelize)冷冻人脑组织的方法,以系统地分析跨越一个冠状脑切面的线粒体分子和生化多样性。我们还提供了一种基于常用MRI数据预测线粒体分布、密度和OXPHOS能力的算法。
大脑体素化
为了系统地绘制人类大脑线粒体的分布、多样性和分子特化,第一个挑战是以与MRI相当的空间分辨率物理分割(partition)冷冻脑组织。这将使得能够将分子和生化特征映射到标准的神经成像立体定向空间(stereotaxic space)(图1a)。
为了应对这一挑战,我们选择了一个在短死后间隔(PMI = 8小时,在-80°C下储存10年)内获得的神经典型大脑。捐赠者为一名54岁男性,其毒理学筛查显示精神活性药物、毒品和酒精均为阴性,神经病理学检查阴性,并且无神经精神疾病史(方法)。将包含皮层和皮层下解剖结构的整个右半球的冠状板(slab),在保持-25°C冷冻状态下进行体素化,以在处理过程中保持酶活性和分子完整性。为了以与MRI相当的3毫米各向同性分辨率物理分割冷冻脑板,我们编程了一台计算机数控(CNC)切割机,以3毫米深度雕刻了一个3×3毫米的方形网格,随后手动收集并索引了总共703个样本(扩展数据图1和2以及补充视频1)。
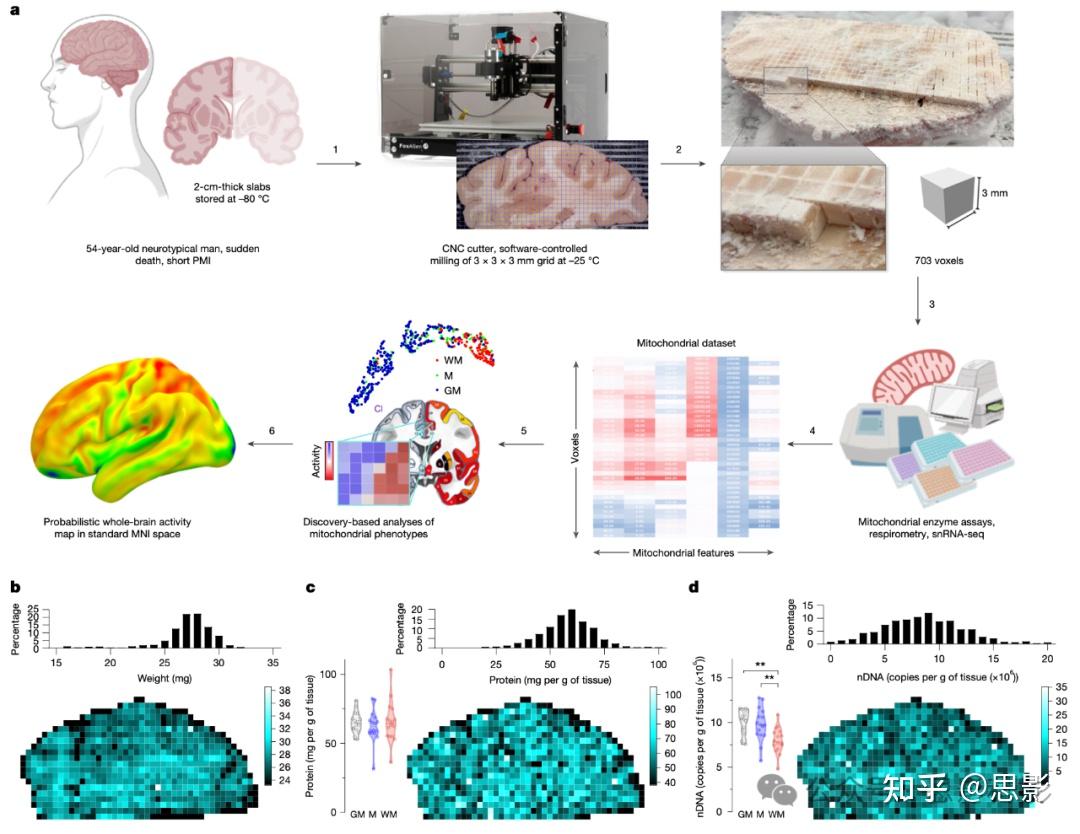
图 1:人脑体素化与绘图
a, 死后脑组织被切成约2厘米厚的冠状板(slab),在制冷剂R-134a中快速冷冻,并在-80°C下储存10年(方法)。
1.将位于前连合中心后方15.51毫米的立体定向MNI坐标处的一块脑板安装在-25°C冷冻室中操作的CNC切割机上(步骤1)。
2.顶部表面被清洁和整平,并使用0.4毫米钻头以3×3毫米的网格铣削至3毫米深度(步骤2)。
3.手动收集脑体素,再次清洁表面,并收集几个50纳米的冷冻切片用于组织学评估。对>700个样本中的每一个进行称重、匀浆,并进行一系列生化测试,以生成与脑切片上体素坐标相关联的线粒体特征矩阵(步骤3)。
4.根据线粒体复合物的相对活性和丰度对每个体素进行表征(步骤4)。
5.进行降维和聚类分析以识别不同的线粒体类型(mitotypes)(步骤5)。将线粒体特征图谱配准到标准的MNI空间,并进行回归分析,以将这些图谱与相同脑区的平均MRI读数相关联。
6.最后,使用MRI数据预测线粒体特征,并将线粒体图谱扩展到全脑(步骤6)。
b–d, 收集到的体素的基本属性,包括其重量(b)、蛋白质含量(c)和核DNA(nDNA)含量(d)。顶部显示了每个参数值的直方图,底部显示了脑切片上值的映射。脑切片左侧的条形图显示了来自同一大脑枕叶的对照灰质(GM)、白质(WM)和混合(M)灰质与白质样本中相应值的重复测量结果,这些样本在检测板中用作归一化对照(扩展数据图2d)。请注意,每个面板中的色标使用与其对应直方图的x轴标签相同的标签。**P < 0.01,通过单因素方差分析(ANOVA)及Tukey事后检验。
大脑线粒体表型分析
为了在数百个人脑物理体素的尺度上进行线粒体表型分析,首先将每个样本随机分配到96孔板中的一个孔(随后使用自定义算法进行反卷积;扩展数据图2和方法),称重,匀浆,并进行质量控制以及总蛋白浓度和核DNA含量的基本表征。这些参数在整个脑切片上显示出相对均匀的质量(图1b-d)。平均体素重量为26.2 ± 4.5毫克(平均值 ± 标准差;根据3×3×3毫米体素尺寸和水的体积密度预测重量 = 27毫克)。如此小的组织样本需要高灵敏度的检测方法来测量线粒体密度和呼吸链(也称为电子传递链,ETC)的酶活性。线粒体密度通过两个线粒体标记物的组合进行评估:柠檬酸合酶(CS)活性和线粒体DNA(mtDNA)密度。反映能量转换能力的OXPHOS酶通过三个标记物进行索引:复合物I(CI;也称为NADH-泛醌氧化还原酶);复合物II(CII;也称为琥珀酸脱氢酶,SDH);和复合物IV(CIV;也称为细胞色素c氧化酶,COX)。这些酶运输电子并产生维持生命的跨膜电位,最终为OXPHOS系统合成ATP(三磷酸腺苷)提供动力。
为了确保数据的稳健性,OXPHOS酶活性在不同实验室中使用两种独立的检测方法进行了量化:一种针对脑组织优化的微型比色法和冷冻组织呼吸测定法(frozen-tissue respirometry)(扩展数据图2)。为了提高整个数据集的技术准确性,来自同一大脑枕叶的标准参考样本(灰质(GM)、白质(WM)以及混合GM和WM组成的体素)覆盖了可能活性的整个范围,在每个批次(八个96孔检测板)中进行重复检测,并用于校正潜在的批次效应。这一过程产生了一个均匀的、MRI分辨率的人脑整个冠状切面上线粒体活性谱的数据集(扩展数据图3a-f)。我们排除了组织量过少(位于脑切片边缘的样本)或酶活性低于检测限(主要来自WM)的体素,这导致大约10%的体素至少缺少一项测量值。所有线粒体特征(CI、CII、CIV、CS和mtDNA)都在633个体素中测定,这需要通过比色法、呼吸测定法、生化和定量PCR(qPCR)检测分析27,820个独立样本,包括重复样本、对照和标准品(方法)。
线粒体密度和OXPHOS能力
线粒体具有分子和功能上的特异性,并展现出广泛的能量转换能力。为了评估线粒体的“质量”和功能特化,我们先前开发了一个简单的线性公式,其中OXPHOS活性除以线粒体质量,得到单个线粒体的OXPHOS能力指数,我们称之为线粒体健康指数(MHI)。在这里,利用手头一个规模大得多的脑线粒体生物化学数据集,我们发现单个线粒体指标的分布呈左偏态(left-skewed)(扩展数据图4a)。
当球状细胞器的半径呈正态分布时,与球状体表面积相关的指标遵循平方根正态分布,而与球状体体积相关的指标则通过立方根进行归一化。这种依赖性的一个公认例子见于致密核心分泌囊泡,其中同样呈左偏态的神经递质含量分布通过立方根变换得到归一化。尽管线粒体的形态比球体更复杂,但对线粒体含量(CS1/3 和 mtDNA1/3)和OXPHOS能力(CI1/2, CII1/2 和 CIV1/2)参数进行体积变换(volumetric transformation)充分地使线粒体特征归一化(即,降低了变异系数、偏度和柯尔莫哥洛夫-斯米尔诺夫距离)。这种变换还使我们能够对GM和WM体素的线粒体特征的正态分布进行反卷积,从而产生一个单个线粒体特征的最终参数化数据集(扩展数据图4b-d)。
正如预期的那样,变换后的线粒体含量标记物之间存在强相关性(Pearson's r2 = 0.46, P < 0.0001;扩展数据图5a),这为整合CS1/3和mtDNA1/3作为MitoD提供了基础,MitoD代表了每个脑体素的线粒体组织密度(图2a,c)。mtDNA拷贝数(mtDNAcn)反映了每个细胞核的mtDNA基因组数量,也在此呈现(扩展数据图3h),但它受到细胞密度的影响;因此,脑区之间的比较不能直接进行。通过比色法和呼吸测定法测量的OXPHOS酶活性也高度相关(扩展数据图5b-d),随后将它们平均以整合为一个稳健的组织呼吸能力(TRC;图2a,b)测量值。TRC代表了每毫克脑组织线粒体OXPHOS能力的直接量化。
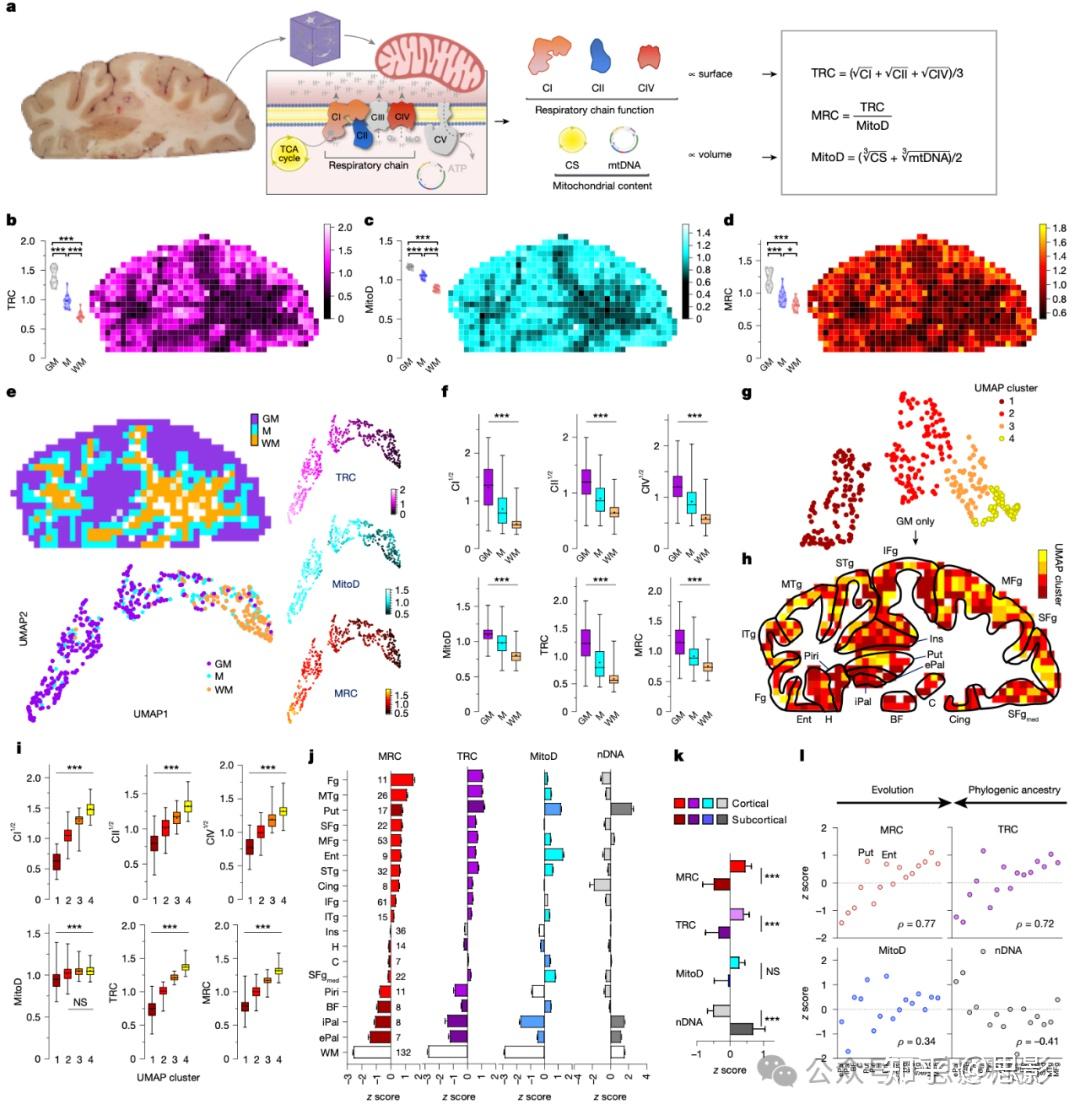
图 2:线粒体密度与呼吸链复合物活性
a, 酶活性检测概览。左图,体素化前的脑板以及线粒体内膜呼吸链复合物示意图。中图,细胞器几何形状、呼吸链复合物活性、CS和mtDNA密度之间的关系。右图,TRC、MRC和MitoD的公式。
b–d, TRC(组织呼吸能力)图谱(b;源自扩展数据图3i,j)、MitoD(线粒体组织密度)图谱(c;扩展数据图3g中两个面板的平均值)和MRC(线粒体呼吸能力)图谱(d)。每个面板左侧的条形图显示了不同检测板上对照GM、WM和混合GM与WM样本重复测量的分布。
e, 基于MNI空间位置,将具有全部6种线粒体特征的633个体素分为GM(n = 325)、WM(n = 132)和混合(n = 176)GM与WM簇。UMAP图显示了簇与每个体素的物理位置(左)或每个体素中TRC、MitoD和MRC的z-score值(右)之间的相关性。b-e中的色标显示了每个面板中显示值的z-score。
f, GM、WW和混合GM与WM体素中线粒体特征的条形图和须线图。
g, GM体素线粒体特征的UMAP图和簇。
h, 将UMAP簇映射到特定脑区(缩写见方法)。
i, 来自g的GM簇中的线粒体特征。对于MitoD,只有簇1与其他簇不同。
j, 映射到特定脑区的体素组(组大小显示在条形旁边)中的线粒体特征。皮层下区域颜色较深。
k, 皮层和皮层下区域的线粒体特征。
l, 测量值与源自比较解剖学研究(方法)的已知脑区系统发育组织的比较。
ρ是斯皮尔曼等级相关系数(Spearman's rho)。*P < 0.05 和 ***P < 0.001,通过ANOVA及Tukey事后检验(f,i)或t检验(k)。NS,不显著。条形图和须线图显示第25至第75百分位数(盒子)、最小值和最大值(须线)、中位数(盒子内的水平线)和平均值(+)。
参考GM体素的所有线粒体指标值均显著高于参考WM体素,而混合GM和WM组成的体素则具有中间值(图2b,c和扩展数据图3,每个面板左侧的条形图)。这一结果与先前基于神经成像的大脑能量代谢估算结果一致。参考GM和WM体素的数据分布不重叠,这突显了它们的差异。因此,检测GM和WM区域之间差异的信噪比很高,这体现在所有线粒体指标的巨大效应量上(GM vs WM的Hedges' g = 3.5-10.7)。与在整个切片上均匀分布的蛋白质和细胞密度测量值(图1b-d)相比,线粒体特征图谱(图2b,c和扩展数据图3g-j)显示出与大脑解剖结构(图2a)重叠的明显异质性。
为了创建一个反映线粒体针对OXPHOS和能量转换的组织特异性特化的MHI体积模拟量,我们计算了一个额外的指标,标记为线粒体呼吸能力(MRC),方法是将每个体
素的TRC(组织呼吸能力)相对于MitoD(线粒体组织密度)进行表达(MRC = TRC / MitoD)(图2a)。如果整个脑切片中每个线粒体都具有相同的OXPHOS能量转换能力,那么MRC在所有脑区的值应该相似。相反,MRC(线粒体呼吸能力)的差异将表明存在具有不同OXPHOS能力或不同OXPHOS特化程度的线粒体。我们的数据确定了MRC在GM和WM参考样本之间以及冠状切面上的解剖结构之间都存在变异(图2d)。因此,除了GM和WM之间预期的线粒体密度差异外,这些结果揭示了最大呼吸能力(以单个线粒体为基础)在人脑中变化的程度。
脑区特异性线粒体
接下来,我们将每个体素的坐标配准到标准的蒙特利尔神经学研究所(MNI)空间,这是MRI研究中最广泛使用的参考神经解剖空间。由解剖学专家(M.T.d.S.)手动将每个体素注释为GM(n = 325)、WM(n = 132)或混合型(n = 176),并标记其立体定向位置(扩展数据图6)。通过对相邻的薄组织切片进行尼氏物质(Nissl substance,标记神经元和胶质细胞)和神经元核标记物NeuN(仅标记神经元)的染色,进一步确认了体素的解剖学身份(扩展数据图1h,i)。
将所有带有完整线粒体参数集的注释体素投影到均匀流形近似与投影(UMAP)空间中,产生了视觉上识别出的簇,这些簇清晰地映射到大体的体素组成(GM和WM;图2e)。相反,当使用UMAP簇来预测体素的来源时,它们分别以82%和64%的准确率映射到GM和WM体素位置(随机水平 = 51%(GM)和21%(WM);扩展数据图7a)。其余体素被分配到包含WM和GM组织的混合类别,这使得误分类率在1-5%之间。此外,GM和WM体素在所有线粒体特征上表现出巨大差异(g = 1.8-2.6;图2f)。与GM相比,WM包含显著更少的线粒体质量和总OXPHOS能力(MitoD和TRC),但WM体素的TRC相对于MitoD表现出不成比例的低值(扩展数据图7b)。因此,反映单个线粒体能量转换特化的MRC在WM体素中最低(图2f)。
一项更详细的、侧重于GM体素的分析产生了四个具有不同线粒体活性谱的UMAP(均匀流形近似与投影)簇(图2g-i和扩展数据图7c)。值得注意的是,尽管皮层和皮层下区域具有相似的MitoD值(图2j,k),但皮层GM区域表现出比皮层下区域更高的TRC(组织呼吸能力)(P < 0.01, t检验;g = 1.1)和MRC(P < 0.01;g = 1.4)值。一个例外是壳核(putamen),这是一个皮层下结构,具有最高的TRC和MitoD值。这一结果可能归因于该区域终末突触、神经突和轴突投射的高密度,包括来自持续活跃的黑质致密部多巴胺能神经元的大型轴突分枝。此外,壳核具有最高的核DNA含量,因此含有众多细胞体。壳核也经历了进化上的功能再生(evolutionary exaptation),这可能解释了这种差异。相反,我们在内侧和外侧苍白球以及几个WM区域观察到高细胞密度和低MRC。
在比较解剖学中,物种间结构的存在与否使得能够对人类主要大脑结构进行系统发育分类。脑干、苍白球和纹状体区域被普遍认为是进化上最古老的,而皮层区域则代表了最近的进化发展。我们的结果显示,TRC和MRC都与脑区的估计系统发育年龄密切相关。在进化时间线上较晚出现的脑区——从爬行动物到哺乳动物,再到灵长类动物和人类——其线粒体表现出更高的MRC值(ρ = 0.72-0.75, P > 0.01;图2l)。这种酶的特化与存在一个伴随代谢需求增加的进化梯度的假说相符。我们的数据表明,这种需求是通过具有高单位线粒体OXPHOS能力的特化线粒体表型(mitotype)来满足的。
细胞类型特异性线粒体
特化的线粒体类型(mitotypes)要么映射到特定的脑区,要么映射到特定的细胞类型,而这些细胞类型在不同脑区的丰度有所不同(WM vs GM;皮层 vs 皮层下在细胞结构上存在差异)。为了分析人脑中细胞类型特异性的分子线粒体类型,我们对从用于上述线粒体酶学测量的体素匀浆中回收的单个细胞核进行了单核RNA测序(snRNA-seq)。线粒体酶检测所需的偏重裂解的化学方法(lysis-oriented chemistry)和剧烈的机械破碎与通常为生成高质量snRNA-seq样本所需的更温和方法形成对比。因此,我们方法的这一局限性应在未来旨在确定细胞类型特异性线粒体类型的研究中加以修正。
我们从四个区域选择了单个体素:皮层(即中颞回,MTg)、海马体、壳核和胼胝体(即WM),从而覆盖了一系列细胞类型组成差异很大的脑区。对50微米厚的脑切片进行神经元和胶质细胞(尼氏,Nissl)或仅神经元(NeuN)的组织化学染色,证实了每个体素预期的细胞身份(图3a和扩展数据图8)。在应用严格的数据清理和质量控制阈值后,我们通过计算总共回收了32,515个细胞核(MTg为8,945个,海马体为7,044个,壳核为6,176个,胼胝体为10,350个),这些细胞核可被归类为9种主要的细胞类型(图3b)。这一结果证实了每个立体定向定义的体素之间预期的细胞组成差异(图3c)。
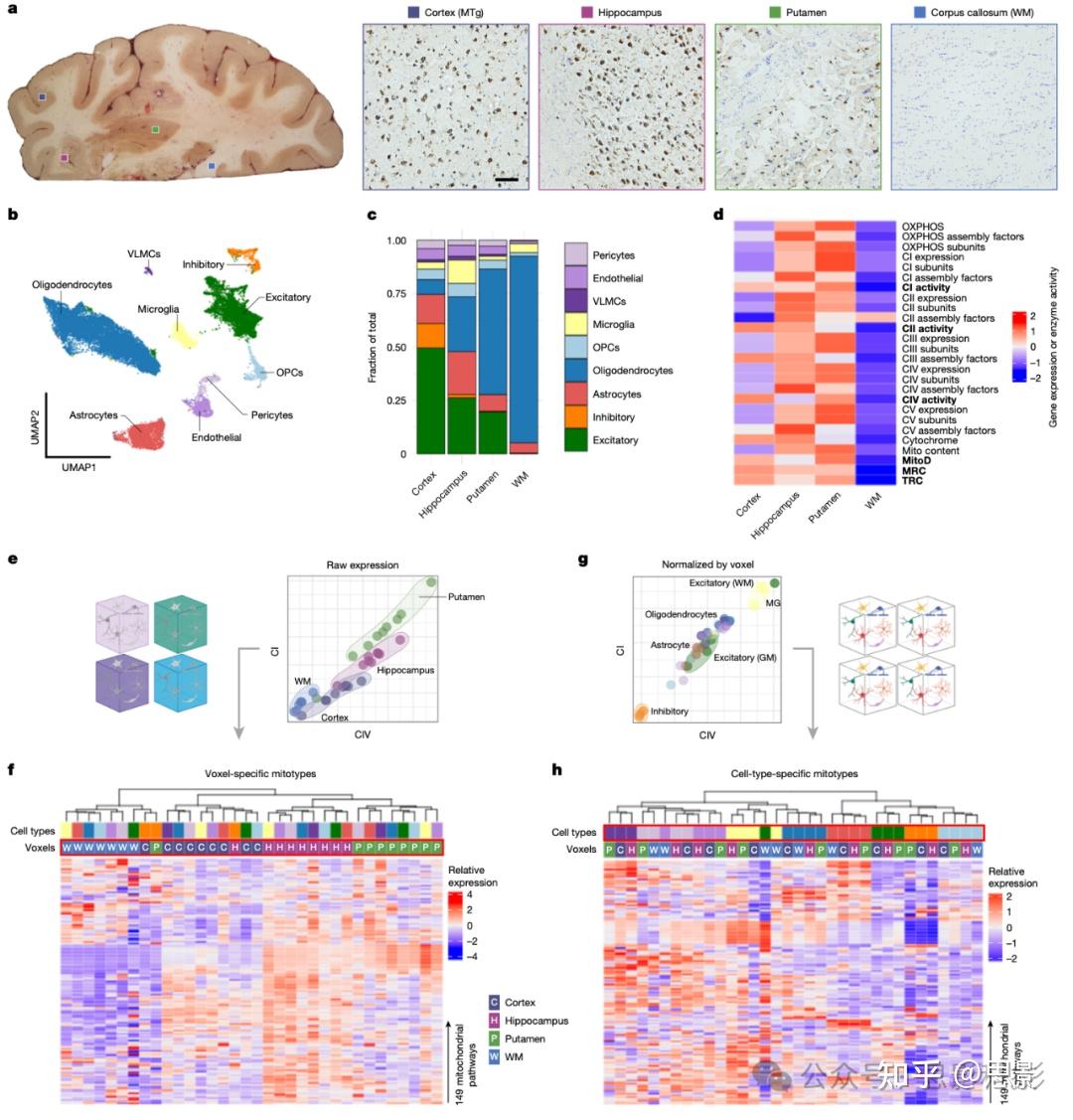
图 3:选定体素的snRNA-seq分析
a, 左图,为snRNA-seq分析选择的体素位置及其组织学评估。右图,薄脑切片用尼氏(蓝色,所有细胞核)染色,并用NeuN(棕色,神经元细胞)进行免疫标记。比例尺,100微米。 b, 对四个体素进行snRNA-seq分析识别出的细胞簇的UMAP图,显示了九种主要细胞类型。OPCs,少突胶质前体细胞;VLMCs,血管软脑膜细胞。
c, 四个体素中每种细胞类型的比例。
d, 每个体素(每个样本的伪批量表达)OXPHOS通路基因表达评分的热图,以及活性测量值(以粗体突出显示)。
e, CI和CIV原始表达水平之间的相关性。每个数据点代表每个体素和细胞类型中OXPHOS亚基的伪批量表达,并按体素类型着色。
f, 149个线粒体通路(补充数据1)表达评分的热图。每列代表一个体素和细胞类型中的原始表达评分,在前两行中用颜色编码(体素颜色同a,细胞类型颜色同c)。请注意样本按其体素类型聚类。
g, 在对每个体素类型中的表达水平进行z-score归一化以考虑区域特异性差异后,CI和CIV表达之间的相关性。每个数据点按其细胞类型着色。
h, 在对每个样本的表达进行归一化后,体素和细胞类型中线粒体通路表达评分的热图。样本按其细胞类型聚类现在很明显。
线粒体的特化源于约1,136个线粒体基因或蛋白质的受调控表达,这已通过MitoCarta3.0清单得到验证。使用伪批量转录组(pseudo-bulk transcriptomes,即模拟批量RNA测序),我们回收了1,118个核编码和mtDNA编码的线粒体基因,并计算了每个体素和OXPHOS亚基的OXPHOS评分。与我们的分子和生化结果一样,WM体素表现出比GM体素更低的mtDNA编码基因表达(图3d;log倍数变化 < -1.5,调整后P < 0.001)。总的来说,尽管仅来自四个体素的数据不足以得出明确结论,但在壳核、海马体和WM中,OXPHOS复合物亚基的相对基因表达与测得的CI、CII和CIV酶活性成比例(图3d)。这一结果为观察到的酶学数据提供了部分趋同证据(convergent evidence);然而,需要在更大的数据集中进行额外验证。一个例外是皮层中CI和CII亚基的低表达,这并未反映在酶活性测量中。尽管回收的神经元丰度较低,壳核在OXPHOS表达和酶活性方面均得分最高。这一结果表明,要么回收的细胞核未能忠实代表脑区中存在的细胞类型,要么存在独立于细胞类型组成的、区域特异性的酶促线粒体类型驱动因素,例如局部活动或功能连接模式。
为了研究不同细胞类型如何构成不同脑区的线粒体类型,我们比较了四个体素中匹配细胞类型的两种OXPHOS复合物(CI和CIV)的mRNA丰度。OXPHOS复合物的原始转录谱按体素而不是细胞类型聚类(图3e和补充数据1)。这一发现表明,驱动每种细胞类型OXPHOS转录组差异的主要因素是其所在的脑区。然后,我们将此分析扩展到149个线粒体通路(MitoCarta3.0),这些通路包含复合多基因评分,旨在降低单个基因的影响,以创建功能相关线粒体类型特化的更稳定估计值。我们再次发现,来自同一脑区的不同细胞类型之间的转录谱比来自不同区域的相似细胞类型之间的转录谱更为相似(图3f)。因此,大脑线粒体类型的大部分变异可归因于区域差异。
值得注意的是,通过对每个体素内的mRNA水平进行z标准化(z scoring)(而不是对整个数据集进行标准化)来消除体素间总体表达水平的变异后,揭示了细胞类型特异性的线粒体类型(图3g)。例如,相对于其他细胞类型,抑制性神经元的OXPHOS复合物表达最低,而内皮细胞的表达最高。当对每个样本的所有线粒体基因表达谱进行归一化时,观察到了类似的按细胞类型而非体素位置进行的稳健聚类(图3h)。
总的来说,我们的单核转录组分析表明,尽管人脑细胞类型之间的线粒体类型差异存在并且在所检查的脑区中是保守的,但影响跨脑区所有细胞类型的线粒体基因表达的全局驱动因素驱动了最显著的线粒体变异。这一结果与对大脑功能组织的拓扑学理解一致。
线粒体与神经成像模态
接下来,我们探究了线粒体类型(mitotypes)是否可能反映在标准的神经成像模态中,包括来自数百个个体(n = 1,870例来自18-35岁健康成年人的多模态脑部MRI扫描)的T1和T2 MRI、BOLD fMRI以及扩散加权图像(扩展数据表1)。这样的相关性将使我们能够利用神经成像数据来预测线粒体特征。我们的目标是将线粒体OXPHOS能力的图谱扩展到整个大脑,并最终利用神经成像数据推导出个性化的、基于MRI的线粒体类型图谱。
如上所述,来自我们冠状切片的每个体素都被配准到标准MNI脑图谱中其对应的、分辨率匹配的体素。每个体素的准确映射都经过了手动验证。GM和WM概率总和低于70%的样本被丢弃,以避免部分容积效应污染。剩余的539个体素,其所有线粒体特征均已测量并正确映射到立体定向空间,被随机分为两组,用于训练模型(80%,即431个体素)和测试模型(20%,107个体素)(扩展数据图9a和补充数据2)。使用逐步线性回归模型,将每个线粒体特征(CI、CII、CIV、MitoD、TRC和MRC)回归到22个基于结构、功能和扩散的神经成像指标上。一些参数,例如作为神经突复杂性代理(proxy)的取向离散指数(orientation dispersion index),与所有线粒体指标呈正相关(扩展数据表1)。相比之下,虽然一些MRI指标与线粒体OXPHOS没有显著关系,但其他指标则显著相关,根据具体特征显示出正或负的回归系数(扩展数据表1)。
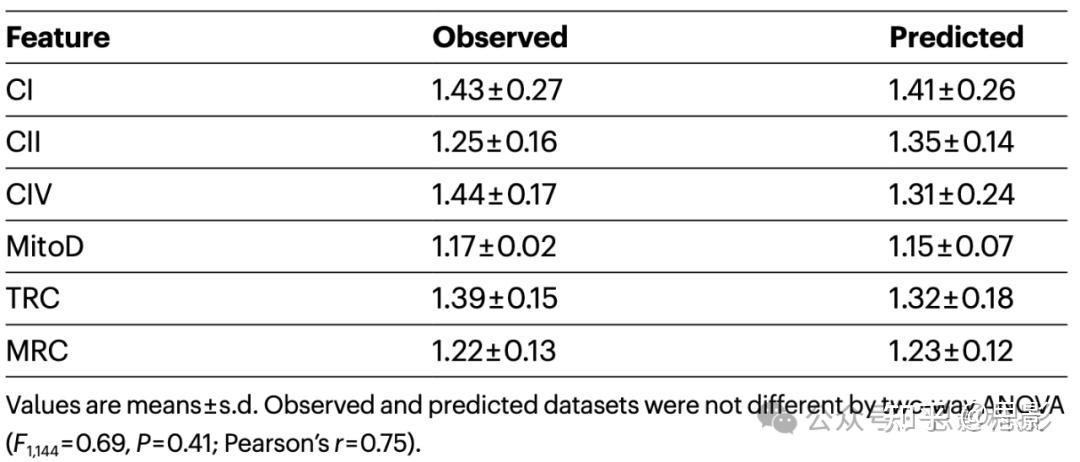
利用回归模型的线性方程,仅基于MRI指标对样本外体素(20%的样本)中线粒体特征的预测准确度在r2 = 0.26到0.36之间(所有P < 0.0001;图4a)。相比之下,使用打乱体素的零模型(null model)进行预测返回的准确度<0.1%(P > 0.7;扩展数据图9c)。当测试样本被归并到它们各自的脑区时,观察值与预期值的相关性增强了两到三倍,这强调了线粒体类型中稳健的区域变异(图4b)。模型的样本外准确性还通过使用基于MRI的模型来预测来自同一捐赠者大脑枕叶(未用于预测模型)的线粒体标记物进行了测试。当比较六个线粒体特征时,模型在观察值和预测值之间产生了显著的一致性(r = 0.75;表1和图4h)。在经过生化检测的冠状切片上投影的预测和观察到的线粒体特征图谱显示出显著的相似性(扩展数据图9d)。这一结果为模型的准确性提供了额外的支持。对变量进行非线性变换的进一步探索并未显著提高样本外预测准确性(详见方法)。
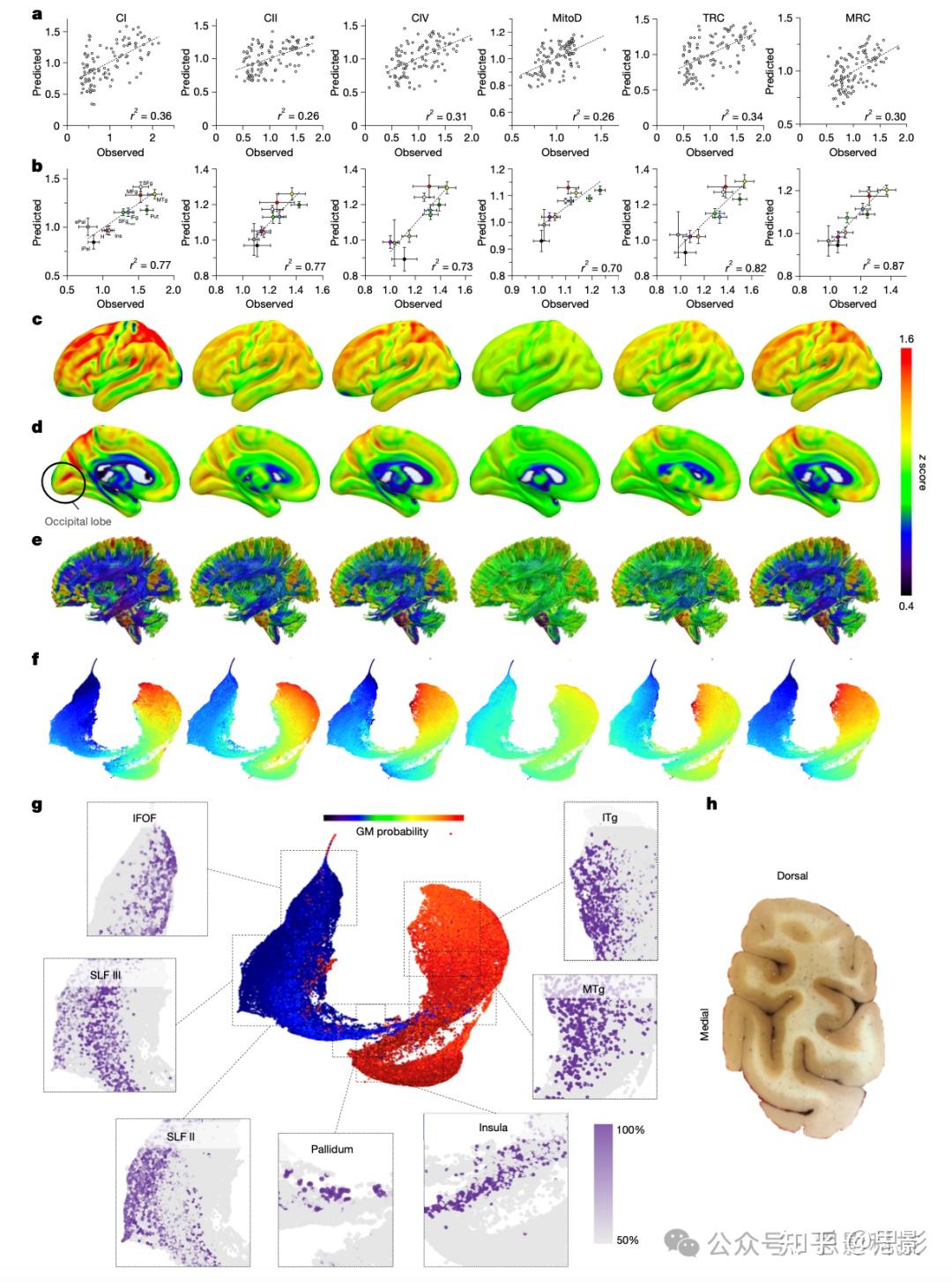
图 4:全脑投影的线粒体活性
a, 线粒体谱20%样本外预测的散点图(扩展数据图9)。
b, 针对脑区而非单个样本的观察到和预测的线粒体特征之间的相关性。学习和测试样本均被使用。n = 5个体素(ePal和iPal),7个体素(MTg),9个体素(MFg),11个体素(IFgmed),15个体素(H, Put和SFg),27个体素(Ins)和33个体素(IFg)。a和b中所有线性回归的斜率均不为零(P < 0.0001;每个图上显示了Pearson's r2)。
c–e, 外侧(c)和内侧(d)表面以及WM连接(e,侧视图)的全脑值预测。
f, 1毫米³分辨率全脑的UMAP(均匀流形近似与投影)嵌入,颜色表示线粒体活性谱。
g, 全脑UMAP图,颜色表示每个点位于WM或GM的概率。插图显示了体素位于特定WM(下额枕束,IFOF;上纵束,SLF)或GM(苍白球,岛叶,MTg,颞下回,ITg)脑结构的概率(P < 0.0001,经Bonferroni校正多重比较)。
h, 铣削前的枕叶脑板图像。用作上样对照(loading controls)的合并GM枕叶样本中线粒体特征的总结(扩展数据图2d),以及通过对MNI空间中十个随机选择的枕叶体素中基于MRI模型预测的线粒体指标进行平均生成的预测值,显示在表1中。
表 1 样本外(枕叶)脑区观察到和预测的线粒体参数之间的相关性
受模型能够基于MRI特征预测样本外线粒体特征能力的鼓舞,我们接下来将其扩展到计算全脑范围内的线粒体密度和OXPHOS活性。由此产生的皮层和WM通路图谱揭示了所有线粒体特征的区域特化(n = 1,827,243个体素,1毫米³分辨率;图4c-e)。线粒体特化对于MRC(线粒体呼吸能力)和OXPHOS酶尤其高,而对于线粒体含量(MitoD)则相对温和。这一结果表明,预计不同脑区在线粒体含量上的差异较小,而在ATP合成的线粒体特化(即MRC)上差异则大得多。相应地,我们基于神经成像的全脑MRC估计值与估计大脑进化(进化扩张和进化变异性)的图谱显示出显著的相关性(扩展数据图10),这一结果与图2l中基于区域的分析一致。
最后,我们生成了预测的全脑线粒体类型(解剖注释置信度 > 80%)的UMAP(均匀流形近似与投影)表征,这进一步证实了所有线粒体特征在GM和WM区域之间的显著对比(图4f,g)。同样,梯度幅度对于CI、CII和CIV活性以及MRC尤其大,这反映了预测的线粒体区域特化。此外,多变量UMAP空间与皮层和皮层下GM区域以及WM束的解剖学和立体定向坐标映射得相对较好,包括我们初始脑切片中未呈现的区域。因此,这些结果建立了一种利用标准MRI参数来预测整个人脑生化和分子线粒体谱的方法。
讨论
我们生成了一份涵盖人脑线粒体含量、酶促OXPHOS活性和特化的图谱,该图谱以与MRI相当的分辨率确定了线粒体类型(mitotypes)的多样性。通过一种物理体素化(physically voxelize)冷冻人脑组织的方法,我们的结果揭示了区域层面和细胞类型层面的因素共同促成了在整个人脑中观察到的分子线粒体类型的多样性。值得注意的是,GM和WM之间不同的线粒体类型与众所周知的脑组织间代谢需求的区域差异一致。这一发现突显了线粒体疾病可能倾向于影响皮层结构,例如卒中样发作(stroke-like episodes)选择性地影响并跨越皮层区域传播,其拓扑结构并不局限于动脉血管区域。这些数据也可能暗示WM中存在代偿机制,这些机制可能在诸如卒中等疾病中减轻皮层缺陷。
在分子层面上为能量转换而特化的线粒体(高MRC)与进化模式的一致性,揭示了为维持人类特有的、增加的能量成本而进化出的潜在亚细胞生物能量基础设施,特别是在与人类特有认知功能相关的区域。近期的工作已经强调了被招募来执行执行功能的皮层脑区如何表达高水平的神经调节剂细胞表面受体,并高度依赖葡萄糖消耗。这种依赖性也与遗传性线粒体疾病个体中进化上先进的认知功能的神经心理学易损性,以及与年龄相关的线粒体基因组缺陷累积所影响的认知领域相一致。
迅速积累的证据表明,神经精神疾病和与年龄相关的脑部疾病涉及线粒体改变,这激发了基础和临床研究界对活体大脑中线粒体生物学进行成像的兴趣。与基于PET的代谢成像并行,我们使用常见的神经成像模态预测全脑分子线粒体类型的模型满足了这一需求,并为弥合认知神经科学与线粒体生物学之间尺度差距的新问题打开了大门。
然而,应注意当前研究的局限性。我们的预测模型基于来自群体模板的神经成像数据,并受到个体间解剖变异性的影响。依赖于单个神经典型人脑样本,类似于其他全脑显微镜计划,强调了需要额外的死后数据集来确定我们的神经成像模型对来自不同年龄和性别的其他健康和病理样本的普适性(generalizability)。一个可普适的模型将意味着,在精确映射到人脑解剖结构的小组织样本(或多个样本)中进行的线粒体检测可以为整个大脑生成预测和假设。这项任务的规模在技术上具有挑战性。尽管如此,绘制亚细胞能量结构(在特定细胞类型和不同脑区之间)的个体间差异,对于推进我们理解支持并可能指导不同大脑中大尺度脑活动动态的能量基础设施至关重要。
总之,我们的物理体素化方法和分子表型研究确定了人脑中线粒体多样性的空间分布,并揭示了GM与WM之间以及关键大脑区域之间的显著差异。这项工作不仅揭示了一个与大脑系统发育平行的梯度,而且还提供了一种可扩展的方法来解读人脑进化、健康发育和疾病的线粒体基础。弥合从细胞器到全脑生物学和神经成像的尺度差距,为理解在各种背景下大脑功能和功能障碍的线粒体和能量基础奠定了基础。




















 1036
1036

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








