
文章目录
ABSTRACT
从静止状态功能磁共振成像(rsfMRI)准确识别自闭症谱系障碍(ASD)是一项具有挑战性的任务,这在很大程度上是由于ASD的异质性。 最近的工作表明,使用以rsfMRI时间序列为输入的递归神经网络,分类精度更高。 但是,表型特征(通常可用且可能带有预测信息)已从模型中排除,并且将此类数据与rsfMRI结合到递归神经网络中并不是一项简单的任务。 在本文中,我们介绍了几种将表型数据与rsfMRI合并到单个深度学习框架中以对ASD进行分类的方法。 我们在自闭症脑成像数据交换的大型,异类首批队列中使用交叉验证框架测试了建议的体系结构。 我们最好的模型达到了70.1%的准确度,优于以前的工作。
1. INTRODUCTION
静止状态功能磁共振成像(rsfMRI)已成为研究自闭症谱系障碍(ASD)的神经病理生理学的重要工具,揭示了自闭症患者与典型神经系统对照相比大脑活动的变化[1]。 找到能够准确地区分ASD和神经型个体的大脑活动差异,将更好地表征ASD的根本原因,从而改善诊断和治疗。
从rsfMRI学习ASD和神经型受试者分类的传统方法是将静止状态连接作为学习算法(例如逻辑回归和支持向量机)的输入特征[2,3]。 然而,最近的研究表明使用递归神经网络的深度学习方法可以提高分类的准确性[4]。 该方法建立在长短期记忆(LSTM)[5]架构的基础上,该架构允许rsfMRI时间序列直接用作输入。
尽管提高了准确性,但很大程度上由于ASD的异构性,ASD分类问题仍然非常具有挑战性。 鉴于该疾病的神经发育特性,人们希望包括表型信息(例如性别,年龄)将提高分类的准确性。 传统的学习算法可以简单地包括诸如附加输入变量等表型特征[6],但是在深度学习中,通常需要针对特定的多模式任务设计网络。 因此,将表型信息组合到为rsfMRI建立的神经网络中并不容易。
在这项工作中,我们探索了将表型数据和rsfMRI数据合并到单个神经网络模型中以对具有ASD和神经型对照的个体进行分类的方法。 我们使用自闭症脑成像数据交换(ABIDE)的第一个数据集测试了提出的网络体系结构,该数据集包括非常大的异类自闭症和对照受试者的表型和神经影像数据[7]。 使用10倍交叉验证对模型进行了验证,并将其与其他最近在ABIDE队列中进行测试的最新工作进行了比较。
2. METHODS
2.1. LSTM for Classification from rsfMRI
最近的研究表明,使用基于LSTM的架构,可以从rsfMRI时间序列输入中直接对ASD进行分类,结果很有希望[4],如图1a所示。 rsfMRI时间序列用作网络的输入 f f f。 LSTM单元在每个时间步的输出将被馈送到具有单个节点的dense层中(即,该节点的权重将在整个时间上共享)。 然后将来自单个节点的输出取平均,得到一个输入到S型激活函数中的单个值,以给出属于ASD类的概率。 在训练期间,将dropout应用于LSTM权重和dense层之后。 我们将此网络用作处理rsfMRI数据进行分类的基线模型。
2.2. Incorporating Phenotypic Information
我们提出了几种将表型和功能磁共振成像数据组合到单个网络中的方法。 它们在此处大致按网络深度的顺序列出,在该深度下,表型数据与rsfMRI信息合并在一起。
-
将表型数据和rsfMRI时间序列直接输入LSTM模型(Phenotype-TS,图1b)。首先沿时间轴重复表型数据,使其具有与rsfMRI时间序列相同的维度。然后,将表型数据沿着特征维连接到时间序列数据,并将其直接输入LSTM。这种方法允许表型数据与LSTM细胞内的rsfMRI数据直接相互作用。


-
直接从rsfMRI输入的LSTM输出中组合表型数据(RawPhenotype-LSTM,图1c)。再次沿时间轴复制表型数据,使其具有与rsfMRI时间序列相同的维度。然后将表型数据和LSTM输出输入到D个dense层中,最后一个层中有一个节点。该模型将允许表型数据与重要的大脑网络进行交互以进行分类[4]。
-
编码表型特征,然后与来自rsfMRI输入的LSTM输出结合(EncPhenotype-LSTM,图1d)。首先,表型数据由一个或多个密集层编码。然后重复表型数据的新表示,以在时间上具有与rsfMRI时间序列相同的维度。然后将转换后的表型信息和LSTM输出输入到D个密集层中,最后一个层中有一个节点。
-
直接将表型数据与rsfMRI输入的得分结合起来(RawPhenotype-rsfScore,图1e)。表型数据和LSTM模型的最终输出被输入到 D D D个密集层中,最后一个层中有一个节点。该模型为原始表型数据提供了对分类评分最直接的影响。
-
对表型特征进行编码,然后与rsfMRI输入的分数结合(EncPhenotype-rsfScore,图1f)。 首先使用一个或多个密集层对表型信息进行编码。 LSTM模型的编码表型和最终输出被输入到 D D D个密集层中,最后是具有单个节点的层。 在组合模式之前,此模型具有最独立的处理。
-
将表型数据设置为目标变量(Phenotype-Target,图1g)。 辅助损失函数改善了对目标目标的学习[8]。 代替使用表型信息作为输入,在多任务学习设置中将表型数据 p p p视为与ASD分类标签l一起估算的辅助目标值,即输出 y = [ l , p ] y = [l,p] y=[l,p]。 由于自闭症分类是主要目标,因此我们使用以下损失函数L:
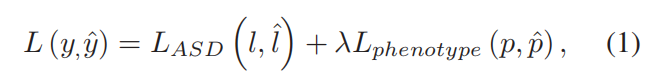
其中 y ^ \hat{y} y^是估计的输出值,LASD是ASD分类标签的损失, L p h e n o t y p e Lphenotype Lphenotype是表型特征的损失,而 λ \lambda λ控制表型损失项的贡献。 虽然表型数据不直接与rsfMRI数据交互,但这种方法训练LSTM模型与表型数据一致。
3. EXPERIMENTS
3.1. Data and Preprocessing
我们对来自ABIDE I队列的数据进行了实验[7]。 我们使用了由Connectome计算系统管道生成的,预处理Connectomes项目发布的数据,而没有全局信号回归,并且具有带通滤波[9]。 这导致来自17个不同成像部位的529名自闭症受试者和571名典型对照。
用于神经网络的rsfMRI输入来自从Craddock 200图集定义的感兴趣区域中提取的平均时间序列[10]。 将每个时间序列归一化为相对于平均信号的变化百分比,并以2 s的间隔重新采样以使数据达到相同的标度。 此外,由于不同的站点使用不同的扫描长度,因此我们将时间序列数据随机裁剪为长度 T = 90 T =90 T=90。每个对象随机进行10次裁剪,从而将数据集扩大到 N = 11000 N = 11000 N=11000个样本。 每个样本的神经网络功能磁共振成像输入 f i f_i fi就是大小为 90 × 200 90×200 90×200的矩阵。
对于表型输入,我们纳入了所有受试者的完整或几乎完整的数据:年龄,性别,惯用性,完全智商和fMRI扫描过程中的眼睛状态(根据成像协议打开或关闭)。 请注意,我们没有将成像位置用作变量,因此该模型将适用于其他位置的新数据。 惯性被编码为序数变量,其中3个级别 { − 1 , 0 , 1 } \{-1,0,1\} {−1,0,1}分别代表左优势,灵巧和右优势。 由于大多数人都是惯用右手的,因此缺乏惯用性数据的受试者被分配到了正确的优势类别。 智商未达到满分的受试者的得分为100,根据定义,这是平均智商。 将每种表型的数据归一化为 [ − 1 , 1 ] [-1,1] [−1,1]。 因此,每个样本的表型数据 p i p_i pi是尺寸为 P = 5 P = 5 P=5的向量。
3.2. Methods Implementation and Evaluation
使用具有二进制交叉熵损失函数的Keras [11]实现神经网络模型,但由于许多表型是连续变量,因此辅助目标模型采用均方误差实现。应用了adadelta优化器和默认初始化。 LSTM输出尺寸固定为32,训练期间的dropout设置为0.5,密实层(最终输出除外)应用了tanh激活。
在实验中,我们测试了
- 1)仅使用rsfMRI的基准模型(图1a),
- 2)仅使用表型数据的多层感知器模型以及
- 3)2.2节中提出的模型。
对于仅表型模型,我们使用1个具有P个节点的隐藏层。对于具有表型数据编码的模型,我们实现了1个具有P个节点的密集层(EncA)和2个密集层,其中第一层具有P个节点,第二层具有1个节点(EncB)。对于表型和rsfMRI信息的合并,我们测试了 D = 1 D = 1 D=1和 D = 2 D = 2 D=2,并且隐藏层中的节点数等于输入数。辅助目标模型以 λ = 0.1 \lambda= 0.1 λ=0.1进行训练。
我们使用10倍交叉验证评估了不同神经网络模型的性能,并根据成像部位进行了分层抽样。使用85%的训练,5%的验证和10%的测试对每一折的数据进行拆分,并将所有样本均分配给一个分区。当验证损失在10个epochs没有减少时,停止训练。使用最终训练的模型对测试数据进行的预测用于评估模型的性能。
我们使用“样本准确性”和“主题准确性”(分别对(增强数据集的)每个样本和每个受试者进行分类的准确性)来评估性能。受试者标签是根据受试者所有样品的平均分数确定的。我们计算了交叉验证折叠的准确性的均值和标准差(SD)。由于使用了相同的数据分割,并且每个模型的观察结果数量很少(10),因此我们使用配对的单尾t检验 ( p < 0.1 ) (p <0.1) (p<0.1)对结果的显着改善进行了测试。最后,为了更好地与使用ABIDE队列的不同子集的先前作品进行比较,我们计算了nominal accuracy 和 chance level(数据集中更常见类别的百分比)之间的差异。
3.3. Results and Discussion
表1总结了不同分类方法的性能。 前几行突出显示了先前在大部分ABIDE队列中进行测试并使用rsfMRI和表型数据的工作。 Parisot等 [12]产生了最高的准确性结果; 然而,他们使用最少的受试者,修剪困难的数据,并且依赖于站点作为表型特征,这使得该模型无法推广到从新的成像站点获得的数据。

表1的底部显示了已实现的神经网络的结果。在几乎所有模型中,受试者精度都优于样本精度,这表明了数据增强的重要性。仅基于rsfMRI的基线模型已经与具有表型信息的最佳发表模型相似[12](请参阅倒数第二栏)。仅使用表型数据会导致chance level增加8.5%,这表明此类信息有助于ASD分类。通常,首先将表型编码为单分数(EncB)的模型会导致准确性较差,而将原始表型数据直接与基线模型输出组合会更好。最佳模型将rsfMRI处理后的表型数据与最终得分结合起来,使用1个隐藏层。与仅使用rsfMRI的模型相比,其准确性提高了2.2%。此外,其准确性比以前最好的工作高2.4%,而没有包括成像部位,这可以改善我们模型的通用性。尽管名义上准确度的提高似乎不大,但ASD分类问题非常困难,随着数据集大小的增加,小的改进将产生更大的影响。
4. CONCLUSIONS
在这项工作中,我们探索了几种不同的神经网络方法,这些方法将表型数据与rsfMRI相结合以对ASD和神经型受试者进行分类。 我们最好的模型将表型数据与rsfMRI得分相结合,在ABIDE数据集上的分类精度为70.1%(比 naive
labeling高18.2%),胜过最近的工作。
未来的方向包括修改架构以合并结构MRI。 而且,包括行为措施可以进一步提高分类准确性。 最后,将探讨将表型特征包括在内对所学习的重要功能网络进行分类的影响。






















 536
536











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








