Android项目常见问题解决办法
目录
(3)修改gradle/wrapper/gradle-wrapper.properties文件
3.解决dlopen failed: library "libomp.so" not found
1.Android Studio 2021开发版本
项目原始Android开发版本Android Studio 、SDK、NDK和CMake相关版本信息,请参考:
历史版本:Android Studio 下载文件归档 | Android Developers
- Android Studio 4.1.1
- JDK Java1.8(Jave 8) JAVA_VERSION="1.8.0_242"
- CMake 3.18.1
PS:如果你升级到Android Studio 2024可能会出现编译异常




2.Android Studio 2024开发版本
如果你的Android Studio是2024版本,或者更高版本,请参考如下修改文件
(1)修改app/build.gradle文件
增加NDK版本:
ndkVersion '21.1.6352462'

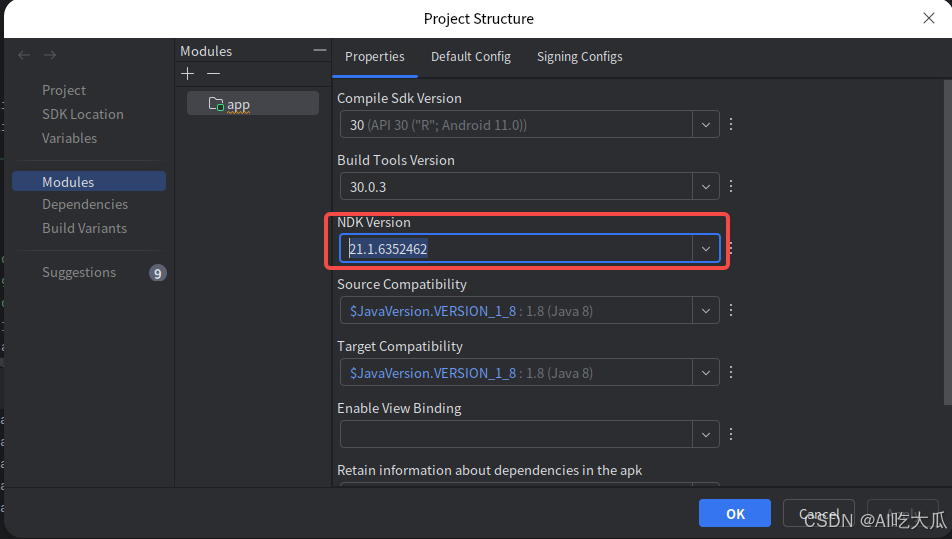
也可以在Project Structure修改NDK版本:
(2)修改build.gradle文件
将gradle升级到
classpath "com.android.tools.build:gradle:7.4.2"
repositories增加镜像url地址:
maven { url 'https://maven.aliyun.com/repository/public' }
maven { url 'https://maven.aliyun.com/repository/google' }

(3)修改gradle/wrapper/gradle-wrapper.properties文件
将gradle-6.5-all.zip->升级到gradle-7.5-bin.zip
distributionUrl=https\://services.gradle.org/distributions/gradle-7.5-bin.zip

修改完后,依次Clean Project-> Rebuild Project->Sync Project with Gradle files,即可正常build了
3.解决dlopen failed: library "libomp.so" not found
Android NDK开发时,如果开启多线程加速,需要用到openMP库,一些版本的差异,会出现dlopen failed: library "libomp.so" not found的错误,即找到libomp.so库

【尊重原则】转载请注明出处:解决dlopen failed: library “libomp.so“ not found_PKing666666的博客-CSDN博客_dlopen failed
解决方法有两种方式
(1)解决方法1
解决思路也很粗暴,直接将NDK里libomp.so拷贝到'src/main/jniLibs'或者'libs',即可解决问题。
比如我使用的是NDK版本是:android-ndk-r21e,在该目录下搜索libomp.so,
至于拷贝哪个处理器版本,需要根据你的平台选择,一般在abiFilters会指定 "armeabi-v7a", "arm64-v8a"平台,拷贝对应的版本即可;如果不存在,可以复制其他平台的libomp.so代替试试。
拷贝的目的地址,由jniLibs.srcDirs指定so库的位置:
举个例子:
- 比如将【aarch64】里面的libomp库复制到项目src/main/jniLibs中的 "armeabi-v7a", "arm64-v8a"目录下(目录不存在,则新建),然后再运行测试(大部分手机都可以这么解决)
- 如果还不能正常运行,则复制【arm】或者其他平台的libomp.so代替试试


(2)解决方法2(不推荐)
为了减小APK的大小,项目只适配了【"armeabi-v7a", "arm64-v8a" 】处理器架构库,这会导致部分手机不支持。build完成后,在app/build/intermediates/cmake/debug/obj可查看到已经编译且支持库;其中libomp.so可能因为一些差异导致没有生成或者没有找到

解决方法:
- 先删除之前build的中间产物:把app/.cxx和app/build目录都删除掉
- 注释掉app/build.gradle文件中:abiFilters "armeabi-v7a", "arm64-v8a" (这样就会生成所有处理器架构库,但打包APK会比较大)
- 重新build,run
- 重新查看app/build/intermediates/cmake/debug/obj,发现已经支持【"armeabi-v7a", "arm64-v8a","x86","x86_64"】等库



























 6778
6778

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










