无监督聚类 – Affinity Propagation
作者:钱魏Way
copy地址:https://www.biaodianfu.com/affinity-propagationap.html
AP(Affinity Propagation)通常被翻译为近邻传播算法或者亲和力传播算法。AP算法的基本思想是将全部数据点都当作潜在的聚类中心(称之为exemplar),然后数据点两两之间连线构成一个网络(相似度矩阵),再通过网络中各条边的消息(responsibility和availability)传递计算出各样本的聚类中心。

=====================================================================
AP算法中的特殊名词:
- Exemplar:指的是聚类中心,K-Means中的质心。
- Similarity(相似度):点j作为点i的聚类中心的能力,记为S(i,j)。一般使用负的欧式距离,所以S(i,j)越大,表示两个点距离越近,相似度也就越高。使用负的欧式距离,相似度是对称的,如果采用其他算法,相似度可能就不是对称的。
- Preference:指点i作为聚类中心的参考度(不能为0),取值为S对角线的值(图1红色标注部分),此值越大,最为聚类中心的可能性就越大。但是对角线的值为0,所以需要重新设置对角线的值,既可以根据实际情况设置不同的值,也可以设置成同一值。一般设置为S相似度值的中值。
- Responsibility(吸引度):指点k适合作为数据点i的聚类中心的程度,记为r(i,k)。如图2红色箭头所示,表示点i给点k发送信息,是一个点i选点k的过程。
- Availability(归属度):指点i选择点k作为其聚类中心的适合程度,记为a(i,k)。如图3红色箭头所示,表示点k给点i发送信息,是一个点k选点i的过程。
- Damping factor(阻尼系数):主要是起收敛作用的。

在实际计算应用中,最重要的两个参数(也是需要手动指定)是Preference和Damping factor。前者定了聚类数量的多少,值越大聚类数量越多;后者控制算法收敛效果。
==================================================================
AP算法流程:
步骤1:算法初始,将吸引度矩阵R和归属度矩阵初始化为0矩阵;
步骤2:更新吸引度矩阵
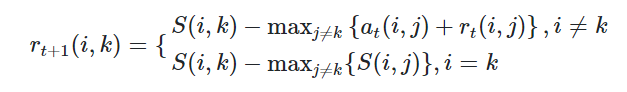
步骤3:更新归属度矩阵步骤4:根据衰减系数 对两个公式进行衰减
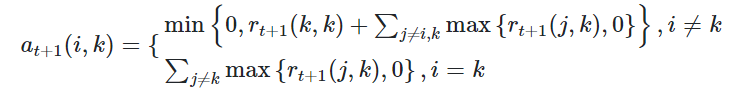
步骤4:根据衰减系数λ对两个公式进行衰减
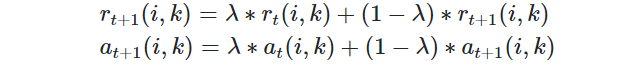
重复步骤2,3,4直至矩阵稳定或者达到最大迭代次数,算法结束。
最终取a+r最大的k作为聚类中心。
===================================================================
Python的机器学习库sklearn中已经实现了AP算法,可以直接调用。
参数设置介绍:
- damping : 衰减系数,默认为5
- convergence_iter : 迭代次后聚类中心没有变化,算法结束,默认为
- max_iter : 最大迭代次数,默认
- copy : 是否在元数据上进行计算,默认True,在复制后的数据上进行计算。
- preference : S的对角线上的值
- affinity :S矩阵(相似度),默认为euclidean(欧氏距离)矩阵,即对传入的X计算距离矩阵,也可以设置为precomputed,那么X就作为相似度矩阵。
训练完AP聚类之后可以获得的结果有
- cluster_centers_indices_ : 聚类中心的位置
- cluster_centers_ : 聚类中心
- labels_ : 类标签
- affinity_matrix_ : 最后输出的A矩阵
- n_iter_ :迭代次数
==========================================================================
from sklearn.cluster import AffinityPropagation
from sklearn import metrics
from sklearn.datasets import make_blobs
import numpy as np
# 生成测试数据
centers = [[1, 1], [-1, -1], [1, -1]]
X, labels_true = make_blobs(n_samples=300, centers=centers, cluster_std=0.5, random_state=0)
# AP模型拟合
af = AffinityPropagation(preference=-50).fit(X)
cluster_centers_indices = af.cluster_centers_indices_
labels = af.labels_
new_X = np.column_stack((X, labels))
n_clusters_ = len(cluster_centers_indices)
print('Estimated number of clusters: %d' % n_clusters_)
print("Homogeneity: %0.3f" % metrics.homogeneity_score(labels_true, labels))
print("Completeness: %0.3f" % metrics.completeness_score(labels_true, labels))
print("V-measure: %0.3f" % metrics.v_measure_score(labels_true, labels))
print("Adjusted Rand Index: %0.3f"
% metrics.adjusted_rand_score(labels_true, labels))
print("Adjusted Mutual Information: %0.3f"
% metrics.adjusted_mutual_info_score(labels_true, labels))
print("Silhouette Coefficient: %0.3f"
% metrics.silhouette_score(X, labels, metric='sqeuclidean'))
print('Top 10 sapmles:', new_X[:10])
# 图形展示
import matplotlib.pyplot as plt
from itertools import cycle
plt.close('all')
plt.figure(1)
plt.clf()
colors = cycle('bgrcmykbgrcmykbgrcmykbgrcmyk')
for k, col in zip(range(n_clusters_), colors):
class_members = labels == k
cluster_center = X[cluster_centers_indices[k]]
plt.plot(X[class_members, 0], X[class_members, 1], col + '.')
plt.plot(cluster_center[0], cluster_center[1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=14)
for x in X[class_members]:
plt.plot([cluster_center[0], x[0]], [cluster_center[1], x[1]], col)
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
Estimated number of clusters: 3
Homogeneity: 0.872
Completeness: 0.872
V-measure: 0.872
Adjusted Rand Index: 0.912
Adjusted Mutual Information: 0.871
Silhouette Coefficient: 0.753
Top 10 sapmles: [[ 1.47504421 0.9243214 0. ]
[-0.02204385 -0.80495334 1. ]
[-1.17671587 -1.80823709 2. ]
[ 0.77223375 1.00873958 0. ]
[ 1.23283122 0.23187816 0. ]
[-0.92174673 -0.88390948 2. ]
[ 1.65956844 -1.44120941 1. ]
[ 0.33389417 -1.98431234 1. ]
[-1.27143074 -0.79197498 2. ]
[ 1.33614738 1.20373092 0. ]]

===========================================================================
优点
- 聚类结果稳定,不受初始质心选择的影响
- 不需要先验设定聚成几类
- 对距离矩阵的对称性没要求。AP通过输入相似度矩阵来启动算法,因此允许数据呈非对称,数据适用范围非常大。
缺点
- AP聚类应用中需要手动指定Preference和Damping factor,这其实是原有的聚类“数量”控制的变体
- big O是 N^2(N是样本数量),所以样本数量越大运行时间越久。最好使用稀疏矩阵,密集矩阵会增加很多运算时间。
























 7461
7461











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








