目录
本博客仅作为个人学习笔记用途,欢迎和各位前辈交流学习
版本 Python=3.10 Ubuntu=24.04.2 LTS MintPy=1.6.1
本文记录博主使用hyp3+mintpy+pyAPS进行小基线集(SBAS-InSAR)时序insar分析操作的全流程,便于后续继续使用时的参考。
HyP3 是ASF提供的免费SAR图像处理服务,让用户轻松获得分析就绪的SAR数据,包括使用GAMMA进行地理编码操作后的RTC雷达影像、使用GAMMA或者isce处理的雷达干涉结果,用户简单地点点点就能获得SBAS前的干涉处理结果,虽然分辨率较低(InSAR-GAMMA对应40、80m分辨率),对于专注于时序insar分析、后处理等任务(对insar预处理无精细需求)的个人用户非常方便。
mintpy 是开源时序InSAR分析软件,支持输入ISCE, ARIA, FRInGE, HyP3, GMTSAR, SNAP, GAMMA生成的干涉序列获取使用SBAS-InSAR处理的时序InSAR形变分析结果,值得一提的是,开发者提供了非常详实的教程、注释和细节功能,很有利于InSAR初学者。
PyAPS 是mintpy中自带的insar大气延迟改正模块,该软件默认使用ERA5数据进行对流层大气延迟改正。
用户注册与准备工作
- 首先在https://urs.earthdata.nasa.gov/users/new中注册ASF Data Search账号,具体不展开。
- 准备python环境,安装mintpy:
conda install -c conda-forge mintpy
- 在https://cds.climate.copernicus.eu/注册ECMWF账号,用于后续pyAPS大气延迟校正中的ERA5数据下载。
注意
注册完成后,进入ERA5,下拉至Terms of use,查看licence并同意,才能后续使用下载。

pyAPS也需要配置,在python环境文件夹中找到pyaps所在的文件夹(如/home/anaconda3/envs/isce/lib/python3.10/site-packages/pyaps3),打开,找到model.cfg文件
在cds profile中找到自己账户对应的api token,复制,并粘贴在model.cfg文件中CDS对应的key中。


至此我们做好了准备工作。
HyP3 SBAS干涉序列生成
数据选择
进入ASF Vertex并登录,在页面中选择研究区域,筛选数据。

点击右侧Filters进行详细筛选,包括起止日期、升降轨、FileType选择L1(SLC)

注意
使用bbox圈出研究区域会把所有和bbox挨着的数据都显示出来,有时候由于研究区域较小,有些frame覆盖的区域太少,需要舍弃掉,如果你想使用单个frame的数据,可以点击这个frame,点击下方Frame旁边的设置图标->set as both来筛选单个frame的数据。

SBAS基线构成
完成后,选择某一frame作为参考影像,点击正下方的SBAS,进入小基线集筛选界面。这里可以调整小基线集的具体干涉对,设置有关阈值等操作,具体见下图。我使用垂直基线400m,时间基线36天,获得了一共117组干涉对。

完成小基线集设置后,点击左上方的On Demand按钮,选择InSAR-GAMMA->Add***即可提交insar干涉图序列生成申请。
InSAR干涉设置
提交On Demand后,在网站右上角找到On Demand图标->On Demand Queue,进入InSAR相关参数的设置。这里设置多视参数,10×2代表40m的pixel size,20×4对应80m,附带产品中勾选了Water Mask、DEM、以及雷达入射角等相关的几何参数文件。

设置完成后,点击提交。Hyp3会自动处理InSAR相关的操作,后续获得的结果都是地理编码过的tif文件,直接下载即可。在On Demand->Submmited Products中可以查看处理的进度。大概需要半天到一整天的时间。
使用Python脚本进行数据下载
输出处理完成之后,点击左上角加购物车按纽添加到Queue。

此时再查看网页右上角的购物车图表,你的数据文件已经全部添加到Downloads中,
打开列表。选择右下角的Data Download下载Python脚本。
这里我是科学上网进行下载的,没有问题,如果报错没网络,那就在ASF网站里用Chrome浏览器下载,点击Download All即可

在终端中打开你准备存放数据的文件夹,使用python运行对应的脚本即可开始下载,使用时,需要在terminal界面中输入你的ASF用户名和密码。python脚本会自动检查你的文件夹中已经下载的文件,所以使用python脚本下载中途中断可以支持重新下载,放心下。

下载完成后,解压所有文件,每个文件夹当中都保存了一组干涉图像的全部信息:
md.txt 文件:记录这个干涉的摘要信息以及数据介绍,可供参考。
_dem.tif:处理所用的数字高程模型(DEM)图像。
_color_phase.png / _color_phase.kmz:干涉图的缠绕相位彩色图像。
_unw_phase.tif:解缠干涉图的TIFF数据。
_unw_phase.png / _unw_phase.kmz:干涉图的解缠后的可视化图像。
_los_disp.tif:视线方向位移图,表示沿雷达视线方向的地表位移。
_vert_disp.tif:垂直方向位移图,假设位移完全为垂直方向。
_corr.tif:相干性图,显示两景SAR影像之间的insar相干性。
_amp.tif:幅度图,显示参考图像的后向散射强度,就是雷达影像。
_lv_theta.tif / _lv_phi.tif:视线向量图,分别表示视线的仰角和方位角。大气校正需要。
_inc_map.tif / _inc_map_ell.tif:入射角图,分别表示局部入射角和椭球体入射角。
_water_mask.tif:水体掩膜,标识水体区域。
所有的tif文件都又对应的xml参数文件。
至此我们准备好了全部数据,接下来开始进行处理。

MintPy时序InSAR处理
数据裁剪
刚拿到手的数据涵盖了Sentinel-1中IW1、2、3三个条带的区域,范围非常大,需要进行裁剪之后再进一步处理。裁剪代码如下,参考了ASF HyP3 Mintpy处理教程。
from pathlib import Path
from typing import List, Union
from osgeo import gdal
def clip_hyp3_products_to_common_overlap(data_dir: Union[str, Path], overlap: List[float]) -> None:
"""Clip all GeoTIFF files to their common overlap
Args:
data_dir:
directory containing the GeoTIFF files to clip
overlap:
a list of the upper-left x, upper-left y, lower-right-x, and lower-tight y
corner coordinates of the common overlap
Returns: None
"""
files_for_mintpy = ['_water_mask.tif', '_corr.tif', '_unw_phase.tif', '_dem.tif', '_lv_theta.tif', '_lv_phi.tif']
for extension in files_for_mintpy:
for file in data_dir.rglob(f'*{extension}'):
dst_file = file.parent / f'{file.stem}_clipped{file.suffix}'
gdal.Translate(destName=str(dst_file), srcDS=str(file), projWin=overlap)
#这里填写数据文件夹的根目录
data_dir =Path('/mnt/d/Baihetan/')
# overlap = [ulx, uly, lrx, lry],研究区域的坐标,Hyp3是UTM投影下的,可以使用GIS软件查看tif获取
overlap = [286999,3036303,297850,2995684]
clip_hyp3_products_to_common_overlap(data_dir, overlap)
运行完成后,各个hyp3干涉文件夹中都会出现**_clipped.tif这种后缀的文件,这就是裁剪获得的文件。
配置mintpy参数文件
Mintpy的运行需要进行详细的配置,里边有非常多的参数和功能需要人工DIY,基本的操作是我们自己定一个配置文件(txt或者cfg)然后使用smallbaselineApp.py smallbaselineApp.cfg直接运行,mintpy会完整运行全部时序处理的流程。
这里我们先自定义一个config文件,在工作文件夹中创建name.txt文件,输入:
mintpy.load.updateMode = no #[yes / no], auto for yes, skip re-loading if HDF5 files are complete
mintpy.load.processor = hyp3 #[isce, aria, hyp3, gmtsar, snap, gamma, roipac, nisar], auto for isce
##---------interferogram stack:
mintpy.load.unwFile = /Path to your dataset/*/*_unw_phase_clipped.tif #[path pattern of unwrapped interferogram files]
mintpy.load.corFile = /Path to your dataset/*/*_corr_clipped.tif #[path pattern of spatial coherence files]
##---------geometry:
mintpy.load.demFile = /Path to your dataset/*/*_dem_clipped.tif #[path of DEM file]
mintpy.load.incAngleFile = /Path to your dataset/*/*_lv_theta_clipped.tif #[path of incidence angle file], optional but recommended
mintpy.load.azAngleFile = /Path to your dataset/*/*_lv_phi_clipped.tif #[path of azimuth angle file], optional
mintpy.load.waterMaskFile = /Path to your dataset/*/*_water_mask_clipped.tif #[path of water mask file], optional but recommended
mintpy.reference.date = no #[reference_date.txt / 20090214 / no], auto for reference_date.txt
mintpy.troposphericDelay.method = pyaps #[pyaps / height_correlation / gacos / no], auto for pyaps
mintpy.troposphericDelay.weatherModel = ERA5 #[ERA5 / MERRA / NARR], auto for ERA5
mintpy.network.minCoherence = 0.6 #[0.0-1.0], auto for 0.7
注意: 使用的时候要把/Path to your dataset这一部分修改成自己的数据文件夹地址,也就是存放了所有hyp3文件夹的数据文件夹地址。
上面的这个参数文件代表的含义:
updateMode:no 表示每次都重头运行全部内容
mintpy.load.processor = hyp3 指定数据为hyp3
中间几个为数据地址
mintpy.reference.date = no 设定第一个日期为参考日期
mintpy.troposphericDelay.method = pyaps #[pyaps / height_correlation / gacos / no], auto for pyaps 使用pyaps进行外部数据法的对流层延迟改正
mintpy.troposphericDelay.weatherModel = ERA5 #[ERA5 / MERRA / NARR], auto for ERA5,使用ERA5数据进行对流层延迟改正
mintpy.network.minCoherence = 0.6 相干性阈值
当然,MintPy的功能远不止这些,如果你想DIY,直接在命令行中输入:
smallbaselineApp.py -g
该命令会在当前文件夹中生成一个smallbaselineApp.cfg文件,文件当中存放了所有配置的完整模板,按照模板中的注释进行DIY配置即可。

执行smallbaselineApp.py
配置完你的配置文件之后,进入工作文件夹,打开terminal,在命令行中运行:
smallbaselineApp.py your_config_file,例如:
smallbaselineApp.py smallbaselineApp.cfg
注意
当前MintPy version 1.6.1版本下载ERA5数据时可能会出现问题
如果在correct_troposphere过程中出现问题,可以参考:
MintPy1.6.1使用PyAPS下载ERA5数据失败处理方法
如果能顺利运行完毕,就说明全部跑通了,可以开始查看结果。
结果查看
运行完成smallbaselineApp.py后,工作文件夹中会出现很多结果,这里挑几个重点结果进行说明。
inputs文件夹中存放了mintpy读入的数据
ifgramStack.h5 全部干涉图相位信息
geometryGeo.h5 地理编码、雷达视角、DEM等相关信息,绘图的时候可以用到
ERA5.h5 使用ERA5计算的各个日期的大气延迟量
工作文件夹中存放了时序InSAR后处理结果
timeseries.h5 经过SBAS解算的累积形变序列
timeseries_ERA5.h5 经过SBAS解算–>ERA5对流层大气延迟改正的累积形变序列
timeseries_ERA5_demErr.h5 经过SBAS解算–>ERA5对流层大气延迟改正–>DEM误差改正的累积形变序列
maskTempCoh.h5 相干性掩膜,绘图的时候滤除时间相干性较差的点。
velocity.h5 形变速率、速率标准差等相关数据
pic文件夹中存放了所有可视化结果
可以看到,mintpy的所有数据都是用hdf5数据类型存储的,结果都是.h5文件,可以使用h5py库进行查看修改。mintpy软件中也自带了很多脚本命令可以使用。
1. 使用info.py查看h5数据
对于一个h5文件我们不清楚它里边储存了什么内容,或者不了解这个mintpy文件工程的内容,可以使用info.py name.h5进行查看,name就是这个h5文件名。例如,我们可以查看./inputs/ifgramStack.h5文件的信息:
info.py ./inputs/ifgramStack.h5
首先会输出该mintpy工程在处理时使用的参数,然后在结尾出给出这个h5文件的数据摘要。
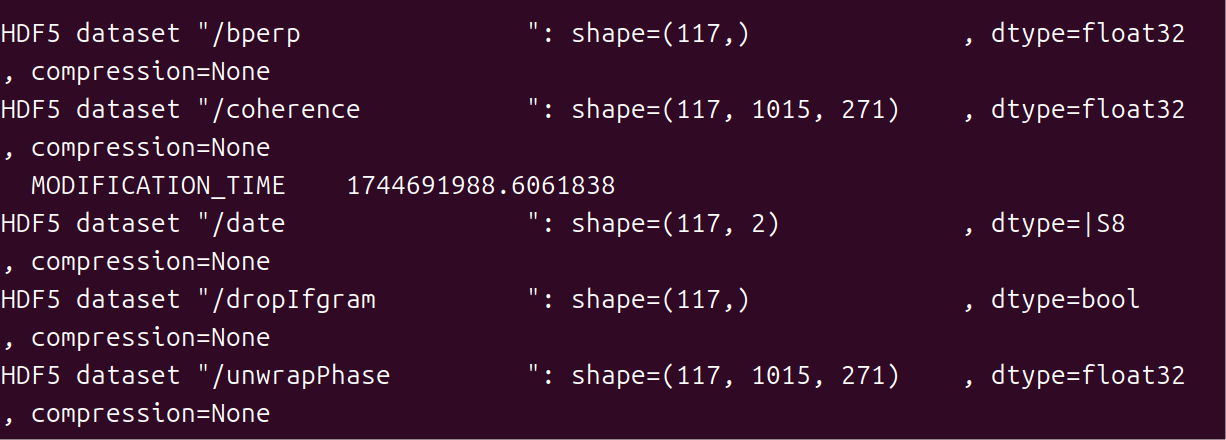
详细查看某一变量,在info.py ./inputs/ifgramStack.h5命令后加入--dset dataset_name即可,例如
info.py ./inputs/ifgramStack.h5 --dset dropIfgram
这个命令会输出ifgramStack.h5文件中存储的dropIfgram这一变量的值,可以看到,本次处理使用了全部的干涉图数据,没有干涉图被drop

2. 使用view.py查看图像数据
view.py是mintpy自带的一个非常强大的可视化脚本,功能很多没办法一一说明,这里简单给个例子来记录。
view.py timeseries.h5 '*2022'
这个命令会画出2022年的timeseries累积形变序列图。timeseries.h5中记录的每一个日期的图命名为:‘timeseries-date’,所以这里'*2022'表示搜索所有2022年的数据,同理,'*202204'表示搜索2022年4月的全部数据。

view.py timeseries.h5 '*202203' '*202204' -d inputs/geometryGeo.h5 --dem-nocontour
绘制了2022年3月-4月的timeseries,-d指定了绘图时使用geometryGeo.h5中的DEM作为DEM底图,--dem-nocontour表示在绘制DEM的时候不加入等高线,结果如下:

接下来我们以某一日期为例比较一下ERA5对流层延迟改正的效果。
view.py timeseries.h5 '*20220306_20220318' -d inputs/geometryGeo.h5 --dem-nocontour --mask no -v -5 5 --title '20220306_20220318' --noaxis
view.py timeseries_ERA5.h5 '*20220306_20220318' -d inputs/geometryGeo.h5 --dem-nocontour --mask no -v -5 5 --title '20220306_20220318 ERA5' --noaxis
'*20220306_20220318' 代表创建timeseries中这两个日期之差,可以用于表示SBAS结算过后的干涉相位差(单位为cm)
--mask 用于指定使用掩膜,no表示不用掩膜,这里我们主要是为了比较一下ERA5的对流层延迟改正效果。
-v vmin vmax 设置colorbar范围。
两幅图的结果如下:

可以看出,原始干涉图中存在较为明显的空间相关的对流层延迟量,pyAPS使用ERA5有效改正了对流层延迟。
除了上述的功能,view.py还有很多可以DIY的选项,具体参数内容可以查看view.py -h
3. 使用tsview.py交互界面查看形变时间序列
运行tsview.py timeseries.h5可以打开一个交互式窗口展示从开始日期到结束日期的timeseries图,通过键盘左右键切换日期;使用鼠标点击某一点,可以展示出该点位对应的形变序列。同时,tsview.py支持同时绘制多个timeseries的序列,例如,我们可以比较某点ERA5改正前后的累积形变序列,运行:
tsview.py timeseries_ERA5.h5 timeseries.h5
在交互界面中,点击某点,小窗口中会显示对应的时间序列,可以看到使用ERA5改正后的序列稳定很多。

这是我最近一次使用mintpy做SBAS的全流程,一路跑下来应该是没有问题的,如果有理解不正确的地方请在评论区指出,也欢迎各位给出更好的方法或者改进之处,共同交流学习。[握手]
























 2410
2410

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








