文章目录
流形学习
先来了解一下流形学习。
流形学习(Manifold Learning)是机器学习中的一种维数约简方法,将高维的数据映射到低维,并依然能够反映原高维数据的本质结构特征。流形学习的前提是假设某些高维数据实际是一种低维的流形结构嵌入在高维空间中。流形学习分为线性流形算法和非线性流形算法,线性流形算法包括主成分分析和线性判别分析,非线性流形算法包括局部线性嵌入(Locally Linear Embedding,LLE)、拉普拉斯特征映射(LE)等。
本文介绍这四种降维方法原理,实现步骤以及实现代码。
线性流形算法使用的是鸢尾花数据集,非线性流形算法使用的是“瑞士卷”数据集。如有错误,欢迎指正。
主成分分析(PCA)
原理
主成分分析(Principal Component Analysis,PCA)是最常用的线性降维方法,它的目标是通过某种线性投影,将高维的数据映射到低维的空间中,并期望在所投影的维度上数据的方差最大,以此使用较少的维度,同时保留较多原数据的维度。
主成分分析的降维是指经过正交变换后,形成新的特征集合,然后从中选择比较重要的一部分子特征集合,从而实现降维。这种方式并非是在原始特征中选择,所以PCA这种线性降维方式最大程度保留了原有的样本特征。
一般对于m条n维数据来说,PCA的步骤为:
① 将原始数据按列组成n行m列矩阵X;
② 计算矩阵X中每个特征属性(n维)的平均向量M(平均值);
③将X的每一行(代表一个属性字段)进行零均值化,即减去M;
④ 按照公式求出协方差矩阵;
⑤ 求出协方差矩阵的特征值及对应的特征向量;
⑥ 将特征向量按对应特征值从大到小按行排列成矩阵,取前k(k<n)行组成基向量P;
⑦ 通过Y=PX计算降维到k维后的样本特征。
公式:

PCA算法目标是求出样本数据的协方差矩阵的特征值和特征向量,而协方差矩阵的特征向量的方向就是PCA需要投影的方向。使样本数据向低维投影后,能尽可能表征原始的数据。协方差矩阵可以用散布矩阵代替,协方差矩阵乘以(n-1)就是散布矩阵,n为样本的数量。协方差矩阵和散布矩阵都是对称矩阵,主对角线是各个随机变量(各个维度)的方差。
实现
为了让大家理解更深一点,这里采用手写的方式,我会打印出每一个的维度,以便于更好理解他们的变化。当然为了方便,手写后面也会有掉包的部分。
在这来迫害 选用鸢尾花来展示,第一步导入数据集,sklearn的包里就有位置在

但我桌面上有个现成的,就拿来用了。
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
f = open('./Iris数据集/iris.csv')
data = pd.read_csv(f)
f.close()
data.head()

接下来把三种不同的类别变为数字
data['Species'][data['Species'] == 'setosa'] = 0
data['Species'][data['Species'] == 'versicolor'] = 1
data['Species'][data['Species'] == 'virginica'] = 2
data.head()

手写版
现在来求他们的均值
X = data.iloc[:,1:-1].values
X_mean = np.mean(X,axis=0)#求均值,axis=0指定列,1指定行
X_mean.shape
#输出为(4,)
零均值化,也叫中心化
X1 = X-X_mean
计算协方差矩阵
X_cov = np.cov(X1.T)#计算协方差矩阵
X_cov.shape#输出为(4, 4)
计算协方差矩阵的特征值及对应的特征向量
这里使用的是numpy的linalg模块,他包含了线性代数的函数,计算特征值和特征向量eig和eigh,都可以。不过eigh的输出是按照升序,即从小到大来排序的。他们都会输出两个矩阵,一个是特征值,另一个是特征向量。
X_val,X_vec = np.linalg.eigh(X_cov)#求解协方差矩阵的特征值和特征向量,升序
print(X_val.shape, X_vec.shape)#输出为(4,) (4, 4)

现在已经知道已经排好序的特征向量了,这里我要降到一维,所以使用最大特征值对应的特征向量,如果要降到二维,就选择最大的两个特征值对应的特征向量即可。
s_c = X_vec.T[-1:]#选择降到一维,这里有转置的,就是.T
s_c.shape#输出为(1, 4)
dot()是矩阵相乘,x为 m×n 阶矩阵,y为 n×p 阶矩阵,则相乘的结果为 m×p 阶矩阵。s_c为(1,4),X1为(150,4)
final_val = np.dot(X1,s_c.T)#这里有转置的
final_val.shape#输出为(150,1)
这样就得到了降维之后的数据,请看手写的和我调库的区别吧。可视化结果:

有点像转了180度
所以我们加上最后一步,重构数据
new_final_val = np.dot(final_val,s_c)+X_mean#重新构造数据
new_final_val.shape#输出为(150, 4)
可视化一下

调库版
from sklearn.decomposition import PCA
pca = PCA(n_components=1)
pca.fit(data.iloc[:,1:-1])
pca_val = pca.fit_transform(data.iloc[:,1:-1])
pca_val.shape#输出为(150, 1)
n_components=k,意思就是要把原数据降到k维
explained_variance_ratio_这个属性可以查看PCA的所有成分的贡献率
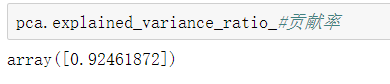
因为降到一维了,所以只有第一个主成分的贡献率。占了所有的92.46%
可视化来看一下,代码:
markers = ['o','*','+']
colors = ['r','b','g']
plt.figure(figsize=(12,4), dpi=80)
ax = plt.subplot(131)
for i in range(150):
ax.scatter(i,final_val[i,0],c=colors[int(data.iloc[:,-1][i])],marker=markers[int(data.iloc[:,-1][i])])
plt.xlabel('手写PCA')
plt.ylabel('PCA')
ax1 = plt.subplot(132)
for i in range(150):
ax1.scatter(i,pca_val[i,0],c=colors[int(data.iloc[:,-1][i])],marker=markers[int(data.iloc[:,-1][i])])
plt.xlabel('调库PCA')
plt.ylabel('PCA')
ax1 = plt.subplot(133)
for i in range(150):
ax1.scatter(i,new_final_val[i,0],c=colors[int(data.iloc[:,-1][i])],marker=markers[int(data.iloc[:,-1][i])])
plt.xlabel('手写重构数据PCA')
plt.ylabel('PCA')
plt.show()

缺点
从PCA的实现原理来看,这种变换并没有改变各样本之间的关系,只是应用了新的坐标系。在本例中是将三维空间转降到二维空间,如果有一个n维的数据,想要降低到k维。那么就取前k个特征值对应的特征向量即可。PCA的主要缺点是当数据量和数据维数非常大的时候,用协方差矩阵的方法解PCA会变得非常低效,解决办法是采用奇异值分解(Singular Value Decomposition,SVD)技术。
奇异值分解(SVD)
原理
对于任意m×n的输入矩阵A,SVD分解结果为:
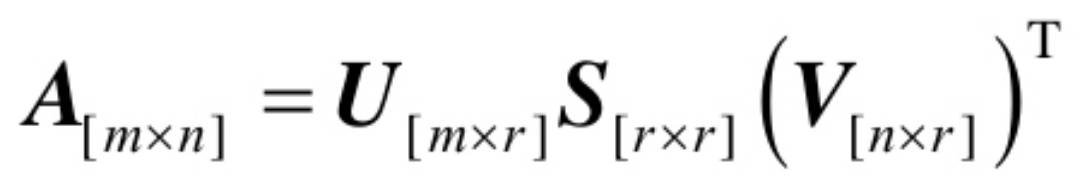
分解结果中U为左奇异矩阵,S为奇异值矩阵,除主对角线上的元素外全为0,主对角线上的每个元素都称为奇异值,V为右奇异矩阵。矩阵U、V中的列向量均为正交单位向量,而矩阵S为对角阵,并且从左上到右下以递减的顺序排序,可以直接借用SVD的结果来获取协方差矩阵的特征向量和特征值。
还是上面提到的numpy.linalg模块,包含了svd方法。
实现

得到左奇异矩阵U、奇异值矩阵S、右奇异值矩阵V。选择U中前2个特征分别作为二维平面的x、y坐标进行可视化,代码如下:
samples,features = data.iloc[:,1:-1].values.shape
U, s, V = np.linalg.svd(data.iloc[:,1:-1].values)
newdata = U[:,:2]
fig= plt.figure()
ax = fig.add_subplot(1,1,1)
markers = ['o','*','+']
colors = ['r','b','g']
for i in range(samples):
ax.scatter(newdata[i,0],newdata[i,1],c=colors[int(data.iloc[:,-1][i])],marker=markers[int(data.iloc[:,-1][i])])
plt.xlabel('SVD1')
plt.ylabel('SVD2')

线性判别分析(LDA)
原理
线性判别分析(Linear Discriminant Analysis,LDA)也称为Fisher Linear Discriminant,是一种有监督的线性降维算法。与PCA不同,LDA是为了使降维后的数据点尽可能容易地被区分。
线性判别分析在训练过程中,通过将训练样本投影到低维度上,使得同类别的投影点尽可能接近,异类别样本的投影点尽可能远离,即同类点的方差尽可能小,而类之间的方差尽可能大;对新样本,将其投影到低维空间,根据投影点的位置来确定其类别。PCA主要是从特征的协方差角度,去找到比较好的投影方式。LDA更多地考虑了标注,即希望投影后不同类别之间数据点的距离更大,同一类别的数据点更紧凑。
LDA的降维过程如下:
① 计算数据集中每个类别下所有样本的均值向量;
② 通过均值向量,计算类间散布矩阵SB和类内散布矩阵SW;
③ 依据公式1进行特征值求解,计算公式1结果的特征向量和特征值;
④ 按照特征值排序,选择前k个特征向量构成投影矩阵U;
⑤ 通过Y=X×U的特征值矩阵将所有样本转换到新的子空间中;
公式1:

手写版
X = data.iloc[:,1:-1].values
y = data.iloc[:,-1]
class_labels = np.unique(y)#储存的是类别,输出是[0,1,2]
mean_vectors = []
for i in range(3):
mean_vectors.append(np.mean(X[y == i],axis=0))#每个类别的特征均值
mean_vectors

计算类内散布矩阵SW
s_w = np.zeros((4,4))
for i,j in zip(range(3),mean_vectors):
sc_class = np.zeros((4,4))
for row in X[y == i]:
row,j = row.reshape(4,1),j.reshape(4,1)
sc_class += (row-j).dot((row-j).T)
s_w += sc_class

计算类间散布矩阵SB
overall_mean = np.mean(X,axis=0)
n_features = X.shape[1]#特征数量,其实在这里就是4啦
s_b = np.zeros((n_features,n_features))
for i,j in enumerate(mean_vectors):
n = X[y == i].shape[0]
j = j.reshape(n_features,1)
overall_mean = overall_mean.reshape(n_features,1)
s_b += n*(j - overall_mean).dot((j - overall_mean).T)
eig_vals, eig_vecs = np.linalg.eigh(np.linalg.inv(s_w).dot(s_b))#这里的np.linalg.inv是求逆的

前k个特征向量构成投影矩阵U,这里的k是1,降到一维。
再通过计算Y=X×U的特征值矩阵,实现降维。
d = np.dot(X,eig_vecs[:,-1])
fig= plt.figure()
ax = fig.add_subplot(1,1,1)
markers = ['o','*','+']
colors = ['r','b','g']
for i in range(150):
ax.scatter(i,d[i],c=colors[int(data.iloc[:,-1][i])],marker=markers[int(data.iloc[:,-1][i])])

调库版
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
lda = LinearDiscriminantAnalysis(n_components=1)#降到1维
lda.fit(X,data.iloc[:,-1].values.astype('float').reshape(X.shape[0],))
X_new = lda.transform(X)
plt.scatter(range(150), X_new[:, 0],marker='o',c=y)
plt.show()

PCA与LDA
LDA在求解过程中需要类内散度矩阵SW和类间散度矩阵SB,其中SW由两类扩展得到,而SB的定义则与两类有所不同,是由每类的均值和总体均值的乘积矩阵求和得到的。目标是求得一个矩阵U使得投影后类内散度尽量小,而类间散度尽量大。在多类情况下,散度表示为一个矩阵。一般情况下,LDA之前会做一次PCA,保证SW矩阵的正定性。
PCA降维是直接与数据维度相关的,例如原始数据是n维,那么使用PCA后,可以任意选最佳的k(k<n)维。LDA降维是与类别个数相关的,与数据本身的维度没关系,例如原始数据是n维的,一共有C个类别,那么LDA降维之后,可选的一般不超过C-1维。例如,假设图像分类,有两个类别为正例和反例,每个图像有1024维特征,那么LDA降维之后,就只有1维特征,而PCA可以选择降到100维。
LDA可以用来分类,大多数还是用来降维。
局部线性嵌入(LLE)
原理
局部线性嵌入是一种典型的非线性降维算法,这一算法要求每一个数据点都可以由其近邻点的线性加权组合构造得到,从而使降维后的数据也能基本保持原有流形结构。它是流形学习方法最经典的工作之一,后续的很多流形学习、降维方法都与其有密切联系。
局部线性嵌入寻求数据的低维投影,保留本地邻域内的距离。它可以被认为是一系列局部主成分分析,被全局比较以找到最佳的非线性嵌入。
步骤
算法的主要步骤分为三步:首先寻找每个样本点的k个近邻点;然后,由每个样本点的近邻点计算出该样本点的局部重建权值矩阵;最后,由该样本点的局部重建权值矩阵和近邻点计算出该样本点的输出值。
局限性
LLE在有些情况下也并不适用,例如数据分布在整个封闭的球面上,LLE则不能将它映射到二维空间,且不能保持原有的数据流形。因此在处理数据时,需要确保数据不是分布在闭合的球面或者椭球面上。
实现
sklearn中dataset中内置了生成此类数据的方法make_swiss_roll,生成样本数为1500个,X为生成的结果,作为LLE的输入。locally_linear_embedding方法执行LLE运算,n_neighbors=10表示近邻点的数量为10,n_components=2表示降维到二维。
from sklearn import manifold, datasets
X, color = datasets._samples_generator.make_swiss_roll(n_samples=1500)
X.shape为(1500, 3), color.shape为(1500,)
X_r, err = manifold.locally_linear_embedding(X,n_neighbors=10,n_components=2)#相邻点10个,降到2维
print(X_r.shape, err)
#(1500, 2) 3.715731105861033e-08
可视化一下原数据
from mpl_toolkits.mplot3d import Axes3D # 空间三维画图
fig = plt.figure()
ax = Axes3D(fig)
ax.scatter(X[:,0],X[:,1],X[:,2],c=color)

再来可视化一下,降维后的数据
plt.scatter(X_r[:,0],X_r[:,1],c=color)
plt.show()

可以看出来经过LLE变换后,样本数据在低维空间上已经明显区分出来。
拉普拉斯特征映射(LE)
拉普拉斯特征映射(Laplacian Eigenmaps,LE)解决问题的思路和LLE相似,是一种基于图的降维算法,使相互关联的点在降维后的空间中尽可能地靠近。
通过构建邻接矩阵为W的图来重构数据流形的局部结构特征,如果两个数据实例i和j很相似,那么i和j在降维后目标子空间中也应该接近。设数据实例的数目为n,目标子空间(即降维后的维度)为m,定义n×m大小的矩阵Y,其中每一个行向量yi是数据实例i在目标子空间中的向量表示。为了让样本i和j在降维后的子空间里尽量接近,优化的目标函数如下:

其中,‖yi−yj‖2为两个样本在目标子空间中的距离,wij是两个样本的权重值,权重值可以用图中样本间的连接数来度量。经过推导,将目标函数转化为以下形式:
Ly=λDy
其中L和D均为对称矩阵,由于目标函数是求最小值,所以通过求得m个最小非零特征值所对应的特征向量,即可达到降维的目的。
步骤
拉普拉斯特征映射的具体步骤如下。
① 构建无向图,将所有的样本以点连接成一个图,例如使用KNN算法,将每个点最近的k个点进行连接,其中k是一个预先设定的值。
②构建图的权值矩阵,通过点之间的关联程度来确定点与点之间的权重大小,例如,两个点之间如果相连接,则权重值为1,否则为0。
③ 特征映射,通过公式Ly=λDy计算拉普拉斯矩阵L的特征向量与特征值,用最小的m个非零特征值对应的特征向量作为降维的结果。
实现
se = manifold.SpectralEmbedding(n_components=2,n_neighbors=10)
Y = se.fit_transform(X)
Y.shape#(1500, 2)
参数的含义和上面的一样,Y是降到2维后的数据,可视化一下。
plt.scatter(Y[:,0],Y[:,1],c=color)
plt.show()























 9914
9914











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










