软件环境:vivado 2017.4 硬件平台:XC7Z020
说起FPGA端最常用、最直接的加密手段,大概就是利用每个芯片独有的ID号 + 加密算法了,所以今天这篇,就来简单提一下如何获取7系列的每个芯片的DNA号。至于为什么说是7系列,是为了与ultrascale的方法区别开,在ultrascale下,这个码是96 bit的,而在7系列及以前,是57 bit的。
第一种方法是利用JTAG。
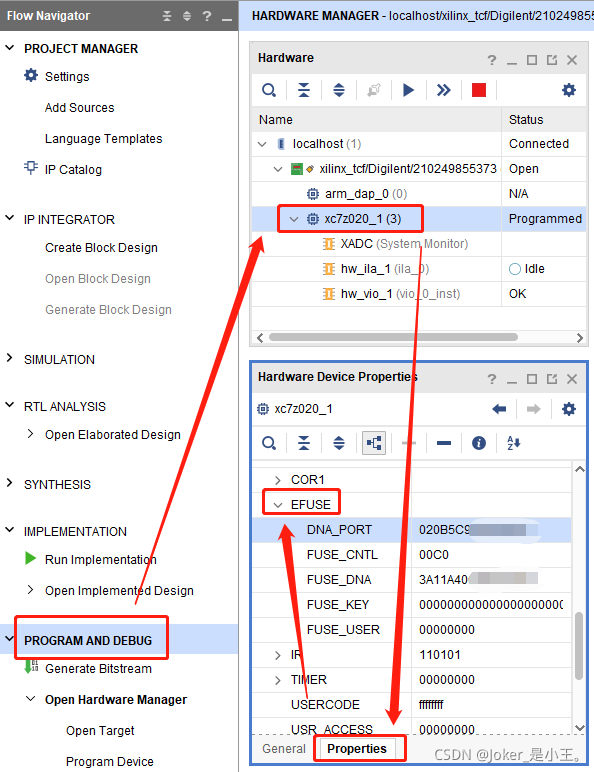
点击左侧的PROGRAM AND DEBUG后,利用JTAG连接板卡,在识别出的PL芯片型号上单击,再在下方点击Properties,属性中找到EFUSE,子项目中比较关心其中两个,一个是DNA_PORT,另一个是FUSE_DNA,接下来说下这两个的关系。
1.Fuse_DNA 是 7 系列和 Zynq 设备中的 64 位唯一标识符;
2.DNA_PORT 是一个 57 位值,最多可与 32 个设备共享;
3.DNA_PORT [0:56] 与 FUSE_DNA [63:7] 对齐,注意括号里MSB和LSB方向,举例如下。

接下来说一下怎么读,读取方法是利用DNA_PORT原语,参考手册UG470。

读取过程示意如下。

当READ = 1时,将FUSE 寄存器中的DNA值压入移位寄存器,而后将READ清0将SHIFT置1,DNA的值会依次从DOUT口移出,将DIN的值依次移入,最先从移位寄存器出来的是MSB,需要注意。
上代码。
module DNA_Port_read(
input sys_clk,
input sys_rst_n
//input dna_read_vio
);
wire dna_read_vio;
reg [1:0] dna_read_en;
reg [7:0] dna_cnt;
wire [56:0] dna_data;
wire dna_valid;
reg [56:0] dna_reg;
wire dna_dout;
wire dna_read;
wire dna_shift;
vio_0 vio_0_inst (
.clk(sys_clk), // input wire clk
.probe_out0(dna_read_vio) // output wire [0 : 0] probe_out0
);
DNA_PORT #(
.SIM_DNA_VALUE(57'h1234567AABBCCDD) // Specifies a sample 57-bit DNA value for simulation
)
DNA_PORT_inst (
.DOUT(dna_dout), // 1-bit output: DNA output data.
.CLK(sys_clk), // 1-bit input: Clock input.
.DIN(1'b0), // 1-bit input: User data input pin.
.READ(dna_read), // 1-bit input: Active high load DNA, active low read input.
.SHIFT(dna_shift) // 1-bit input: Active high shift enable input.
);
always@(posedge sys_clk)
begin
if(!sys_rst_n)
dna_read_en <= 2'd0;
else
dna_read_en <= {dna_read_en[0:0],dna_read_vio};
end
reg [7:0] dna_cnt_state;
always@(posedge sys_clk)
begin
if(!sys_rst_n)
begin
dna_cnt <= 8'd0;
dna_cnt_state <= 8'd0;
end
else
begin
case(dna_cnt_state)
8'd0:begin
if(dna_read_en == 2'b10)
dna_cnt_state <= 8'd1;
end
8'd1:begin
dna_cnt <= dna_cnt + 1'b1;
if(dna_cnt == 8'd99)
dna_cnt_state <= 8'd2;
end
8'd2:begin
dna_cnt <= 8'd0;
dna_cnt_state <= 8'd0;
end
endcase
end
end
always @ (posedge sys_clk)
begin
if(!sys_rst_n)
begin
dna_reg[56:0] <= 57'd0;
end
else
begin
if(dna_shift)
dna_reg[56:0] <= {dna_reg[55:0],dna_dout};
end
end
assign dna_read = (dna_cnt == 8'd1) ? 1'b1 : 1'b0;
assign dna_shift = ((dna_cnt >= 8'd10) && (dna_cnt <= (8'd10 + 8'd57 - 8'd1))) ? 1'b1 : 1'b0;
assign dna_data = dna_reg;
assign dna_valid = (dna_cnt == 8'd99) ? 1'b1 : 1'b0;
ila_0 ila_0
(
.clk(sys_clk),
.probe0(dna_data),
.probe1(dna_valid),
.probe2(dna_dout),
.probe3(dna_read),
.probe4(dna_shift),
.probe5(dna_reg),
.probe6(dna_cnt),
.probe7(dna_read_en)
);
endmodule
测试中使用vio来触发DNA_PORT的读取,再read信号置位后等待了10个clk,目的是确保DNA_PORT的值加载进了移位寄存器中(当然,这10个clk只是我测试用,不一定非要10个),而后shift拉高,将移位寄存器输出的MSB依次移位进dna_reg中,确保所有数据移出后,最后将valid拉个脉冲,整个读取过程结束。
上板测试结果如下。






 本文详细介绍了如何在FPGA 7系列(如XC7Z020)中通过JTAG接口获取芯片的DNA号。DNA号是每个芯片独特的57位标识符,可用于加密等安全应用。文中提供了使用DNA_PORT原语读取DNA号的VHDL代码示例,并解释了读取过程中DNA值如何从移位寄存器中移出。通过vio触发读取并利用ILA进行测试,确保数据正确读取。
本文详细介绍了如何在FPGA 7系列(如XC7Z020)中通过JTAG接口获取芯片的DNA号。DNA号是每个芯片独特的57位标识符,可用于加密等安全应用。文中提供了使用DNA_PORT原语读取DNA号的VHDL代码示例,并解释了读取过程中DNA值如何从移位寄存器中移出。通过vio触发读取并利用ILA进行测试,确保数据正确读取。
















 2164
2164

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








