antiSMASH 7.0:新的和改进的检测、调节、化学结构和可视化预测
antiSMASH 7.0: new and improved predictions for detection, regulation, chemical structures and visualisation

Article,2023-05-04
Nucleic Acids Research, [IF 19.16]
DOI:https://doi.org/10.1093/nar/gkad344
原文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad344/7151336
第一作者:Kai Blin
通讯作者:Kai Blin;Marnix H Medema; Tilmann Weber
主要单位:
丹麦科技大学诺和诺德基金会生物可持续性研究中心 (The Novo Nordisk Foundation Center for Biosustainability, Technical University of Denmark, Kgs. Lyngby, Denmark)
荷兰瓦赫宁根大学生物信息学组 (Bioinformatics Group, Wageningen University, Wageningen, The Netherlands)
- 摘 要 -
微生物产生小的生物活性化合物作为其次级或专性代谢的一部分。通常,此类代谢物具有抗菌、抗癌、抗真菌、抗病毒或其他生物活性,因此在医学和农业应用中发挥着重要作用。在过去十年中,基因组挖掘已成为一种广泛使用的方法,用于探索、获取和分析这些化合物可用的生物多样性。自 2011 年以来,“抗生素和次级代谢物分析 shell--antiSMASH”(https://antismash.secondarymetabolites.org/) 一直支持研究人员进行微生物基因组挖掘任务,既可以作为免费使用的 Web 服务器,也可以在 OSI 批准的开源许可证下作为独立工具。它是目前用于检测和表征古菌、细菌和真菌中生物合成基因簇 (BGC) 中使用最广泛的工具。在这里,我们展示了 antiSMASH 的更新版本 7。antiSMASH 7 中支持的簇类型的数量从 71 种增加到 81 种,并且在化学结构预测、酶合成路线可视化和基因簇调控等方面进行了改进。
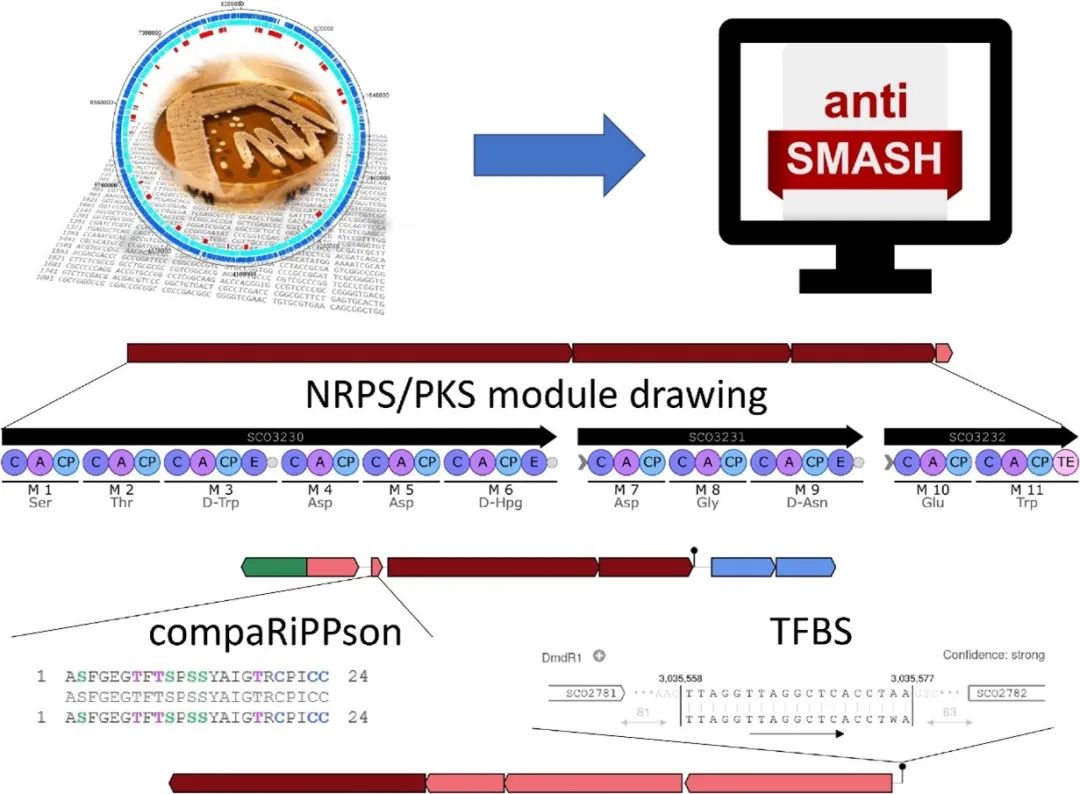
摘要图
- 引 言 -
微生物产生小的生物活性化合物构成了许多药物和作物保护剂的基础。传统上,新化合物是通过从天然来源中提取、化学分离、纯化和测试化合物的“查找和研磨”工作流程发现的。这种方法现在通常通过测序和随后的基因组和宏基因组数据挖掘来补充,以确定天然产物生物合成途径。用于“基因组挖掘”的软件工具,即在基因组中搜索二级/特殊代谢物(secondary/specialised metabolite, SM)生物合成基因簇(biosynthetic gene clusters, BGC),已经存在了十多年。
自 2011 年发布以来,antiSMASH已成为使用最广泛的SM BGC微生物基因组挖掘工具。围绕 antiSMASH,已经开发了一个包含或利用 antiSMASH 结果的独立工具生态系统,例如抗生素抗性目标搜索器 (ARTS 2) (http://arts.ziemertlab.com)、质谱引导肽挖掘工具 Pep2Path (http://pep2path.sourceforge.net/)、sgRNA 设计工具 CRISPY- web 2(https://crispy.secondarymetabolites.org/),BGC网络和集群平台 BiG-SCAPE,以及相关的大数据 BGC 集群工具 BiG-SLiCE(https://github.com/medema-lab/bigslice)。反过来,antiSMASH 还可以通过使用 antiSMASH 6 中引入的侧边加载机制,合并和显示来自其他工具(例如 DeepBGC (https://github.com/Merck/deepbgc))的BGC预测。antiSMASH BGC 预测包含在许多基因组和面向 BGC 的数据库中,例如联合基因组研究所的集成微生物基因组数据库及其生物合成基因簇IMG-ABC图谱、用于微生物基因组注释和分析的MicroScope平台、手动管理的BGC的MIBiG数据库、BGC家族数据库BiG-F AM和antiSMASH数据库。
antiSMASH使用基于规则的方法来识别 SM 生产中涉及的许多不同类型的生物合成途径,对于编码非核糖体肽合成酶 (NRPS)、I 型和 II 型聚酮合酶 (PKS) 以及核糖体合成和翻译后修饰肽 (RiPP) 类别的镧氏肽、套索肽、活性肽和硫肽而言,特定簇分析可以提供有关所执行的生物合成步骤的更多信息,因此也可以提供对所产生化合物的更详细的预测。
在这里,我们展示了 antiSMASH 的第 7 版。它通过添加和更新 BGC 检测规则、通过预测 LogoMotif 数据库 (https://logomotif.bioinformatics.nl/) 中表示的转录因子结合位点来增强调节功能检测,并为NRPS和PKS簇,PFAM和TIGRFAM结构域比对添加新的可视化,以及列出具有动态搜索和过滤功能的区域中所有基因的表格。
- 新功能和更新 -
① 新簇的类型和动态检测配置文件
antiSMASH 使用人工策划和验证的“规则”来定义基因组区域中需要存在哪些核心生物合成功能才能构成 BGC。为了识别这些生物合成功能,antiSMASH 使用来自 PFAM、TIGRFAMs、SMART、BAGEL、Yadav和自定义模型的隐马尔可夫模型 (pHMM)。antiSMASH 6 包含 71 种BGC类型的规则。在 antiSMASH 7 中,这个数字增加到 81,增加了对 2-脱氧链霉胺氨基糖苷、氨基多羧酸金属素、含精氨酸的环二肽 (RCDP)、crocagins、甲烷杆菌素、真菌孢子素、NRP-金属载体、片状金属载体和类真菌-RiPP。NRP金属载体BGC以前一般由NRPS规则检测,但现在根据编码参与金属螯合的官能团生物合成的基因被特异性识别。磷酸盐规则已更新,旧规则保留在“类似磷酸盐”的名称下。除了改进的磷酸烯醇丙酮酸 (PEP) 变位酶检测模型外,还利用支持模型(补充表 S1-S2)来减少误报并改进聚类边界的划定(补充图 S1)。
由于并非 BGC 的所有功能都可以使用 pHMM 捕获,因此 antiSMASH 7 添加了创建由 Python 代码定义的动态配置文件的选项。目前正用于检测基于M.KKN[IL]的氰基蛋白前体。P….PV.R中保守序列基序太小而无法在 pHMM 中可靠地拾取。
② NRPS和PKS改进
为了改进真菌基因簇中的PKS注释,我们增加了肉碱-AT(CAT)、产物模板(PT)和硫代半胱氨酸/β消除裂解酶(SH)结构域的检测。细菌反式酰基转移酶聚酮合成酶(TRAN-AT PKS)的酮合成酶(KS)结构域现在也使用Transat等的亚型特异性PHMMS进行注释。PKS KS结构域和NRPS缩合(C)结构域可以提交给最近发布的自然产物结构域搜索者(NapDoS2)版本2进行系统发育分析。最近的MIBiG3版本增加了超过2000个NRPS腺化(A)和相关结构域的底物特异性。为了让我们的用户从更多的信息中受益,我们已经用新的“NRPYS”库(https://github.com/kblin/nrpys/))取代了我们自2011年以来一直提供的NRPSPredictor2A域基板预测工具,该库允许我们将Stchehaus代码查找表从以前的554个条目更新到现在的2319个条目。由于Stchehaus使用的10个氨基酸(AA)编码在新的数据集中并不总是解析到单个底物预测,很可能是由于所涉及的A结构域的底物混杂,NRPYS报告了所有相同质量的10个AA编码比对,按照Rausch等人在NRPS Predictor 1中使用的描述,按照预测在A域活性部位周围8˚A半径内的34个AA的最高匹配进行排序。为了完全替代,NRPyS仍然运行NRPSPredictor 2中的原始支持向量机(SVM)模型。
③ RiPP前体比较
为了帮助用户评估RIPP前体多肽的新颖性,我们开发了CompaRiPPson分析,将已识别的RIPP前体的(预测)核心多肽与antiSMASH-DB和MIBiG 3.1数据库中的RIPP前体进行比较。这些数据库的比对分别提供,反SMASH-DB比对包含一个更大的数据集,在3.0版中包含10583个预测前体,而不是来自MIBiG的28个经过实验验证和注释的前体。前体比对由抗SMASH-DB的前体基因位点标签标记,MIBiG由化合物名称标记。按序列身份排序,具有相同前体序列的数据库比对被分组在一起。查询和比对以对齐方式显示(图 1A)
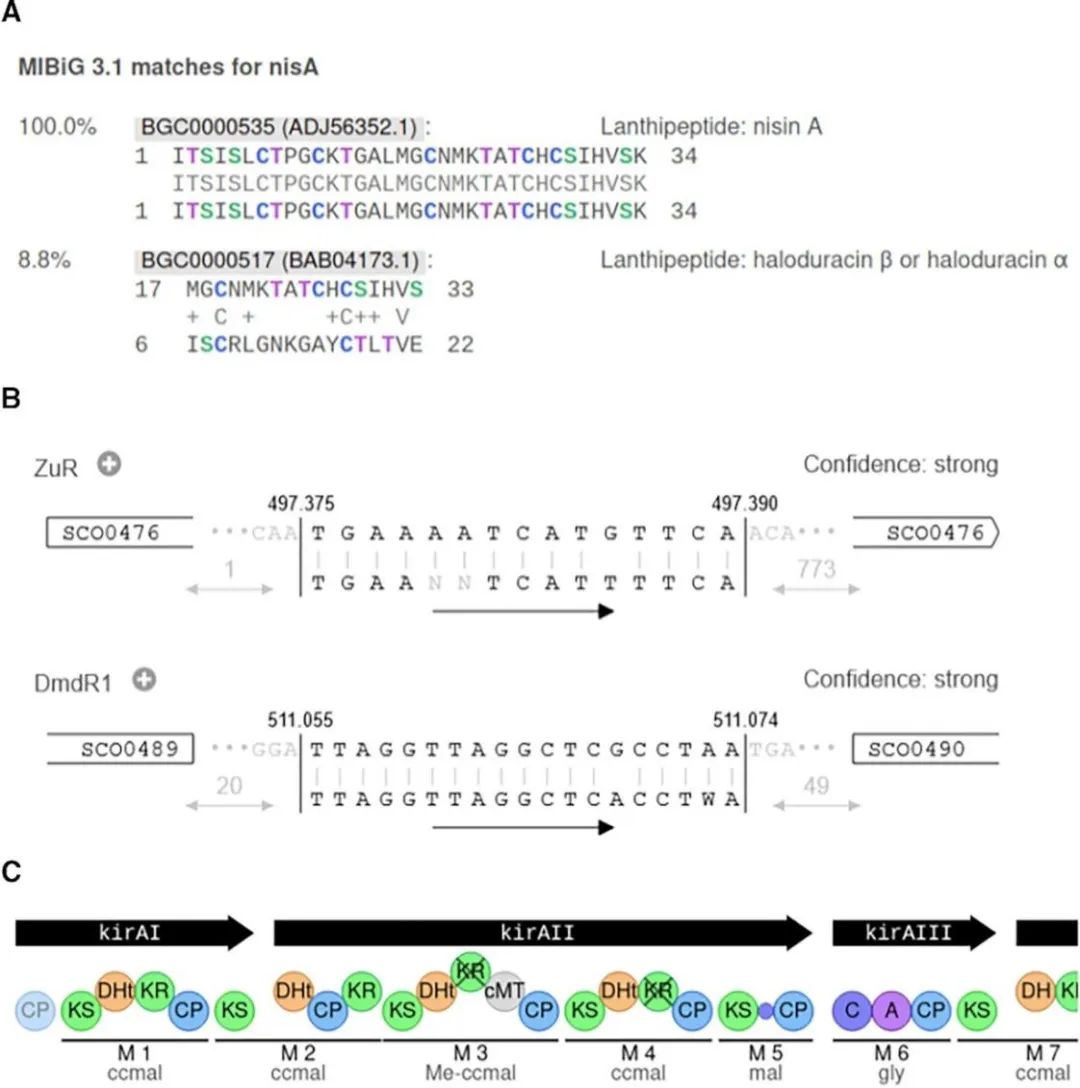
图1 新的antiSMASH可视化示例。
(A) 图1显示了lanthipeptide I类乳链菌肽A输入序列的CompaRiPPSon-MIBiG匹配,自我比对的匹配率为100%,与另一种lanthipeptide的匹配率低得多(8.8%)。(B) 显示了天蓝色链霉菌A3上的两个高可信度TFBS finder比对。第一个比对,推定的ZurR结合位点,位于基因SCO0476的起始位置,ATG起始密码子的最后两个碱基是结合位点的前两个碱基。DmdR1比对位于SCO0489和SCO0490之间和上游。(C) 图1显示了在PKS/NRPS基罗霉素基因簇(MIBiG ID:BGC0001070)上的Streptomyces colinus Tü365杂合反式的第一个模块。
④ 转录因子结合位点预测
LogoMotif数据库(https://logomotif.bioinformatics.nl/)包含一系列经过实验验证的转录因子结合位点(TFBS)图谱和相应的位置权重矩阵(PWM),重点关注放线菌。antiSMASH TFBS 查找器模块使用这些 PWM 来注释假定的 TFBS。根据比对分数,TFBS 查找器分别显示强、中或弱的置信度。结合位点显示在其基因组环境中,指示与周围基因的方向和距离(图1B)。所有比对结果都链接到LogoMotif网站,以获取有关特定配置文件的更深入信息。
⑤ 基因表
现在,每个区域都在一个可过滤的互动表中列出了所有包含的基因特征。可以通过在搜索框中输入搜索词来过滤基因(支持纯文本和正则表达式)。与过滤器匹配的基因将显示在区域视图中,如果启用,该视图将自动缩放到所选内容。用于过滤的信息目前包括基因的名称、其生物合成类型和基因功能注释(例如smCOG点击)。
⑥ 更新了可视化和其他优化
RPS和PKS簇的新可视化以传统的出版风格以预测的组装顺序绘制酶结构域和模块,这使研究人员可以使用antiSMASH矢量图形作为其出版质量图的起点(图1C)。一个区域中的PFAM和TIGRFAMs域点击现在以与现有NRPS / PKS域可视化类似的方式显示。
在MIBiG 3.1版本之后,KnownClusterBlast和ClusterCompare数据库进行了更新。
- 结论和未来展望 -
利用antiSMASH等工具对天然产物BGC进行基因组挖掘是现代天然产物发现工作流程的基础。随着本文中的添加和更新,antiSMASH正在不断更新,以保持微生物天然产品基因组挖掘的首选解决方案。开源的antiSMASH软件继续为自然产品领域蓬勃发展的计算工具生态系统做出贡献。除了直接提供微生物天然产物预测外,antiSMASH还作为其他工具的技术平台,如植物天然产物预测工具PlantiSMASH、初级代谢基因簇预测工具gutSMASH以及其他目前正在开发的工具。在未来的更新中,我们将继续改进化合物结构和亚组分预测的工作,为不同的分类群添加额外的TFBS图谱(例如来自Jaspar的真菌图谱),以及与生态系统中的其他工具集成。我们还开始提供一个网站,在https://experimentalsmash.secondarymetabolites.org/ 上试用未来可能的antiSMASH功能。
参考文献
Kai Blin, Simon Shaw, Hannah E Augustijn, Zachary L Reitz, Friederike Biermann, Mohammad Alanjary, Artem Fetter, Barbara R Terlouw, William W Metcalf, Eric J N Helfrich, Gilles P van Wezel, Marnix H Medema, Tilmann Weber, antiSMASH 7.0: new and improved predictions for detection, regulation, chemical structures and visualisation, Nucleic Acids Research, 2023;, gkad344, https://doi.org/10.1093/nar/gkad344
- 第一作者兼通讯作者 -

丹麦科技大学诺和诺德基金会生物可持续性研究中心
Kai Blin
第一作者兼通讯作者:Kai Blin,是天然产物基因组挖掘组计算生物学团队的负责人。该团队正在从事antiSMASH基因组挖掘软件的开发以及其他工具和数据库的周边生态系统。我们正在进行的研究重点是改进我们可以从基因组挖掘结果中获得的预测,包括结构、生物活性和调控。同时也致力于使用机器学习工具来应对这些挑战。
- 通讯作者 -

荷兰瓦赫宁根大学
Marnix Medema
教授
Marnix Medema,荷兰瓦赫宁根大学生物信息学教授。Medema小组开发和应用算法,用于微生物生物合成途径及其产物的宏基因组鉴定和功能预测,旨在揭示微生物组的化学语言并加速药物发现。该小组还参与开发用于鉴定生物合成基因簇的antiSMASH软件,并开发了一系列其他工具和数据库,以描述其多样性并确定其在微生物组中的功能作用(例如,MIBiG,BiG-SCAPE,BiG-SLiCE,BiG-MAP,PIKAChU等)。

丹麦技术大学诺和诺德基金会生物可持续性研究中心
Tilmann Weber
教授
Tilmann Weber,丹麦技术大学诺和诺德基金会生物可持续性中心教授,也是天然产物基因组挖掘小组副主任。他的主要研究兴趣集中在破译分子途径和通过结合遗传,生化和生物信息学方法对天然产物的生物合成进行工程设计。他是开发用于自动基因组挖掘(CLUSEAN,antiSMASH,antiSMASH-DB)和次级代谢物生物合成途径分析软件的先驱。他的团队能够首先阐明elfamycin家族抗生素的生物合成途径,并深入参与开发基于CRISPR的放线菌代谢工程工具。
往期精品(点击图片直达文字对应教程)










 antiSMASH7.0更新了BGC检测规则、增强转录因子检测、改进了NRPS和PKS预测,并提供了更丰富的可视化功能,以提升微生物代谢物的预测和分析能力。
antiSMASH7.0更新了BGC检测规则、增强转录因子检测、改进了NRPS和PKS预测,并提供了更丰富的可视化功能,以提升微生物代谢物的预测和分析能力。














































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








