蛋白质是一切生命活动的执行者,由20种天然存在的蛋白质氨基酸构成。与高歌猛进的DNA测序技术相比,高灵敏、高通量的蛋白质测序技术的发展严重滞后,目前仅Edman降解法和质谱法能做到蛋白质测序 [1,2]。然而,Edman降解法的序列分析长度有限,质谱法的检测限和动态范围阻碍了其对于低丰度蛋白质的分析鉴定和新型生物标志物的发现。单分子蛋白质测序技术可以提供更高的灵敏度和准确度,有望助力单细胞蛋白质组学的发展。2023年,Nature 杂志将单分子蛋白质测序技术评为未来一年对科技创新产生巨大影响的七项新技术之一 [3]。
纳米孔是一种超灵敏、无标记的单分子分析技术,已成为新一代的DNA测序工具 [4]。然而,目前缺乏可以同时分辨所有20种蛋白质氨基酸及其翻译后修饰的纳米孔传感器,纳米孔蛋白质测序技术也尚未真正实现。与核酸相比,蛋白质具有更复杂的高级结构和更丰富的化学多样性,且电荷分布不均匀,很难以可控的方式实现蛋白质在纳米孔中的单向易位。此外,相比于核酸的4种核苷酸基本单元,蛋白质由20种氨基酸组成。无论采取哪种读出方式,检测20个可区分的信号本身就是一个很大的挑战,准确解读蛋白质上的翻译后修饰信息更是对分析方法的检测分辨率提出了更高的要求。
近日,南京大学化学化工学院黄硕教授课题组构建了一种工程化的耻垢分枝杆菌膜蛋白A (MspA) 纳米孔,实现了全部蛋白质氨基酸及它们的翻译后修饰检测。研究团队在MspA纳米孔的收缩区域引入了一个镍离子-次氮基三乙酸(Ni-NTA)适配体,利用氨基酸和金属镍离子的配位相互作用,实现了全部蛋白质氨基酸的直接检测和区分,准确率高达98.8%,是世界上第一个能完全分辨所有20种蛋白质氨基酸的纳米孔。在本篇工作中,作者亦采用了同种孔道,进而同时区分了4种常见的翻译后修饰氨基酸。
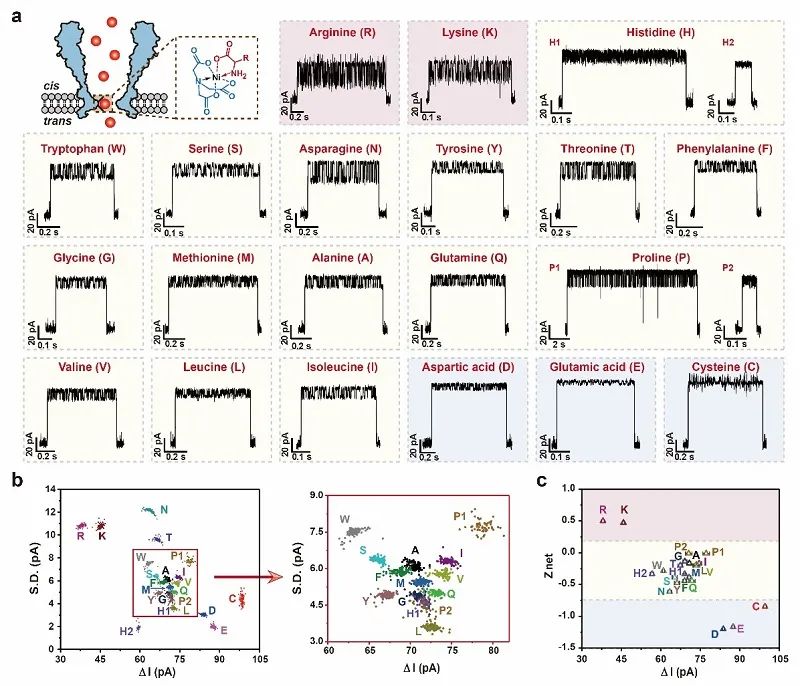
图1. 基于Ni-NTA修饰的MspA纳米孔对20种氨基酸的检测
不同于传统的纳米孔传感策略所依赖的尺寸排阻效应,该纳米孔传感器对于氨基酸本身所带电荷更为敏感。在该体系中,负电荷更多的氨基酸普遍具有更大的阻塞电流幅度,而正电荷氨基酸的阻塞电流幅度偏小,这对于体积或分子质量差异小的氨基酸分辨是非常有利的。此外,作者也采用分子动力学模拟探究了氨基酸传感过程中孔道内部的分子运动。模拟和实验共同证明,孔道上修饰的镍离子对于信号的产生具有至关重要的作用,这种独特的传感模式结合具有高分辨率的MspA纳米孔,共同产生了具有高度特征性和一致性的氨基酸电流信号,为多种氨基酸的同时分辨打下了坚实的基础。
随后,研究团队将该策略用于多肽的氨基酸组成分析。研究团队使用天然存在的三肽GHK以及合成的八肽TLEIYNRF作为模型肽,用具有广泛底物相容性的亮氨酸氨肽酶将不同的靶标肽消化为单个的氨基酸,然后利用所开发的纳米孔传感器检测消化产物,获得了相应的离子电流事件。机器学习被用来自动识别氨基酸的身份,最终重构出的氨基酸图谱与原始肽序列完全一致,证明了该策略用于识别蛋白裂解氨基酸的能力。
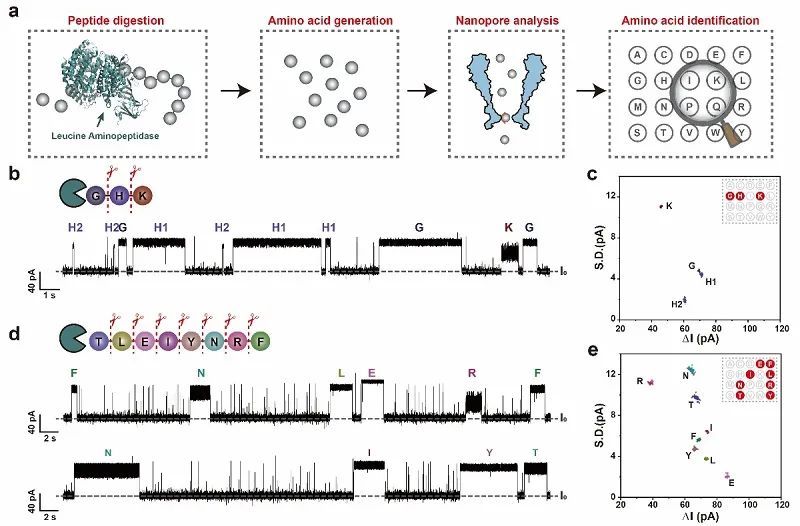
图2. 基于纳米孔策略的肽的氨基酸组成鉴定
该纳米孔传感器还可以在分辨20种氨基酸的同时直接分辨翻译后修饰(PTMs)。PTMs是蛋白质在翻译后的化学修饰,在多种蛋白质功能的调节中具有重要作用。准确鉴定PTMs对于理解细胞功能和相应的生理和病理过程至关重要。研究团队利用所构建的纳米孔传感器成功检测了四种经典的修饰氨基酸:双甲基精氨酸 (Me-R)、O-乙酰苏氨酸 (Ac-T)、2-乙酰基-1-beta-(L-天冬酰胺)-1,2-双脱氧-D-葡萄糖 (GlcNAc-N) 和O-磷酸丝氨酸 (P-S),它们分别是甲基化、乙酰化、糖基化和磷酸化修饰的代表。这四种修饰氨基酸可以与20种蛋白质氨基酸完全分辨,24种氨基酸的机器学习区分准确率为98.6%,对纳米孔蛋白质测序和翻译后修饰的检测具有里程碑意义。
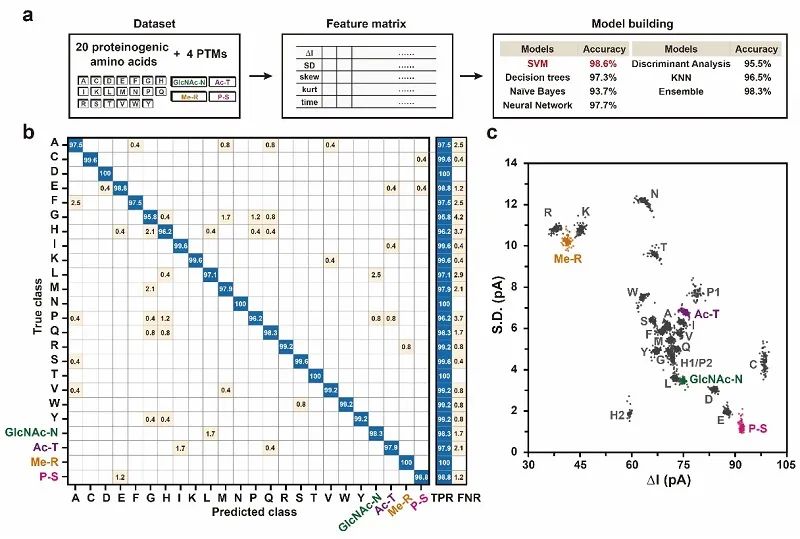
图3. 机器学习辅助20种天然氨基酸及4种修饰氨基酸的识别
小结
本研究构建了具有唯一金属离子修饰位点的工程化MspA纳米孔道,实现了全部20种氨基酸和4种代表性翻译后修饰氨基酸的直接检测和完全区分。该传感策略具有卓越的稳定性和鲁棒性,可以维持长时测量和耐受复杂的检测环境。为了实现事件的自动化分拣和避免人为判断误差,研究团队引入了机器学习算法来进行氨基酸鉴定。其中,二次支持向量机(SVM)模型对于24种氨基酸的区分准确率为98.6%。该策略也被用于分析复合氨基酸营养片中的氨基酸成分,证明该技术在临床诊断和营养学分析中具有应用潜力。另外,该纳米孔传感器可以准确鉴定由肽酶切割肽链产生的氨基酸,为实现基于纳米孔的单分子蛋白质测序提供了坚实的分辨率基础。
相关论文该工作近日在Nature Methods 杂志发表。南京大学化学化工学院黄硕教授为该论文唯一通讯作者,南京大学博士生王可凡、张善雨和周骁为该论文共同第一作者。
原文(扫描或长按二维码,识别后直达原文页面):

Unambiguous discrimination of all 20 proteinogenic amino acids and their modifications by nanopore
Kefan Wang, Shanyu Zhang, Xiao Zhou, Xian Yang, Xinyue Li, Yuqin Wang, Pingping Fan, Yunqi Xiao, Wen Sun, Panke Zhang, Wenfei Li & Shuo Huang*
Nat. Methods, 2023, DOI: 10.1038/s41592-023-02021-8
参考文献:
1. Alfaro, J. A. et al. The emerging landscape of single-molecule protein sequencing technologies. Nat Methods 18, 604-617 (2021).
2. Restrepo-Perez, L., Joo, C. & Dekker, C. Paving the way to single-molecule protein sequencing. Nat Nanotechnol 13, 786-796 (2018).
3. Eisenstein, M. Seven technologies to watch in 2023. Nature 613, 794-797 (2023).
4. Deamer, D., Akeson, M. & Branton, D. Three decades of nanopore sequencing. Nat Biotechnol 34, 518-524 (2016).
导师介绍
黄硕
https://www.x-mol.com/university/faculty/56491
课题组网站:
http://hysz.nju.edu.cn/bionano
往期精品(点击图片直达文字对应教程)























































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








