植物合成生物学正逐渐在农业和医药领域崭露头角,有望为农业生产、人类医疗保健和环境可持续性等重要问题提供解决方案。以植物为底盘的合成开关及环路研究、合成代谢工程等不断涌现,相较于单细胞的微生物底盘系统,以植物为底盘的合成生物学研究仍处于初级阶段。尽管组学研究的蓬勃发展为这一领域提供了诸多基础,但如何从笼统的定性描述进入可数学描述的定量可控,是目前植物合成生物学研究急需跨越的阶段。
近日,来自中国科学院分子植物科学卓越创新中心的王勇教授团队在Quantitative Biology上发表题为“From qualitative to quantitative: the state of the art and challenges for plant synthetic biology”的综述文章(点击文末“阅读原文”下载PDF全文)。文章回顾了当前植物合成生物学研究在元件和环路的标准化和定量表征方等方面所取得的进展,并提出了在植物合成生物学“设计-构建-测试-学习”的迭代循环中,定量化研究的匮乏,为植物合成生物学研究中新生物特性的设计所带来的挑战。

全文概要
目前在植物系统中已经成功生产了很多高附加值的代谢产物和功能蛋白,NAND、OR等复杂的逻辑门环路更是为以前不能实现的复杂调控提供了新的可能性。然而,目前植物合成生物学的研究主要集中在定性的功能实现上,对于定量描述及精细控制方面远远不足。在实现合成系统的过程中,每个成分的剂量特性都会对系统整体的性能产生影响。因此,对于植物底盘中定量调控的深入研究显得至关重要。
随着组学数据的急剧增长和生物信息技术的快速发展,已经发展出了很多功能基因描述性数据库。遗憾的是,这些植物数据库中的功能序列要成为合成生物学的元件工具还缺乏精细化表征,尤其缺乏标准化和定量表征。这也导致了植物底盘中难以理性设计和构建可定量预测的合成系统。针对上述问题,本文概述了目前在元件的标准化、定量表征和定量调控环路方面的已有的一些研究进展。
元件的标准化
标准化的元件有助于高效的设计和组装多功能的遗传环路。在植物中使用更多的是一些能够同时组装多个DNA片段的方法,例如基于attL/attB位点特异性重组的Multisite Gateway和基于IIS型限制性内切酶的Golden Gate等。英国生物技术和生物科学研究委员会(Biotechnology and Biological Sciences Research Council,BBSRC)也针对Golden Gate组装方法定义了植物合成生物学的通用标准语法。
元件的定量表征
通过粒子轰击、农杆菌侵染和原生质体转化等方法,可以在植物中快速表征元件或环路的特性。目前在利马豆子叶、本氏烟草叶片、BY2悬浮细胞、拟南芥原生质体、高粱原生质体这些植物底盘中都已经有对调控元件进行规模化的定量表征的研究工作(图1)。
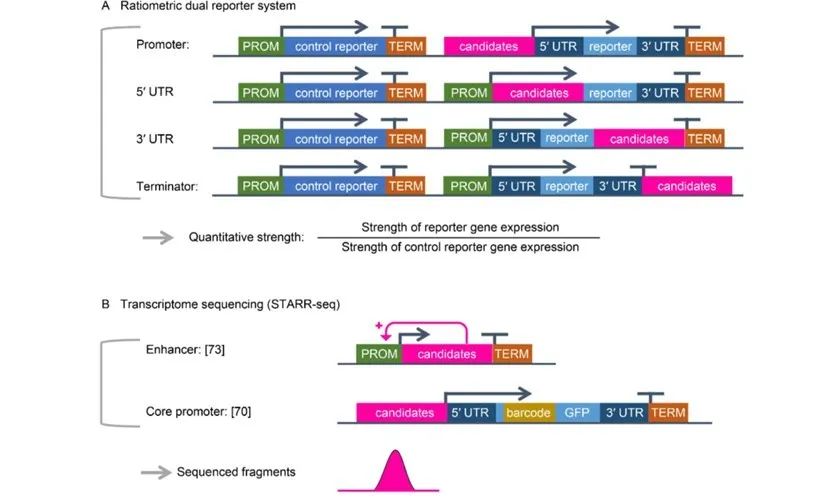
图1. 不同遗传元件的定量表征方法
计算机工具更是可以根据基因元件的序列结构和定量特性进行加工和建模,并通过理性设计构建新的强度可预测的元件工具(图2)。机器学习算法,如支持向量机(SVM)或神经网络,极大地促进了生物元件的定性/定量预测和新元件的设计。但是这些研究目前只局限在特定的几种植物底盘中,且元件的数量和类型有限。
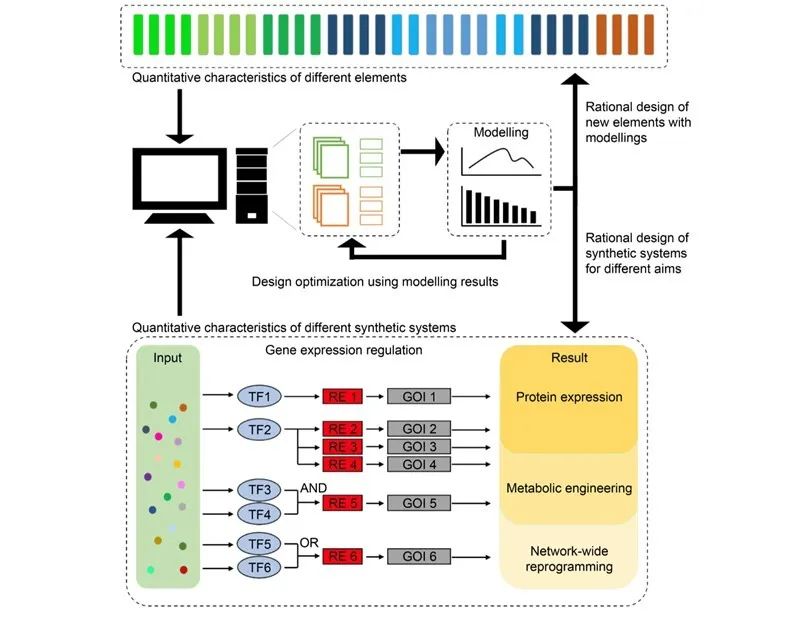
图2. 通过计算机工具进行合成系统的合理设计
定量调控环路
多基因调控是植物合成生物学不可避免的趋势。然而在植物底盘中引入多个基因时,实际应用到的调控元件数量是有限的。因此,开发具有定量特性的新元件和可量化预测的合成系统具有重要意义。基于CRISPR的合成系统由于其高准确性和同时调控多基因的能力而备受关注,也为定量调控的合成环路设计提供了新的策略。CRISPR-Cas9被用于编辑CLE启动子的碱基序列来控制基因的表达剂量,从而实现表型的梯度变化。RNA支架在酵母和人源细胞中已经被证明可以同时实现对基因的激活和抑制,但是在植物底盘中还没有报道过这种多层次的定量调控。
在此基础上,文章提出了植物合成生物学从定性描述发展为可数学描述的定量可控的研究中所面临的几个重要挑战。
1. 基因元件缺乏定量描述
在构建功能可预测的合成系统时,除了元件的功能特性之外,对元件以及元件之间相互作用的定量理解也至关重要。具有定量参数的基因元件已被广泛应用于微生物的基因重编程,包括启动子强度、转录速率、翻译速率、抑制因子/激活因子数量、结合亲和力和蛋白降解速率等。Cello等软件工具可以辅助人工遗传环路在大肠杆菌和酿酒酵母底盘中的理性设计,其中的核心正是描述基因表达的数学模型(图2)。
2. 基因元件正交性不足
正交性指的是元件能够独立于底盘细胞和其他基因元件而发挥功能。许多元件在不同的植物底盘细胞中功能表现不一致,缺乏足够的正交性。元件重构可以通过简化内部成分,去除导致低效的序列和受内源调控的序列部分,同时仍保留元件的生物学功能。此外,位置效应和周围序列的干扰也可能导致强度变化,绝缘子可以通过调节染色质结构缓解这种效应。如何构建所需的合成系统并实现系统与宿主底盘之间的完全正交仍需进一步研究。
3. 构建能力受限
植物底盘中已经发展了一些标准化的基因组装方法。但无切割痕迹的大片段基因组装技术还有待发展,目前还没有植物中合成染色体的研究报告。
4. 缺乏预测遗传环路的基因编译器
生物过程可以被描述为复杂的信号通路网络,并被转化为计算机模拟的数学模型。基因编译器利用准确的数学模型使得计算机模拟、预测和控制生物过程成为可能。支原体和大肠杆菌的全细胞模型已经被报道。Virtual Physiological Rat (VPR)也在可预测医学方面取得了显著进展。但是,植物细胞中的定量表征研究远落后于这些底盘,严重缺乏能够用于定量描述调控网络的多尺度模型。
综上所述,植物合成生物学的定量化研究还处于起步阶段。对此,作者认为微生物底盘中的一些出色的合成设计值得借鉴和学习。考虑到植物多细胞、多组织的特性,在设计和构建环路中更需要各种具有定量描述的元件工具。此外,如何准确地定量描述不同类型细胞中的代谢差异和多细胞群体之间的互作关系也都是值得考虑的问题。
植物合成生物学有望成为解决农业、医药、能源等各领域内问题的重要途径。但在其应用和商业化的过程中,还需要定量化的工程原理,以保证一些更实用、具有鲁棒性的合成系统。目前植物定量合成生物学研究所面临的主要挑战在于对元件的定量认知不足、工具的兼容性低以及植物细胞底盘定量描述的落后。希望这些挑战能在进一步的研究中受到关注。

QB期刊介绍
Quantitative Biology (QB)期刊是由清华大学、北京大学、高等教育出版社联合创办的全英文学术期刊。由高等教育出版社和Wiley双平台出版和发行。QB主要刊登生物信息学、计算生物学、系统生物学、理论生物学和合成生物学的最新研究成果和前沿进展,并为生命科学与计算机、数学、物理等交叉研究领域打造一个学术水平高、可读性强、具有全球影响力的交叉学科期刊品牌。
QB期刊目前已被ESCI, Scopus, CSCD等国内外重要数据库收录。IF2022=3.1,Citescore2022=5.7。
往期精品(点击图片直达文字对应教程)























































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








