题目:Large language models generate functional protein sequences across diverse families
发表年份:2023
期刊:Nature biotechnology
影响因子:46.9
作者:Ali Madani, Ben Krause, Rric R. Greene等
作者单位:加州大学生物工程与治疗科学系

摘 要

利用人工智能的方法生成人工蛋白质序列可以为生物医学和环境挑战提供突破性的解决方案。本文训练的基于深度学习的语言模型—ProGen,可以在大型蛋白质家族中生成功能性的人工蛋白质序列。该蛋白质语言模型通过简单的学习,预测来自数千个蛋白质家族的2.8亿个蛋白质序列的下一个氨基酸,而无需明确的生物物理建模。将模型生成的人工蛋白根据五种不同的抗菌溶菌酶家族微调后进行实验评估。人工蛋白的活性和催化效率与典型的天然溶菌酶相似,但与任何已知的自然进化蛋白的同源性低至31.4%。具有酶活性的人工蛋白的X射线晶体结构概括了天然蛋白中活性位点残基的保守折叠和定位。而且,通过准确预测人工分支酸变位酶和苹果酸脱氢酶蛋白的功能,展示了该语言模型适应不同蛋白质家族的能力。

主要内容

1、ProGen的功能以及工作原理
ProGen是一个基于数百万原始蛋白质序列训练的蛋白质语言模型,可以生成跨多个家族和功能的人工蛋白质。经过训练,ProGen可以从零开始生成任何蛋白质家族的全长蛋白质序列,与天然蛋白质有不同程度的相似度。在通常情况下,当一个蛋白质家族的一些序列数据可用时,可以利用预先训练好的语言模型与家族特异性序列进行微调的技术,进一步提高ProGen捕捉蛋白质家族对应的局部序列邻域分布的能力。
ProGen是基于transformer的神经网络架构来构建的(图1e),它以从左到右的方式生成序列,逐个口令,其中下一个口令以所有先前生成的口令为条件。从UniParc、UniprotKB、Pfam和NCBI收集了2.8亿条蛋白质序列构建数据集用于训练(图1d)。经过训练后,ProGen可以通过指定一个控制标签(例如来自Pfam的蛋白质家族标识符)从头开始生成新的蛋白质序列(图1c)。ProGen的一个关键组件是条件生成,即由作为语言模型输入的属性标签控制的序列生成(图1a)。对于蛋白质数据,控制标签是诸如蛋白质家族、生物过程和分子功能等属性,这些属性可用于公共蛋白质数据库中的大部分序列(图1b)。通过在序列的开始指定一个或多个控制标签,可以显着限制序列空间的生成,提高生成质量。
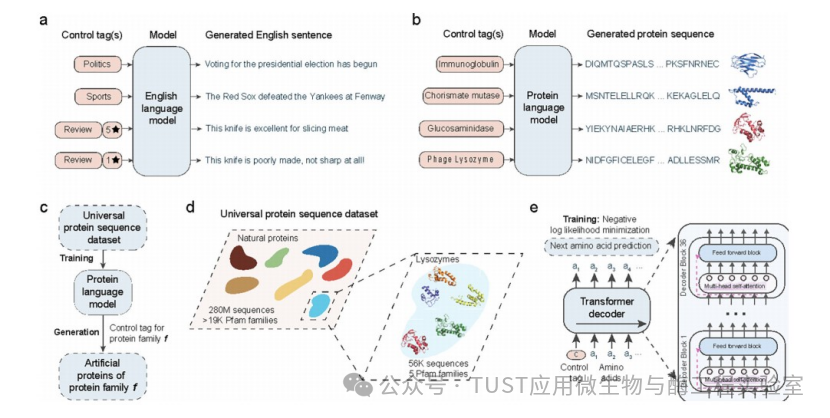
图1 基于条件语言建模的人工蛋白质生成
2、ProGen生成的序列
首先从噬菌体溶菌酶、农药酶、葡萄糖苷酶、糖苷水解酶和转糖基化酶这五个溶酶菌家族中收集了包括55948个序列的数据集,以获得阳性对照并对ProGen进行微调。经过微调后,通过提供每个家族的Pfam ID作为控制标签,使用ProGen生成了100万个人工序列。生成的人工序列涵盖了五个溶酶菌家族(图2a),包含不同的蛋白质折叠,活性位点结构和酶促机制。尽管人工序列可能从序列同一性计算上与自然序列存在差异(图2b),但在每个家族中形成天然蛋白和人工蛋白的独立多序列比对时,它们表现出相似的残基位置熵(图2c)。这表明该模型已经捕获了进化守恒模式,而没有对显式对齐信息进行训练。
为了通过实验评估ProGen在与天然蛋白质的一系列序列差异中的性能,作者选择了100个序列,这些序列根据自然序列的生成质量和多样性进行过滤,作为包含2.8亿个蛋白质的训练数据集中任何蛋白质的“max ID”进行测量。全长基因被合成并通过无细胞蛋白合成和亲和层析纯化。阳性对照组(100个天然蛋白)中,通过色谱峰和带可视化分析,72%的蛋白表达良好。ProGen生成的蛋白与任何已知的天然蛋白(max ID为40-90%)在序列一致性的所有箱中表达同样好(72/100),图2e。
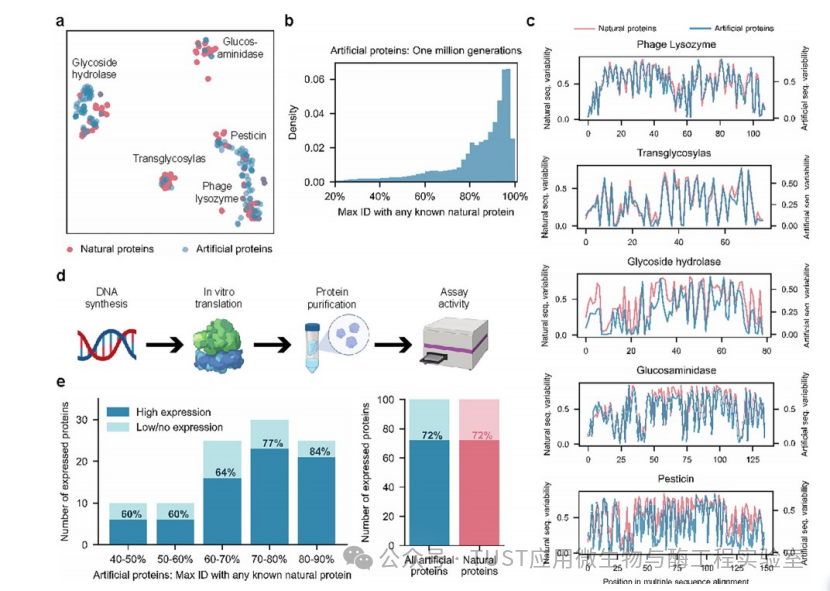
图2 实验生成的人工抗菌蛋白种类丰富且表达良好
3、ProGen的生成质量
根据荧光素标记的溶球微球菌细胞壁的猝灭释放检测活性,从每个表达的100个集合中随机选择90个蛋白质。以96孔板的形式制备蛋白质,提取随时间变化的荧光曲线,图3a。以自然进化的范例蛋白——蛋清溶菌酶(HEWL)为阳性对照,以泛素为阴性对照。
在模型生成的人造蛋白中,73%(66/90)是功能性的,并且在家族中表现出高水平的功能性(图3c)。具有代表性的天然蛋白具有相似的功能水平,59%(53/90)的总蛋白被认为具有功能。而且人造蛋白与天然蛋白的活性水平相匹配,即使在较低的序列水平上与任何已知的天然蛋白也相同(图3b)。在天然和人工蛋白质中,少量蛋白质在一个数量级内,其活性明显高于所有阴性对照。这些高度活跃的异常值显示了ProGen产生序列的潜力。
在100个人工蛋白中,L056(max ID为69.6%)和L070(max ID为89.2%)两种蛋白对大肠杆菌BL21菌株在16℃过夜诱导时表达良好,并具有杀菌活性。通过测量纯化的表观低分子量物种的KM值,其中L056和L070都具有高活性,并且具有与HEWL相当的Michaelis-Menten参数(图3d)。总的来说,L056和L070具有与HEWL相当的催化活性和杀菌能力。而且,L056和L070从它们各自最接近的同源序列中分离出来的突变在位置或结构元素上没有偏倚。总之,这些结果表明ProGen可以产生具有接近天然活性的人工蛋白质。
接下来,检查了人工蛋白质的结构差异。作者测定了L056的2.5 Å分辨率晶体(图3e)。整体折叠与预测相似,trRosetta预测的主链结构的Cα RMSD为2.9 Å, WT T4溶菌酶结构的Cα RMSD为2.3 Å。最大的结构分歧发生在包含残基18-31的β发夹中。该区域形成底物结合间隙的底部,并且是对底物结合很重要的铰链结合运动的一部分。
为了检验ProGen是否能产生具有“模糊区”(twilight zone)功能的蛋白,即两个蛋白在功能上不具有相似性,作者为两个溶菌酶家族(PF00959和PF05838)生成了95个新的人工序列,它们与任何已知天然蛋白的最大序列同源性低于40%。在所选择的序列中,89个序列中有78个(88%)表达良好,78个序列中有24个(31%)是可溶的。然后纯化了6个高表达蛋白,发现它们都具有活性,但Michaelis-Menten活性明显低于HEWL或先前生成的人工蛋白L056和L070(图3f)。
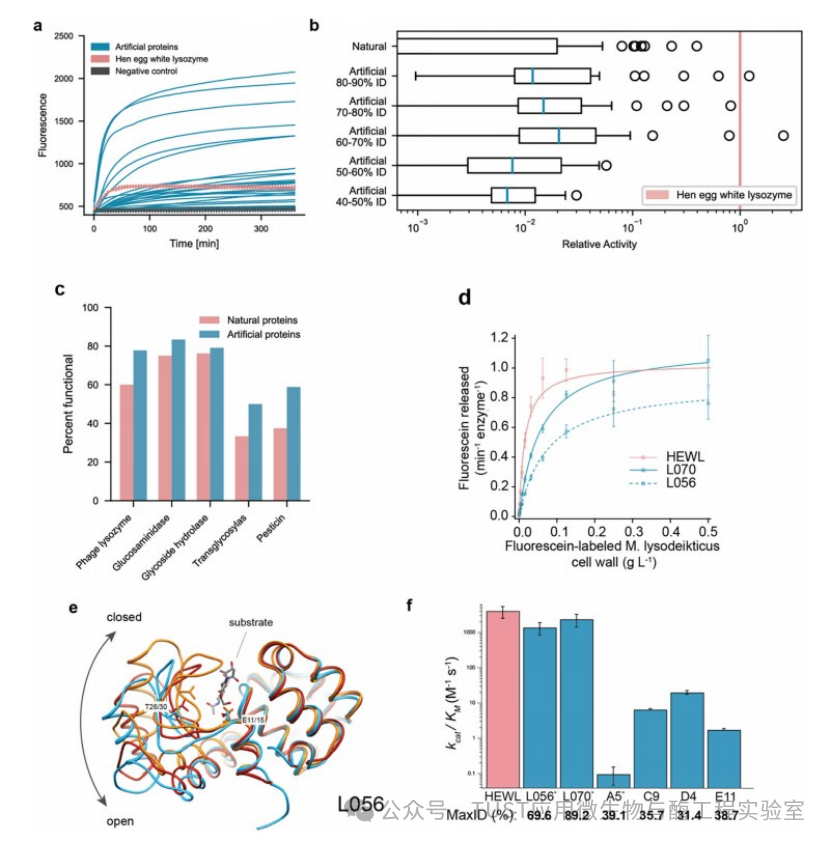
图3 人工蛋白质序列在功能上与任何已知蛋白质的同源性低至44%,表现出与高度进化的天然蛋白质相当的催化效率,并表现出与已知天然折叠相似的结构
4、ProGen在其他蛋白质系统中的适用性
在跨越许多家族的通用蛋白质序列数据集上进行训练,ProGen设计来自任何家族的蛋白质,并提供相应的控制标签作为输入。为了在溶菌酶家族之外探索这种能力,评估了ProGen在生成和预测功能全长序列方面的表现,这些序列来自于其他方法已经应用过的家族:分支酸变位酶(CM)和苹果酸脱氢酶(MDH)。将ProGen微调到这些家族的未对齐蛋白序列,并为每个家族生成64000个独特序列。生成的蛋白质表现出与自然序列文库相似的保守性(图4a,4d)。利用先前发表的CM和MDH蛋白序列及其实验测量的分析数据,还评估了ProGen对这些序列的模型似然与其相对活性的一致性,并将其与原始研究中使用的生成方法(bmDCA和proteinGAN)进行了比较。具体来说,使用ProGen测量了人工序列的每个标记对数似然,并使用它们来预测人工序列是否应该起作用。在CM函数数据上,ProGen对数似然的曲线下面积(AUC)为0.85,显著优于bmDCA的AUC (p<0.0001,双尾检验,n=1617),后者的AUC为0.78(图4c)。在MDH函数数据上,ProGen对数似然的AUC为0.94(图4f),优于ProteinGAN判别器评分的AUC为0.87 (p<0.1,双尾检验,n=56)。总之,ProGen的模型可能性与两个不同蛋白质数据集(分支酸变位酶和苹果酸脱氢酶)的实验测量数据更一致,而不是来自专门为这些家族量身定制的原始研究的序列生成方法。
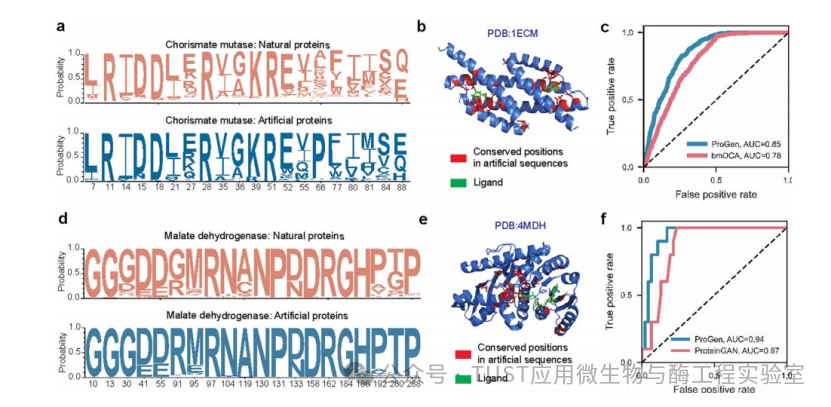
图4 条件语言建模在其他蛋白质系统中的适用性

结论

本研究表明,仅用进化序列数据训练的最先进的基于transformer的条件语言模型可以生成跨蛋白质家族的功能性人工蛋白质。进一步的分析表明,模型ProGen已经学会了一种灵活的蛋白质序列表示,可以应用于不同的家族,如溶菌酶、分支酸变位酶和苹果酸脱氢酶。该模型的应用包括生成高概率的功能蛋白的合成文库,用于新发现或迭代优化。结合不断增加的序列数据来源和更具代表性的控制标签,可以预见,使用基于深度学习的语言模型来精确设计新型蛋白质以解决生物学、医学和环境中的重要问题,是极具潜力的。

原文链接

https://doi.org/10.1038/s41587-022-01618-2

END

文案编辑:孟祥波
审 稿:彭 冲
排 版:魏立坤
高颜值免费 SCI 在线绘图(点击图片直达)
往期精品(点击图片直达文字对应教程)






 研究人员开发的ProGen语言模型,通过训练能生成功能性的人工蛋白质序列,适用于多种蛋白质家族。实验表明,人工蛋白活性与天然蛋白相当,且结构保守,证明了AI在蛋白质设计领域的潜力。
研究人员开发的ProGen语言模型,通过训练能生成功能性的人工蛋白质序列,适用于多种蛋白质家族。实验表明,人工蛋白活性与天然蛋白相当,且结构保守,证明了AI在蛋白质设计领域的潜力。













































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








