模型评估
学习目标
- 知道模型评估中的SSE、“肘”部法、SC系数和CH系数的实现原理
1 误差平方和(SSE)
误差平方和(SSE \The sum of squares due to error)具体概念通过如下举例介绍:
举例:
(下图中数据-0.2, 0.4, -0.8, 1.3, -0.7, 均为真实值和预测值的差)

在k-means中的应用:
误差平方和公式

- K 表示聚类中心的个数(簇个数)
- Ci 表示簇
- p 表示样本值
- mi 表示簇的质心
先计算每个簇中的每个点和质心点的差值平方和,再计算所有簇的差值平方和

公式各部分内容:

上图中: k=2
- SSE图最终的结果,对图松散度的衡量.(eg: SSE(左图))
- SSE随着聚类迭代,其值会越来越小,直到最后趋于稳定:

- 如果质心的初始值选择不好,SSE只会达到一个不怎么好的局部最优解.

2 “肘”方法(用来画图的)
“肘”方法 (Elbow method) 主要是用于
确定聚类算法中的K值,
简单流程
- 遍历k值,2-100
- 计算每个k值模型对应的sse值
- 绘制k值和sse值的折线图,肘部图
具体流程如下:

(1)对于n个点的数据集,迭代计算k from 1 to n,每次聚类完成后计算每个点到其所属的簇中心的距离的平方和;
(2)平方和是会逐渐变小的,直到k==n时平方和为0,因为每个点都是它所在的簇中心本身。
(3)在这个平方和变化过程中,会出现一个拐点也即“肘”点,
下降率突然变缓时即认为是最佳的k值。
在决定什么时候停止训练时,肘形判据同样有效,数据通常有更多的噪音,在
增加分类无法带来更多回报时,我们停止增加类别。
代码实现
my_kmeans.inertia_ # 获取SSE值
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
from sklearn.metrics import calinski_harabasz_score
def dm01_SSE误差平方和求模型参数():
sse_list = []
# 产生数据random_state=22固定好
x, y = make_blobs(
n_samples=1000,
n_features=2,
centers=[[-1, -1], [0, 0], [1, 1], [2, 2]],
cluster_std=[0.4, 0.2, 0.2, 0.2],
random_state=22)
for clu_num in range(1, 100):
my_kmeans = KMeans(n_clusters=clu_num, max_iter=100, random_state=0)
my_kmeans.fit(x)
sse_list.append(my_kmeans.inertia_ ) # 获取sse的值
plt.figure(figsize=(18, 8), dpi=100)
plt.xticks(range(0, 100, 3), labels=range(0, 100, 3))
plt.grid()
plt.title('sse')
plt.plot(range(1, 100), sse_list, 'or-')
plt.show()
# 通过图像可观察到 n_clusters=4 sse开始下降趋缓, 最佳值4
3 轮廓系数法(SC)
轮廓系数法(Silhouette Coefficient)结合了聚类的凝聚度(Cohesion)和分离度(Separation),用于评估聚类的效果:

目的:
内部距离最小化,外部距离最大化

计算样本i到同簇其他样本的平均距离ai,ai 越小样本i的簇内不相似度越小,说明样本i越应该被聚类到该簇。
计算样本i到最近簇Cj 的所有样本的平均距离bij,称样本i与最近簇Cj 的不相似度,定义为样本i的簇间不相似度:bi =min{bi1, bi2, ..., bik},bi越大,说明样本i越不属于其他簇。
求出所有样本的轮廓系数后再求平均值就得到了
平均轮廓系数。
平均轮廓系数的取值范围为[-1,1],系数越大,聚类效果越好。
簇内样本的距离越近,簇间样本距离越远
案例:
下图是500个样本含有2个feature的数据分布情况,我们对它进行SC系数效果衡量:

n_clusters = 2 The average silhouette_score is : 0.7049787496083262
n_clusters = 3 The average silhouette_score is : 0.5882004012129721
n_clusters = 4 The average silhouette_score is : 0.6505186632729437
n_clusters = 5 The average silhouette_score is : 0.56376469026194
n_clusters = 6 The average silhouette_score is : 0.4504666294372765
n_clusters 分别为 2,3,4,5,6时,SC系数如下,是介于[-1,1]之间的度量指标:
每次聚类后,每个样本都会得到一个轮廓系数,当它为1时,说明这个点与周围簇距离较远,结果非常好,当它为0,说明这个点可能处在两个簇的边界上,当值为负时,暗含该点可能被误分了。
从平均SC系数结果来看,K取3,5,6是不好的,那么2和4呢?
k=2的情况:

k=4的情况:

n_clusters = 2时,第0簇的宽度远宽于第1簇;
n_clusters = 4时,所聚的簇宽度相差不大,因此选择K=4,作为最终聚类个数。
总结
- S=(b-a)/max(a,b)
- a:簇内的平均误差 误差→距离
- b:簇外最近簇的平均误差
- s取值范围是[-1,1],越接近1,聚类效果越好
代码实现
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
from sklearn.metrics import silhouette_score
def dm02_轮廓系数SC():
tmp_list = []
# 产生数据random_state=22固定好
x, y = make_blobs(n_samples=1000, n_features=2, centers=[[-1, -1], [0, 0], [1, 1], [2, 2]],
cluster_std=[0.4, 0.2, 0.2, 0.2], random_state=22)
for clu_num in range(2, 100):
my_kmeans = KMeans(n_clusters=clu_num, max_iter=100, random_state=0)
my_kmeans.fit(x)
ret = my_kmeans.predict(x)
tmp_list.append(silhouette_score(x, ret)) # sc
plt.figure(figsize=(18, 8), dpi=100)
plt.xticks(range(0, 100, 3), labels=range(0, 100, 3))
plt.grid()
plt.title('sse')
plt.plot(range(2, 100), tmp_list, 'ob-')
plt.show()
# 通过图像可观察到 n_clusters=4 取到最大值
dm02_轮廓系数SC()
4 CH系数(用的少)
CH系数(Calinski-Harabasz Index)追求的是:
类别内部数据的协方差越小越好,类别之间的协方差越大越好(换句话说:类别内部数据的距离平方和越小越好,类别之间的距离平方和越大越好)。
这样的Calinski-Harabasz分数s会高,分数s高则聚类效果越好。
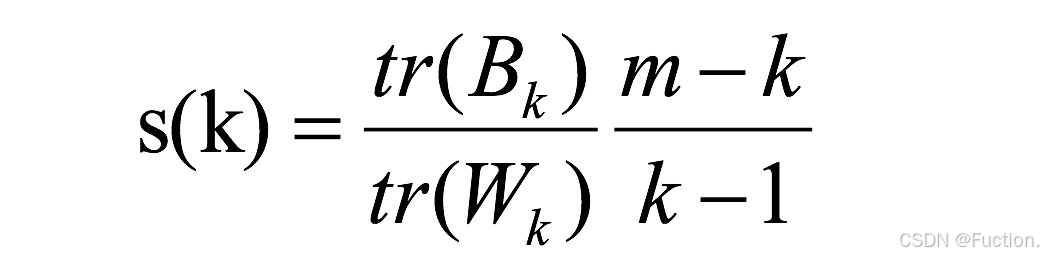
tr为
矩阵的迹,
Bk为类别之间的协方差矩阵,
Wk为类别内部数据的协方差矩阵;
m为训练集样本数,
k为类别数。

使用矩阵的迹进行求解的理解:
矩阵的对角线可以表示一个物体的相似性
在机器学习里,主要为了获取数据的特征值,那么就是说,在任何一个矩阵计算出来之后,都可以简单化,只要获取矩阵的迹,就可以表示这一块数据的最重要的特征了,这样就可以把很多无关紧要的数据删除掉,达到简化数据,提高处理速度。
CH需要达到的目的:
用尽量少的类别聚类尽量多的样本,同时获得较好的聚类效果。
代码实现
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
from sklearn.datasets import make_blobs
from sklearn.metrics import calinski_harabasz_score
def dm03_ch系数():
tmp_list = []
# 产生数据random_state=22固定好
x, y = make_blobs(
n_samples=1000,
n_features=2,
centers=[[-1, -1], [0, 0], [1, 1], [2, 2]],
cluster_std=[0.4, 0.2, 0.2, 0.2], random_state=22)
for clu_num in range(2, 100):
my_kmeans = KMeans(n_clusters=clu_num, max_iter=100, random_state=0)
my_kmeans.fit(x)
ret = my_kmeans.predict(x)
tmp_list.append(calinski_harabasz_score(x, ret)) # sc
plt.figure(figsize=(18, 8), dpi=100)
plt.xticks(range(0, 100, 3), labels=range(0, 100, 3))
plt.grid()
plt.title('ch')
plt.plot(range(2, 100), tmp_list, 'og-')
plt.show()
5 小结
- sse
- 误差平方和的值越小越好
- 肘部法
- 下降率突然变缓时即认为是最佳的k值
- SC系数
- 取值为[-1, 1],其值越大越好
- CH系数
- 分数s高则聚类效果越好
- CH需要达到的目的:用尽量少的类别聚类尽量多的样本,同时获得较好的聚类效果。
对比与选择
•适用场景:轮廓系数法适用于样本数量较多且簇形状较为规则的情况,能够较好地反映样本在聚类中的归属感。而CH系数法适用于样本数量较少且簇形状较为不规则的情况,能够较好地反映簇内数据的紧密度和簇间数据的分离度。
• 计算复杂度:轮廓系数法的计算复杂度较高,因为需要计算每个样本与其他样本的距离。CH系数法的计算复杂度相对较低,主要依赖于簇内和簇间的协方差矩阵
• 选择依据:如果数据集的样本数量较多且簇形状较为规则,可以选择轮廓系数法来评估聚类效果。如果数据集的样本数量较少且簇形状较为不规则,可以选择CH系数法来评估聚类效果
























 4421
4421

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








