C - 基因工程
小Hi和小Ho正在进行一项基因工程实验。他们要修改一段长度为N的DNA序列,使得这段DNA上最前面的K个碱基组成的序列与最后面的K个碱基组成的序列完全一致。
例如对于序列”ATCGATAC”和K=2,可以通过将第二个碱基修改为”C”使得最前面2个碱基与最后面两个碱基都为”AC”。当然还存在其他修改方法,例如将最后一个碱基改为”T”,或者直接将最前面两个和最后面两个碱基都修改为”GG”。
小Hi和小Ho希望知道在所有方法中,修改碱基最少的方法需要修改多少个碱基。
Input
第一行包含一个整数T(1 <= T <= 10),代表测试数据的数量。
每组测试数据包含2行,第一行是一个由”ATCG”4个大写字母组成的长度为N(1 <= N <= 1000)的字符串。第二行是一个整数K(1 <= K <= N)。
Output
对于每组数据输出最少需要修改的碱基数量。
Sample Input
2
ATCGATAC
2
ATACGTCT
6 Sample Output
1
3 【解析】
这道题还是一道很不错的题,接下来讲一下解法:
为了方便,我们可以先复制一个原串,如下:
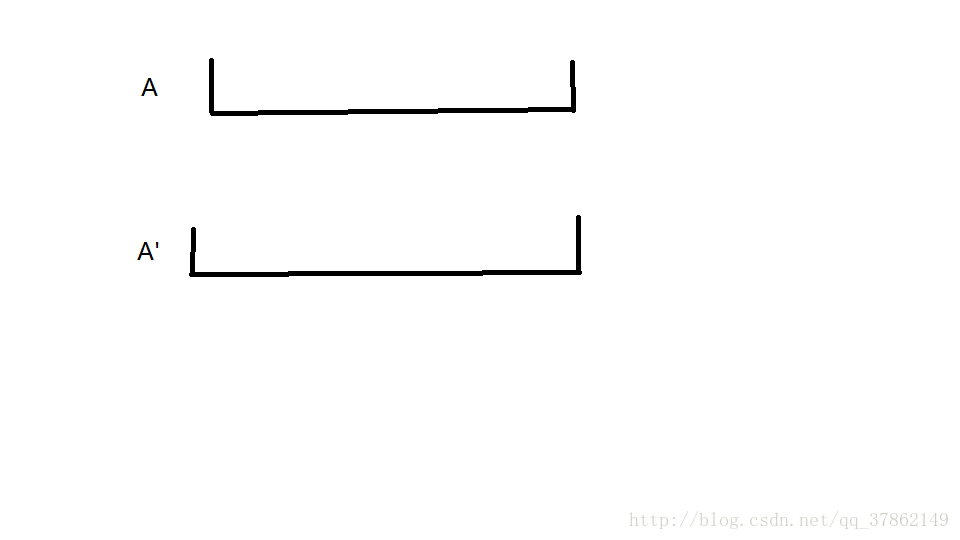
看图已知,1与2相等(复制过来的),若要满足题意,则2与3相等,那么3与4相等……如此,我们便可以发现了这样简单的规律。若要满足题意,这些点都是必须要满足的。这里讲述的是当k<1/2n的情况,当k>1/2n时,也一样。
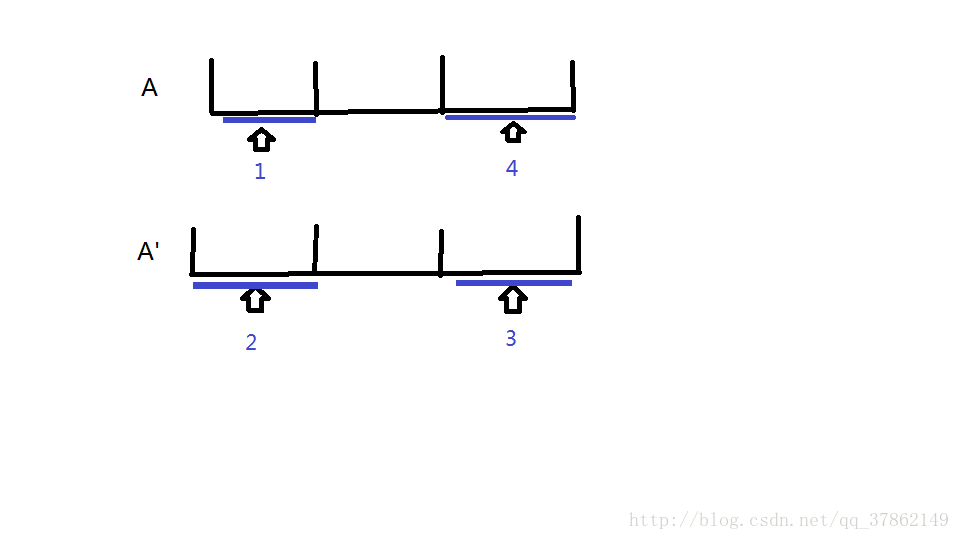
后我们再进行一个小的贪心即可。
【代码】
#include<cstdio>
#include<iostream>
#include<cstring>
#include<string>
#include<algorithm>
using namespace std;
string s;
int T[6];
int t,k;
int main()
{
scanf("%d",&t);
while(t--)
{
cin>>s;
int n=s.size();
scanf("%d",&k);
int ans=0;
for(int i=0;i<min(k,n-k);i++)
{
int tot=0;
T[1]=T[2]=T[3]=T[4]=0;
for(int j=i;j<n;j+=n-k)
{
tot++;
if(s[j]=='A')
T[1]++;
if(s[j]=='T')
T[2]++;
if(s[j]=='C')
T[3]++;
if(s[j]=='G')
T[4]++;
}
ans+=tot-*max_element(T+1,T+5);
}
printf("%d\n",ans);
}
}







 这篇博客探讨了基因工程在HihoCoder 1052问题中的应用,涉及如何修改DNA序列使得前K个碱基与后K个碱基相同。博主通过举例说明问题并提供了解题思路,指出在满足条件的情况下,需要修改的碱基数量的最小值。文章包含了题目输入输出格式,并给出了解题代码。
这篇博客探讨了基因工程在HihoCoder 1052问题中的应用,涉及如何修改DNA序列使得前K个碱基与后K个碱基相同。博主通过举例说明问题并提供了解题思路,指出在满足条件的情况下,需要修改的碱基数量的最小值。文章包含了题目输入输出格式,并给出了解题代码。














 487
487

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








