前言
这个例子展示了如何使用 NeuroKit 来分析长时间的生理数据(大于10秒) ,比如静息状态。
import neurokit2 as nk
import pandas as pd
import matplotlib.pyplot as plt
数据集
数据来自1名参与者的静息状态数据(试图不去想任何事情,闭上眼睛),以100Hz 频率(5 x 60x100 = 30000个数据点)记录的5分钟生理信号,包含以下信号: 心电ECG,脉搏波PPG,呼吸RSP。
# 在线数据集
data = nk.data("bio_resting_5min_100hz")
数据预处理和可视化
我们重点分析心电ECG信号,可以使用 ecg _ process ()进行数据预处理。然后,使用 ecg _ plot ()可视化这些信号(示例中只选择3000个数据点(30s)进行可视化)。
# 心电信号ECG预处理
ecg_signals, info = nk.ecg_process(data["ECG"], sampling_rate=100)
nk.ecg_plot(ecg_signals[:3000], sampling_rate=100)


特征提取
心率变异性(HRV)是指逐次心跳周期差异(R-R)的变化情况。在这里,我们将使用peaks,即心跳峰值的出现,作为以下 HRV 函数中的输入参数来提取特征指标。

# 峰值R查找
peaks, info = nk.ecg_peaks(data["ECG"], sampling_rate=100)


时域特征
# 根据peaks值提取时域特征值
hrv_time = nk.hrv_time(peaks, sampling_rate=100, show=True)
hrv_time

频域特征
hrv_freq = nk.hrv_frequency(peaks, sampling_rate=100, show=True, normalize=False)
hrv_freq

非线性特征
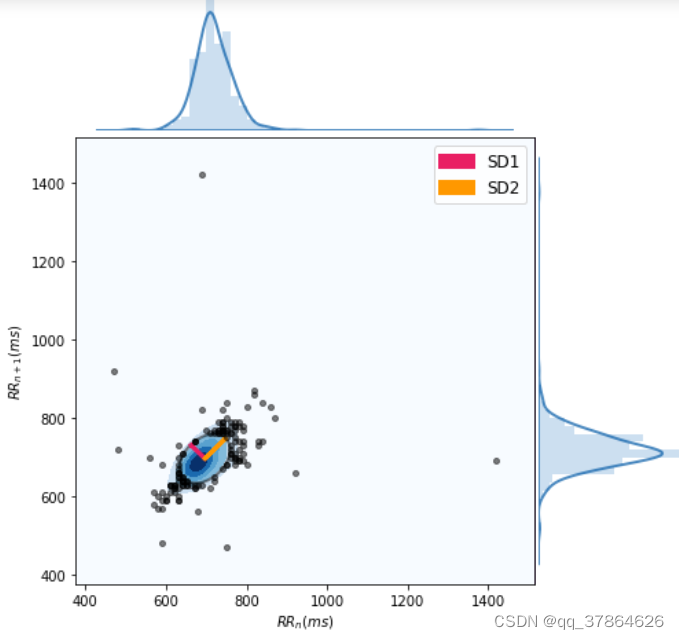
hrv_nonlinear = nk.hrv_nonlinear(peaks, sampling_rate=100, show=True)
hrv_nonlinear







 这篇博客演示了如何利用NeuroKit库处理和分析长时间的生理信号,特别是心电图(ECG)。首先,从数据集中获取静息状态的ECG、PPG和RSP信号,然后进行预处理和可视化。接着,通过查找ECG的R峰值来提取HRV的时域、频域和非线性特征,展示了一系列与心脏健康相关的指标。
这篇博客演示了如何利用NeuroKit库处理和分析长时间的生理信号,特别是心电图(ECG)。首先,从数据集中获取静息状态的ECG、PPG和RSP信号,然后进行预处理和可视化。接着,通过查找ECG的R峰值来提取HRV的时域、频域和非线性特征,展示了一系列与心脏健康相关的指标。


















 6436
6436

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








