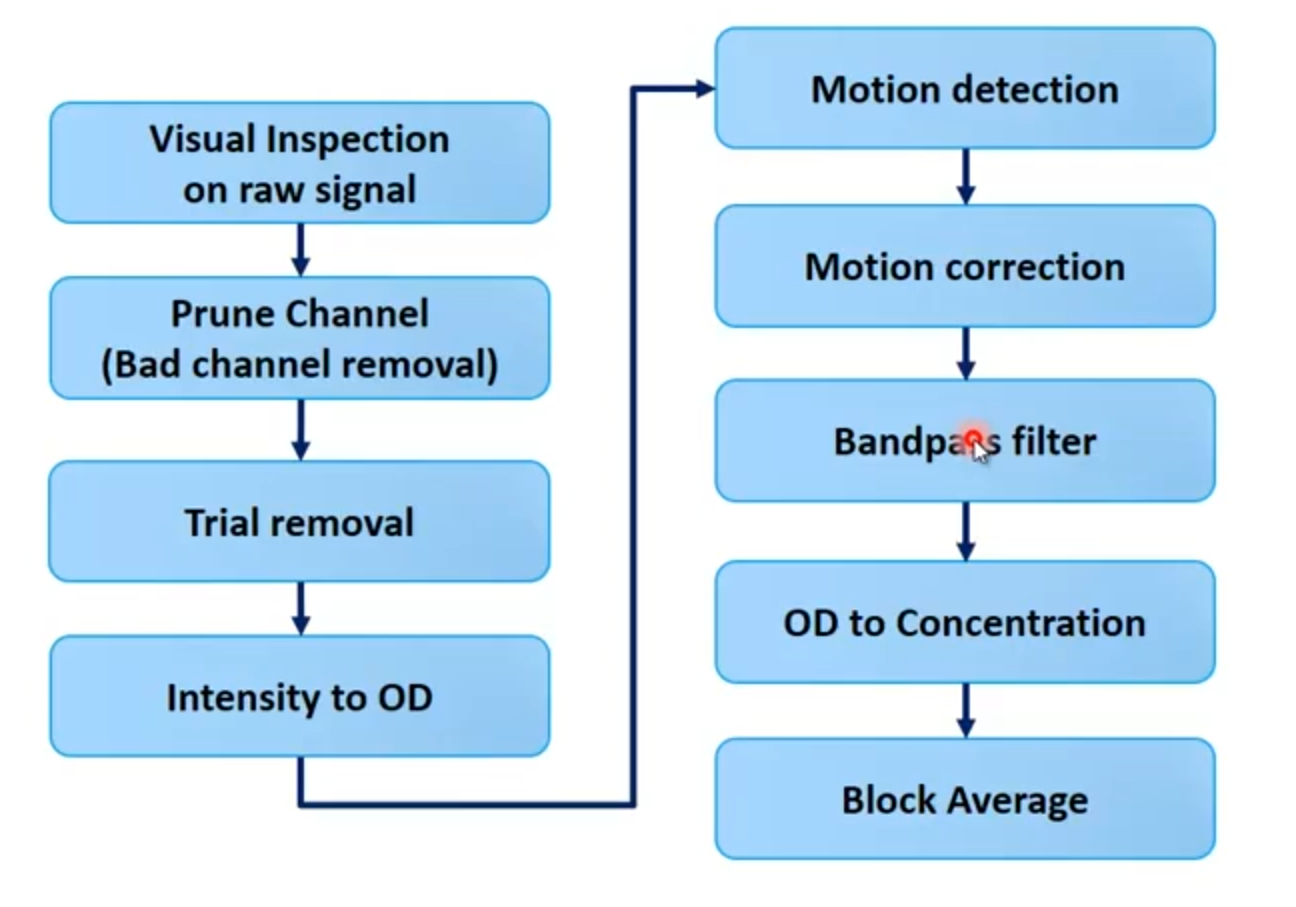
卢家峰处理的工作流
0.01-0.1或者0.2Hz的这个频率区间范围内。
0.2-2.5HZ的有心跳呼吸等非脑功能血氧相关的特征。
查看信号
因为头部的动作会带来血氧信号的改变,所以我们对于近红外数据可以进行相关的剔除。但是我使用的数据,Aux(加速度计感应信号并不存在)。

在Homer2中的View中查看plot power spectrum

Homer2和Homer3存在差异。


我们可以看到这些信号的主要分析频率集中在2.5Hz以下。
真正有效的信号是0.01Hz到0.1Hz之间。
心跳周期在0.83Hz左右,会有心跳。
因为运动任务心跳会加速,可能在1.1Hz左右。

好像确实找不到心跳的peak。
血管壁的是0.1HZ左右。

近程红外处理器的剔除
数据的筛查


刺激的设置
新版本的homer3设置为了tsv文件编辑器

 对于同样事件的处理,我们可以删除剩余的时间标记,使用次数和实验时长来切割。
对于同样事件的处理,我们可以删除剩余的时间标记,使用次数和实验时长来切割。

如果有这个选项,可以通过这个信号来进行运动起始的更准确的判断。

分析流

推荐的处理流程

对于channel的判断和移除
裁剪后,做插值,
使用小波和
(需要先计算出两个值之后再使用)CBSI都比较好用。CBSI 的核心思想是利用氧合血红蛋白(HbO)和脱氧血红蛋白(HbR)信号之间的负相关关系来校正信号。

多次运动矫正,处理的效果最好。
并非是最优的,但是可以覆盖很多场景。
如果stmi处理好的话,可以直接来一个hmrR_BlockAvg。
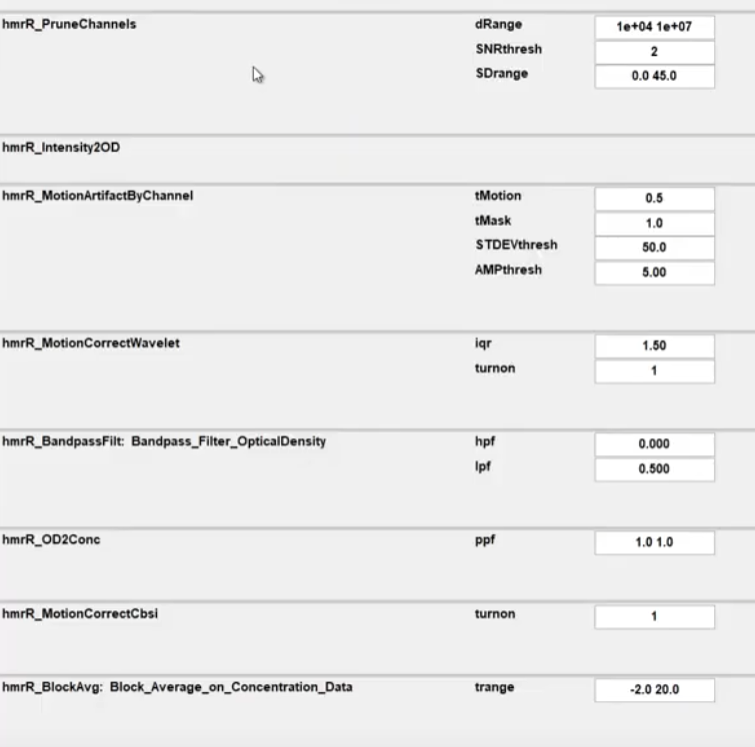

参数的选择,因为如果是光的强度信息(其强度一般在几千到几万)但具体范围取决于设备、光源功率、探测器灵敏度以及实验设置。
所以视频中的大概率是光密度的raw data

这是推荐的参数,但是仍然需要根据实际的数据进行调整。
因为涉及到的传感器的单位,测得的值的域比较奇怪,所以不进行通道的滤除。

处理之后的HbO和HbR单位不太对。

其变化趋势是正确的。但是数值不太对应的上。
通过一些


在研究 fNIRS 数据的变化性时,可以使用多种指标来量化信号的变化特征。这些指标可以帮助你更好地理解大脑活动的动态特性。以下是常用的变化性指标及其计算方法:
通常使用变异系数来判断有效性。
1. 标准差(Standard Deviation, SD)
- 定义:标准差是衡量数据离散程度的指标,反映了信号围绕均值的波动情况。
- 适用场景:用于评估信号的整体波动性。
- 公式:
S D = 1 N − 1 ∑ i = 1 N ( x i − x ˉ ) 2 SD = \sqrt{\frac{1}{N-1} \sum_{i=1}^N (x_i - \bar{x})^2} SD=N−11i=1∑N(xi−xˉ)2
其中:- ( x_i ) 是第 ( i ) 个时间点的信号值。
- ( \bar{x} ) 是信号的均值。
- ( N ) 是时间点的总数。
2. 变异系数(Coefficient of Variation, CV)
- 定义:变异系数是标准差与均值的比值,用于比较不同信号之间的相对变化性。
- 适用场景:适用于比较不同通道或不同受试者之间的变化性。
- 公式:
C V = S D x ˉ CV = \frac{SD}{\bar{x}} CV=xˉSD
3. 均方根(Root Mean Square, RMS)
- 定义:均方根是信号幅度的平均值,反映了信号的整体强度。
- 适用场景:用于评估信号的整体能量。
- 公式:
R M S = 1 N ∑ i = 1 N x i 2 RMS = \sqrt{\frac{1}{N} \sum_{i=1}^N x_i^2} RMS=N1i=1∑Nxi2
4. 信号幅度面积(Signal Amplitude Area, SAA)
- 定义:信号幅度面积是信号绝对值的时间积分,反映了信号的总变化量。
- 适用场景:用于评估信号的总变化量。
- 公式:
S A A = ∑ i = 1 N ∣ x i ∣ SAA = \sum_{i=1}^N |x_i| SAA=i=1∑N∣xi∣
5. 动态范围(Dynamic Range)
- 定义:动态范围是信号最大值与最小值的差值,反映了信号的波动范围。
- 适用场景:用于评估信号的极端变化。
- 公式:
D y n a m i c R a n g e = max ( x i ) − min ( x i ) Dynamic\ Range = \max(x_i) - \min(x_i) Dynamic Range=max(xi)−min(xi)
6. 近似熵(Approximate Entropy, ApEn)
- 定义:近似熵是衡量信号复杂性的指标,反映了信号的随机性和不可预测性。
- 适用场景:用于评估信号的复杂性。
- 计算方法:
- 需要设置参数 ( m )(嵌入维度)和 ( r )(容差)。
- 具体计算方法较为复杂,可以参考相关文献或使用工具箱(如 MATLAB 的
approximateEntropy函数)。
7. 样本熵(Sample Entropy, SampEn)
- 定义:样本熵是近似熵的改进版本,用于衡量信号的复杂性。
- 适用场景:用于评估信号的复杂性,适用于短时间序列。
- 计算方法:
- 类似于近似熵,但更稳定。
8. 功率谱密度(Power Spectral Density, PSD)
- 定义:功率谱密度是信号在频域上的能量分布,反映了信号的频率特性。
- 适用场景:用于分析信号的频率成分。
- 计算方法:
- 使用傅里叶变换计算信号的功率谱。
- 可以提取特定频带(如 0.01-0.1 Hz)的能量作为变化性指标。
9. 滑动窗口分析(Sliding Window Analysis)
- 定义:在时间序列上滑动一个固定大小的窗口,计算每个窗口内的变化性指标(如标准差、RMS 等)。
- 适用场景:用于分析信号的变化性随时间的变化。
10. 分形维数(Fractal Dimension, FD)
- 定义:分形维数是衡量信号自相似性的指标,反映了信号的复杂性和不规则性。
- 适用场景:用于评估信号的复杂性。
- 计算方法:
- 常用的方法包括 Higuchi 分形维数和盒计数法。
11. Hurst 指数(Hurst Exponent)
- 定义:Hurst 指数是衡量时间序列长期依赖性的指标,反映了信号的自相似性。
- 适用场景:用于评估信号的长程相关性。
- 计算方法:
- 使用重标极差分析(R/S 分析)或小波变换。
12. 相位同步(Phase Synchronization)
- 定义:相位同步是衡量两个信号之间相位关系的指标,反映了信号之间的协同性。
- 适用场景:用于分析不同通道或区域之间的功能连接。
代码示例
以下是一个 MATLAB 代码示例,计算 HbO 信号的标准差和变异系数:
% 假设 HbO 是一个时间序列(nTimePoints × 1)
HbO = data.procResult.conc_HbO(:, 1); % 取第一个通道的 HbO 数据
% 计算标准差
SD = std(HbO);
fprintf('Standard Deviation: %.4f\n', SD);
% 计算变异系数
CV = std(HbO) / mean(HbO);
fprintf('Coefficient of Variation: %.4f\n', CV);
相关文献
以下是一些关于 fNIRS 变化性指标的文献:
-
标准差和变异系数:
- Scholkmann, F., Kleiser, S., Metz, A. J., Zimmermann, R., Mata Pavia, J., Wolf, U., & Wolf, M. (2014). A review on continuous wave functional near-infrared spectroscopy and imaging instrumentation and methodology. NeuroImage, 85, 6-27.
-
近似熵和样本熵:
- Richman, J. S., & Moorman, J. R. (2000). Physiological time-series analysis using approximate entropy and sample entropy. American Journal of Physiology-Heart and Circulatory Physiology, 278(6), H2039-H2049.
-
分形维数和 Hurst 指数:
- Higuchi, T. (1988). Approach to an irregular time series on the basis of the fractal theory. Physica D: Nonlinear Phenomena, 31(2), 277-283.
-
功率谱密度:
- Welch, P. D. (1967). The use of fast Fourier transform for the estimation of power spectra: A method based on time averaging over short, modified periodograms. IEEE Transactions on Audio and Electroacoustics, 15(2), 70-73.
总结
- 变化性指标可以帮助你量化 fNIRS 信号的动态特性。
- 常用的指标包括标准差、变异系数、均方根、近似熵、功率谱密度等。
- 根据研究目标选择合适的指标,并结合具体实验设计进行分析。
如果你有更多问题或需要进一步的帮助,请随时告诉我!
ai生成的
使用ai,构建一个数据处理的流程说明:
未测试。
回顾任务目标和数据处理流程来明确需要提取的数据。以下是详细的梳理:
1. 任务回顾
- 任务目标: 评估帕金森病人的运动能力,使用近红外脑功能数据(fNIRS)。
- 运动范式: 手指对捏、拿方块、握拳维持。
- 数据类型: 近红外脑功能数据(fNIRS),主要关注氧合血红蛋白(Oxy-Hb)、脱氧血红蛋白(Deoxy-Hb)和总血红蛋白(Total-Hb)的变化。
- 回归目标: 可能是运动能力评分、任务完成时间或准确率等连续变量。
2. 需要提取的数据
根据任务目标,您需要提取以下数据:
(1) 血红蛋白浓度数据
- 氧合血红蛋白(Oxy-Hb): 反映大脑皮层的神经活动。
- 脱氧血红蛋白(Deoxy-Hb): 反映局部氧耗。
- 总血红蛋白(Total-Hb): 反映局部血流量变化。
(2) 试次数据
- 每个试次(手指对捏、拿方块、握拳维持)的血红蛋白浓度变化。
- 试次的基线数据(任务前的静息状态)。
- 试次的任务数据(任务执行期间)。
(3) 时间序列特征
- 均值: 试次期间血红蛋白浓度的平均值。
- 标准差: 试次期间血红蛋白浓度的波动情况。
- 峰值: 试次期间血红蛋白浓度的最大值。
- 斜率: 血红蛋白浓度随时间的变化趋势。
(4) 频域特征
- 功率谱密度: 反映血红蛋白浓度变化的频率特性。
(5) 运动分型相关数据
- 根据运动分型量表(震颤/中轴症状得分),提取不同分型病人的血红蛋白浓度特征。
3. 数据提取流程
以下是具体的数据提取步骤:
(1) 提取血红蛋白浓度数据
- 使用
hmrOD2Conc将光密度(OD)数据转换为血红蛋白浓度数据。 - 提取 Oxy-Hb、Deoxy-Hb 和 Total-Hb 的时间序列。
(2) 提取试次数据
- 根据任务时间标记,截取每个试次的数据。
- 提取基线时间段(如任务前 5 秒)和任务时间段(如任务执行期间 10 秒)。
(3) 计算时间序列特征
- 对每个试次,计算 Oxy-Hb、Deoxy-Hb 和 Total-Hb 的均值、标准差、峰值和斜率。
(4) 计算频域特征
- 对每个试次,使用傅里叶变换或小波变换计算功率谱密度。
(5) 结合运动分型
- 根据运动分型量表,将病人分为步态障碍为主、震颤为主、强直-迟缓型。
- 提取不同分型病人的血红蛋白浓度特征。
4. 代码示例
以下是一个简单的 MATLAB 代码示例,展示如何提取试次数据和计算特征:
% 假设 conc 是血红蛋白浓度数据,shape 为 (时间点, 通道数, 血红蛋白类型)
% trials 是试次时间标记,shape 为 (试次数, 2),每行包含试次的开始和结束时间
% 提取试次数据
baseline_window = 5 * fs; % 基线时间段(5秒)
task_window = 10 * fs; % 任务时间段(10秒)
for i = 1:size(trials, 1)
start_time = trials(i, 1);
end_time = trials(i, 2);
% 提取基线数据
baseline_data = conc(start_time - baseline_window : start_time, :, :);
% 提取任务数据
task_data = conc(start_time : end_time, :, :);
% 计算均值
baseline_mean = mean(baseline_data, 1);
task_mean = mean(task_data, 1);
% 计算标准差
baseline_std = std(baseline_data, 0, 1);
task_std = std(task_data, 0, 1);
% 计算峰值
baseline_peak = max(baseline_data, [], 1);
task_peak = max(task_data, [], 1);
% 计算斜率
baseline_slope = polyfit(1:size(baseline_data, 1), baseline_data, 1);
task_slope = polyfit(1:size(task_data, 1), task_data, 1);
% 保存结果
trial_features(i).baseline_mean = baseline_mean;
trial_features(i).task_mean = task_mean;
trial_features(i).baseline_std = baseline_std;
trial_features(i).task_std = task_std;
trial_features(i).baseline_peak = baseline_peak;
trial_features(i).task_peak = task_peak;
trial_features(i).baseline_slope = baseline_slope;
trial_features(i).task_slope = task_slope;
end
5. 总结
您需要提取的数据包括:
- 血红蛋白浓度数据(Oxy-Hb、Deoxy-Hb、Total-Hb)。
- 试次数据(基线数据和任务数据)。
- 时间序列特征(均值、标准差、峰值、斜率)。
- 频域特征(功率谱密度)。
- 运动分型相关特征。
通过以上步骤,您可以系统地提取所需数据,并为后续的回归分析提供高质量的特征输入。如果有进一步的问题,欢迎随时提问!
针对您的任务(帕金森病人的运动能力评估,使用近红外脑功能数据),以下是推荐的预处理流程及对应的 Homer2 函数:
1. 数据导入与初步整理
- 函数:
hmrFlowInput - 作用: 导入原始数据并进行初步整理,确保数据格式正确。
2. 光强度数据转换为光密度(OD)
- 函数:
hmrIntensity2OD - 作用: 将原始光强度数据转换为光密度(OD)数据,这是后续分析的基础。
3. 去除全局噪声与漂移
- 函数:
hmrBandpassFilt - 作用: 使用带通滤波器(如 0.01-0.5 Hz)去除低频漂移和高频噪声(如心跳、呼吸)。
- 参数建议:
- 低频截止频率:0.01 Hz(去除漂移)。
- 高频截止频率:0.5 Hz(去除心跳等高频噪声)。
4. 运动伪影检测与校正
- 函数:
hmrMotionArtifact+hmrMotionCorrectWavelet(或hmrMotionCorrectPCA) - 作用:
- 先使用
hmrMotionArtifact检测运动伪影。 - 然后使用
hmrMotionCorrectWavelet(小波变换)或hmrMotionCorrectPCA(PCA方法)校正运动伪影。
- 先使用
- 选择依据:
- 如果数据中运动伪影较多且复杂,推荐使用
hmrMotionCorrectWavelet。 - 如果数据中运动伪影较少且简单,推荐使用
hmrMotionCorrectPCA。
- 如果数据中运动伪影较多且复杂,推荐使用
5. 剔除无效通道
- 函数:
enPruneChannels - 作用: 剔除信号质量较差或无效的通道,确保后续分析的可靠性。
6. 光密度数据转换为血红蛋白浓度
- 函数:
hmrOD2Conc - 作用: 将光密度(OD)数据转换为氧合血红蛋白(Oxy-Hb)、脱氧血红蛋白(Deoxy-Hb)和总血红蛋白(Total-Hb)浓度数据。
7. 剔除刺激相关伪影
- 函数:
enStimRejection - 作用: 剔除与任务刺激相关的伪影或噪声,确保数据纯净。
8. 数据标准化
- 函数: 自定义标准化(如 z-score 标准化)
- 作用: 对每个通道的数据进行标准化,消除通道间的基线差异。
- 实现方式:
- 使用 MATLAB 或 Python 的标准化函数(如
zscore)。
- 使用 MATLAB 或 Python 的标准化函数(如
9. 试次截取与基线校正
- 函数: 自定义脚本
- 作用:
- 根据任务时间标记截取每个试次的数据。
- 使用试次前的基线时间段(如任务前 5 秒)进行基线校正。
- 实现方式:
- 使用 Homer2 的
hmrBlockAvg或自定义脚本。
- 使用 Homer2 的
10. 数据可视化与检查
- 函数:
plotTrials - 作用: 绘制试次数据,检查预处理效果。
完整预处理流程示例
% 1. 导入数据
rawData = hmrFlowInput('your_data_file.nirs');
% 2. 光强度转光密度
dod = hmrIntensity2OD(rawData);
% 3. 带通滤波
dod_filtered = hmrBandpassFilt(dod, 0.01, 0.5, rawData.fs);
% 4. 运动伪影检测与校正
[motionArtifact, motionIndex] = hmrMotionArtifact(dod_filtered, rawData.fs);
dod_corrected = hmrMotionCorrectWavelet(dod_filtered, motionIndex);
% 5. 剔除无效通道
dod_pruned = enPruneChannels(dod_corrected);
% 6. 光密度转浓度
conc = hmrOD2Conc(dod_pruned, rawData.ppf);
% 7. 剔除刺激相关伪影
conc_clean = enStimRejection(conc, rawData.stim);
% 8. 数据标准化
conc_normalized = zscore(conc_clean);
% 9. 试次截取与基线校正
% 假设 trials 是试次时间标记
trials = [start1, end1; start2, end2; ...];
baseline_window = 5 * rawData.fs; % 基线时间段(5秒)
conc_trials = hmrBlockAvg(conc_normalized, trials, baseline_window);
% 10. 数据可视化
plotTrials(conc_trials);
总结
通过以上步骤,您可以完成近红外数据的预处理,包括:
- 数据导入与转换。
- 噪声去除与运动伪影校正。
- 通道筛选与数据标准化。
- 试次截取与基线校正。
函数说明
function dod = hmrFlowInput( d )
for i = 1:size(d,2);
lst = find(isnan(d(:,i)));
d(lst,i) = nanmean(d(:,i));
clear lst
end
% percent change
dm = mean(abs(d),1);
nTpts = size(d,1);
dod = (abs(d)./(ones(nTpts,1)*dm));
if ~isempty(find(d(:)<=0))
warning( 'WARNING: Some data points in d are zero or negative.' );
end
这个函数 hmrFlowInput 的主要用途是对输入的近红外光强度数据进行预处理,具体包括以下步骤:
1. 处理缺失值(NaN)
- 代码:
for i = 1:size(d,2) lst = find(isnan(d(:,i))); d(lst,i) = nanmean(d(:,i)); clear lst end - 作用:
- 检查每一列(通常是每个通道的光强度数据)是否存在缺失值(NaN)。
- 如果存在缺失值,用该列的均值(忽略 NaN 计算)填充缺失值。
- 意义:
- 确保数据中没有缺失值,避免后续计算出错。
2. 计算光强度的百分比变化
- 代码:
dm = mean(abs(d),1); nTpts = size(d,1); dod = (abs(d)./(ones(nTpts,1)*dm)); - 作用:
- 计算每个通道的光强度均值
dm。 - 将每个通道的光强度数据除以其均值,得到百分比变化(
dod,即光密度变化)。
- 计算每个通道的光强度均值
- 意义:
- 将光强度数据标准化为相对于均值的百分比变化,消除不同通道之间的基线差异。
3. 检查数据有效性
- 代码:
if ~isempty(find(d(:)<=0)) warning( 'WARNING: Some data points in d are zero or negative.' ); end - 作用:
- 检查数据中是否存在零或负值。
- 如果存在,发出警告提示。
- 意义:
- 光强度数据应为正值,零或负值可能是数据采集错误或异常,需要进一步检查。
4. 输出结果
- 输出变量:
dod: 光密度变化数据,表示光强度相对于均值的百分比变化。
总结
这个函数的主要用途是对近红外光强度数据进行预处理,包括:
- 处理缺失值(NaN)。
- 计算光强度的百分比变化(光密度变化,
dod)。 - 检查数据中是否存在零或负值,并发出警告。
适用场景:
- 在近红外数据分析中,光强度数据通常需要转换为光密度变化(
dod),以便后续分析(如计算血红蛋白浓度)。 - 这个函数是数据预处理的一部分,通常会在
hmrIntensity2OD或其他光密度转换函数之前使用。
示例
假设输入数据 d 是一个矩阵,表示多个通道的光强度数据(每列是一个通道,每行是一个时间点):
d = [100, 200, 300;
NaN, 210, 310;
110, 220, 320];
dod = hmrFlowInput(d);
输出 dod 是光密度变化数据,表示每个通道的光强度相对于其均值的百分比变化。
截取一部分时间点来观察处理后的图像

























 2116
2116

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








