我用的是rstudio server ,BiocManager::install("limma")运行时出现了该报错
这是一个基础的编译器,但是R找不到
我在终端运行which gfortran,能找到一个

用whereis,找到了三个

我在rstudio中运行Sys.getenv("PATH"),发现没有which gfortran发现的那个编译器的路径,但有/user/bin,而且其中也有该编译器。

也就是说我现在运行的R虚拟环境是一个独立的环境,它可能不会直接使用我在本地系统中安装的编译器或其他依赖项,它默认使用which gfortran中的那个,但我在R环境变量中并没有添加该路径。运行该命令添加:
Sys.setenv(PATH = paste(Sys.getenv("PATH"), "/home/majortom/anaconda3/envs/R_4.3.1/bin", sep=":"))
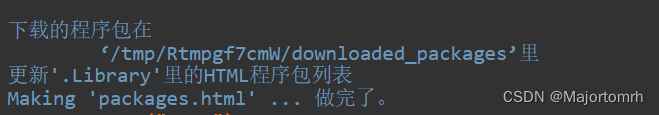
ok完成
验证一下
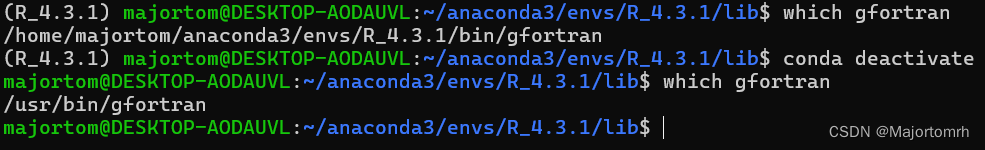
确实如此
tip修改环境变量方法:
options("repos") #查看当前设置源,一般选清华
chooseBioCmirror()
清华源6(默认)
国外源2+翻墙





















 4044
4044

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








