在经过与老师的探讨后,我们决定将之前创新实训的课题,继续深挖,开发一套基于人工智能模型的生物序列分析平台,内置了传统nlp模型与生物信息中的各类模型的整合,打算做到60个算法的集合,最终我们实现了两个平台上一共56个的算法。
本博客主要记录的内容是本人在智能生物药物推荐平台的工作日志:
项目的意义:
我们使用了基于用户-药物-疾病的三重推荐系统,通过引入基因等额外的辅助信息通过异构图网络的辅助计算相似度的方式确保了推荐在相关领域的专业性,这样的工作可以极大提高用户在自主查询疾病与药物的时候的高效,我们通过可视化对应的选择路径,也为用户选择药物做了一定的安全保证。我们的系统也可以通过推荐的相似度计算,来预测某些疾病的并发症等等,然后合理推荐用户相关的药物,真正处于一种专家的角度考虑用户的需求。
刚开始打算将本系统的python后台作为动态的数据,通过前端-后端- python脚本的形似进行,但是因为对于药物相似度检测,与药物种类的稀疏性,我们预先将所有药物分析的推理过程与预测结果都预测出来,然后将其保存在mysql数据库中,最终通过阶段性的新药与数据库的更新,保证最终的推荐与检索功能具有一定的实时性。
下面是平台开发的过程:
一、开发环境:
- 因为使用的环境复杂,我们部署的服务器也是学校内的公用服务器,因此我们用Anaconda来进行本实验的环境管理:进入Anaconda的官网进行下载,对应的操作系统的版本,然后点击Download按钮就开始下载了,找到下载好的后缀为sh的安装包,在Linux里面.sh文件是可执行的脚本文件,需要用命令bash来进行安装。然后我们需要修改profile文件, sudo vi /etc/profile来匹配最终安装后的新环境。在文件的末尾加代码: export PATH=$PATH:/home/software/anaconda3/bin, 这个代码需要根据自己安装anaconda的路径来确定。创建虚拟环境:conda create --name pytorch python=3.8。最后激活对应的环境:source activate pytorch
- 在激活的环境下,安装对应的python包,直接通过pip install进行,需要安装的包大致有:selenium,lxml,urllib,pytorch(torch),numpy,pandas等,如果在运行过程中出现了其他安装包未安装的问题,可以通过报错所提示的缺失包通过pip install或者conda install来进行安装.
- 前端:安装nodejs,直接去nodejs的官网下载安装包以后进行安装。使用npm安装cnpm(一个淘宝镜像,可以解决某些包安装不了的问题),yarn等,然后对前端提供的代码进行相应操作的包安装,同样,因为所需要的安装包过多,可以通过递归安装的方式进行安装,具体安装内容已经在readme中写好。
- 后端调度代码,首先需要maven包管理程序,在官网找到对应版本的maven进行安装,然后直接安装,进入后端环境之后,根绝代码所提供的配置信息进行依赖驱动包的下载与安装,这些都是通过maven来管理的。后端还涉及到一部分云端管理的服务器部署方面,可以用nginx这个软件进行转发部署等,具体安装流程也是通过远程镜像将安装包下载到远程服务器上,然后通过对应的启动部署脚本与配置单进行部署。
二、系统设计与数据的收集过程:
系统设计:代码模块组成,功能
1.数据收集模块:首先通过爬虫爬到需要处理的数据集,然后用python构造数据集以后,通过人工划分出训练集与测试集
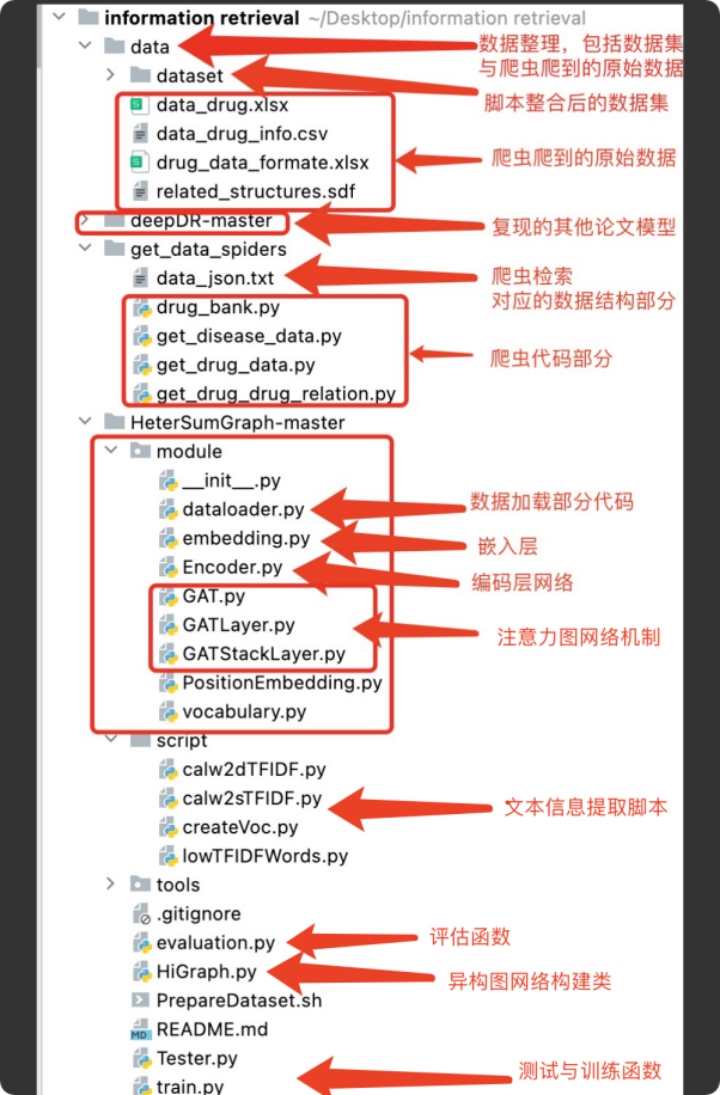
2.模型构造模块:搭建基于注意力机制的异构图模型,然后去将已经有的数据集中的特征去拟合异构图中的不同种类的节点与元路径,通过已经收集的到的数据集对模型进行训练与测试,然后将模型部署到一台性能比较不错对GPU服务器上。
3.对比复现模块:通过在已经有的研究中比较本实验使用对方法,对谷歌学术上面的文章进行了调研,发现已经有人做这个方向的研究了,但是基于注意力机制的异构图模型还没有人去用,由于数据集量与目前选择的异构图构架较为简单,所以性能比不上文献中收集到的模型的性能,但也在已有方法上面进行了复现与比较。
4.后端服务管理:用Springboot写了一套管理python代码预测调用,并部署到阿里云服务器上面。使用MVC的构架,有利于后期的拓展与维护,这样可以有效加强系统的健壮性。
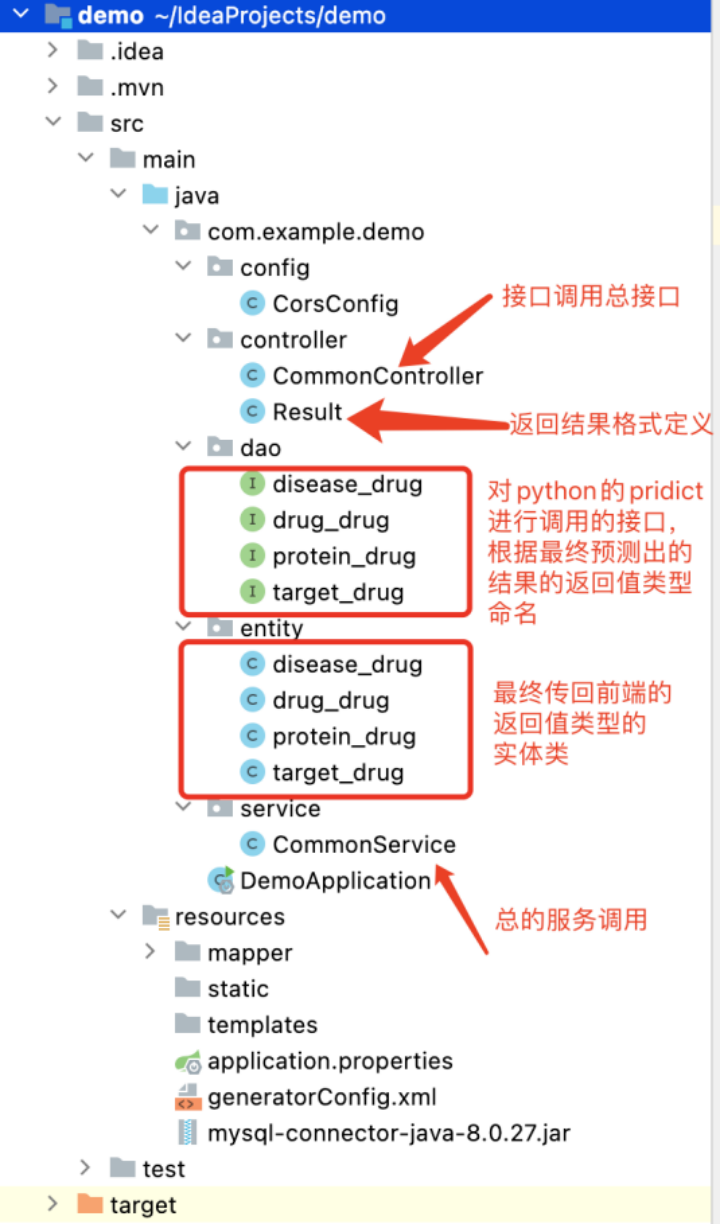
5.前端构图展示:用React搭配mobx内存管理写了一套基于函数式组建的前端展示页面,具有的功能有,通过注意力机制的异构图模型,预测某一个疾病的潜在药物,对论文存在的模型进行了复现结果的展示。
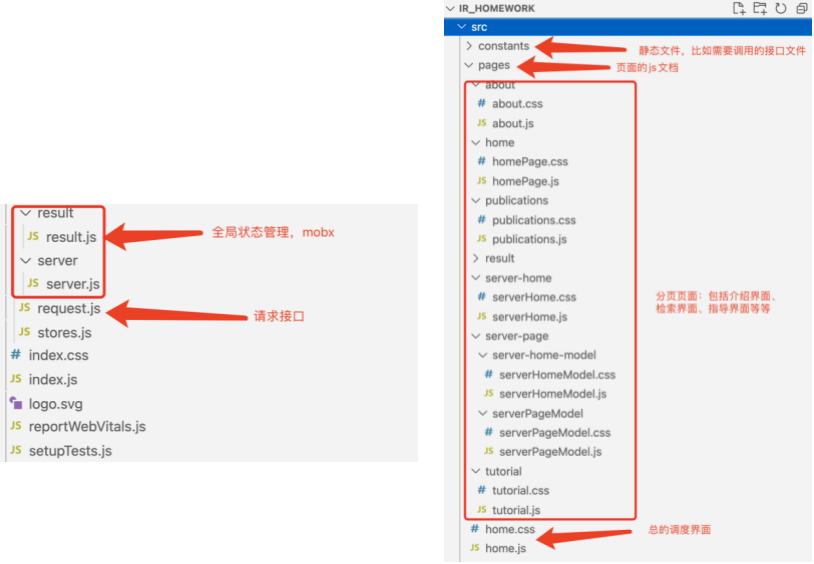
详细设计:每个模块的具体介绍
(1)数据收集:
(1)收集了疾病和对应药物的具体信息,信息来源自https://www.drugfuture.com/fda/,使用爬虫技术,在网页的网络请求中抓取到对应网页,以及药物信息对应的结构体
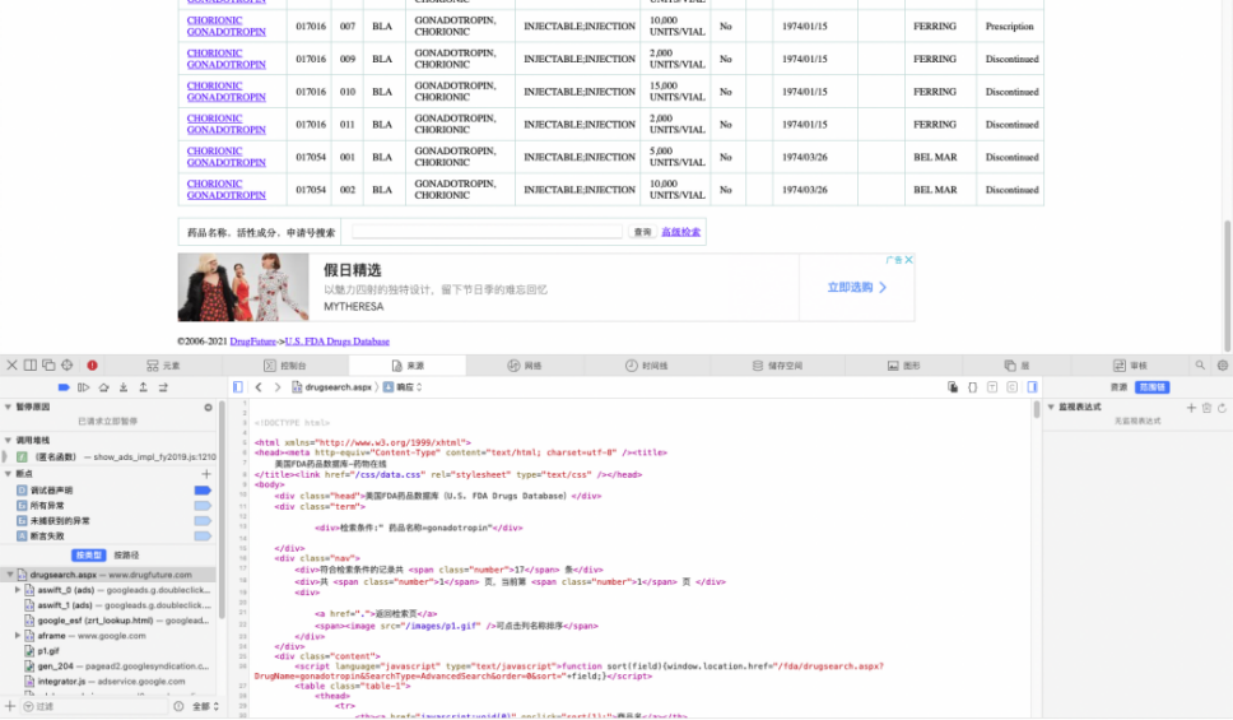

CHORIONIC GONADOTROPIN , 089313 , 001 , ANDA, GONADOTROPIN, CHORIONIC , Injectable; Injection, 5,000 UNITS/VIAL , No , , -- , , QUAD PHARMS , Discontinued的结构进行收集,最终得到一个对应的csv表格,具体有差不多20000条数据
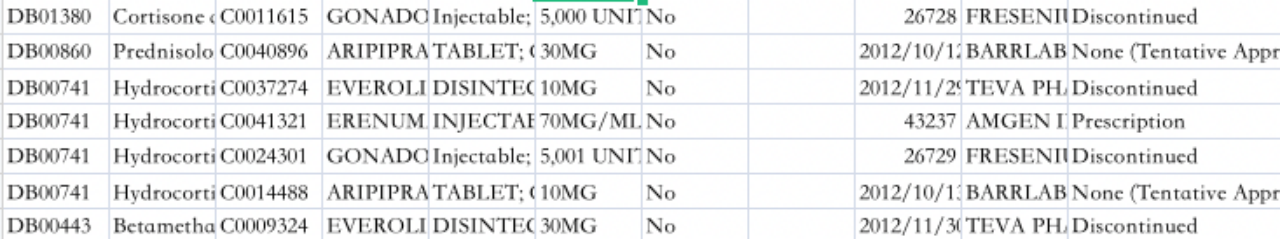
( 获取到的几个药物id与药物名称)
爬取到到对应信息结构。包括名字信息,活性成分信息,。所针对疾病名称等,发现本过程的爬虫收集的信息总类不是特别的多,对药物与疾病直接的关联程度不太强,如果仅仅使用本数据集进行模型训练与最终预测的的话,可能效果并不是太好,于是构建了一个小型的神经网络,去拟合了一下(一个具有4层全连接与双层LSTM的网络用来提取对应药物的embedding,然后通过embedding进行聚类,对分在同一类中的模型的疾病进行置信度划分。最终得到一个具有得分的疾病list信息)和之前预期一样,效果确实不是特别好,这个跟网路结构(没有使用太深的网络)与信息特征的不敏感性和数据量有着很大的关系,不能在这方面直接判定,于是下一步继续整合方案,去用爬虫整合更多的有关的药物信息。
(2)在搜索药物信息的时候,根据已有的药物名称与药物编号去爬取drugbank中的数据,找到对应的API,对应API可以爬取到很丰富的信息,比如作用与蛋白质的位置,药物与药物之间的关联等信息,爬取最终得到一个对应的data集
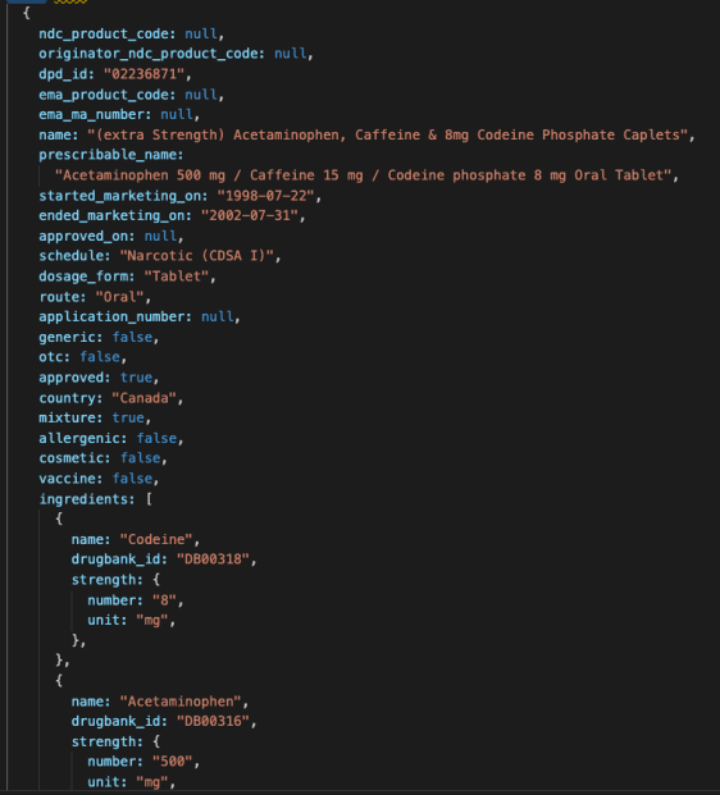
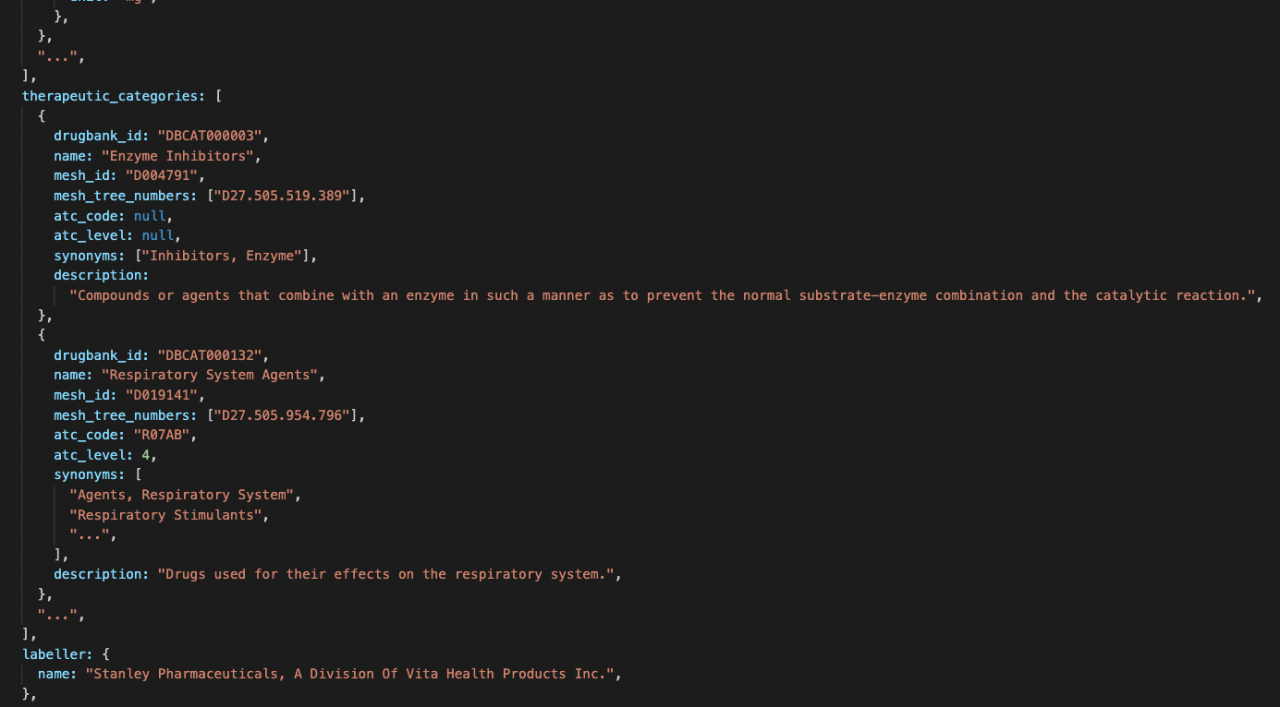
其中,在特征划分中,有一定作用的是therapeutic_categories(治疗类别)、schedule、ingredient(成分)等信息。将这些信息融入到之前爬到的数据中。然后在drugbank的官方API中,有一个对药物bond(关联,一般是药物定药物的目标、酶、载体和/或转运体键列表,这样或许可以通过对药物的target等信息与疾病表现出的生理特征之间建立一点特征相关的联系),于是,用python写了接口爬取此类信息,得到的信息事例如下:
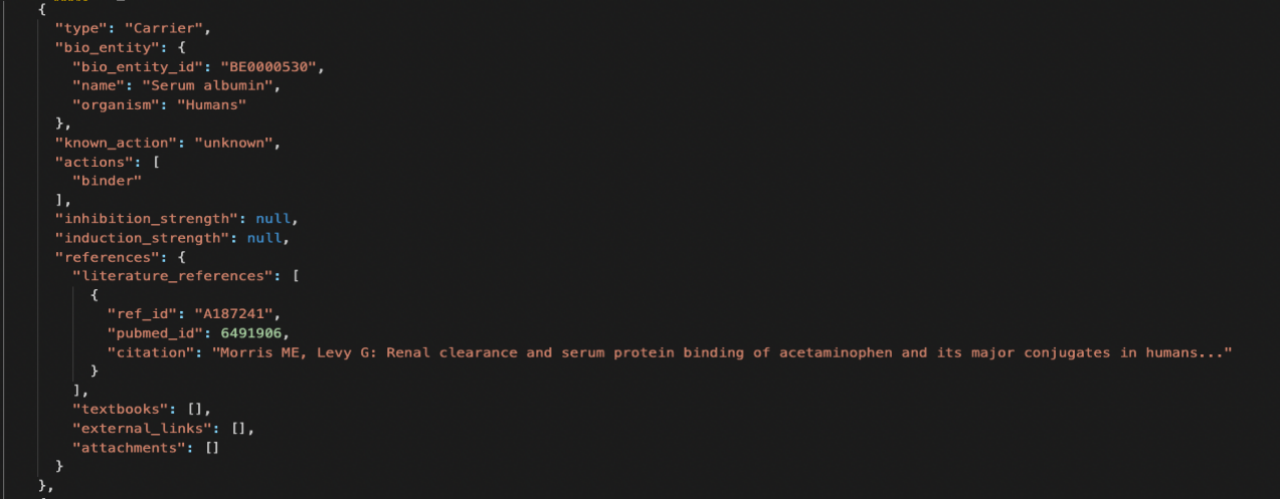
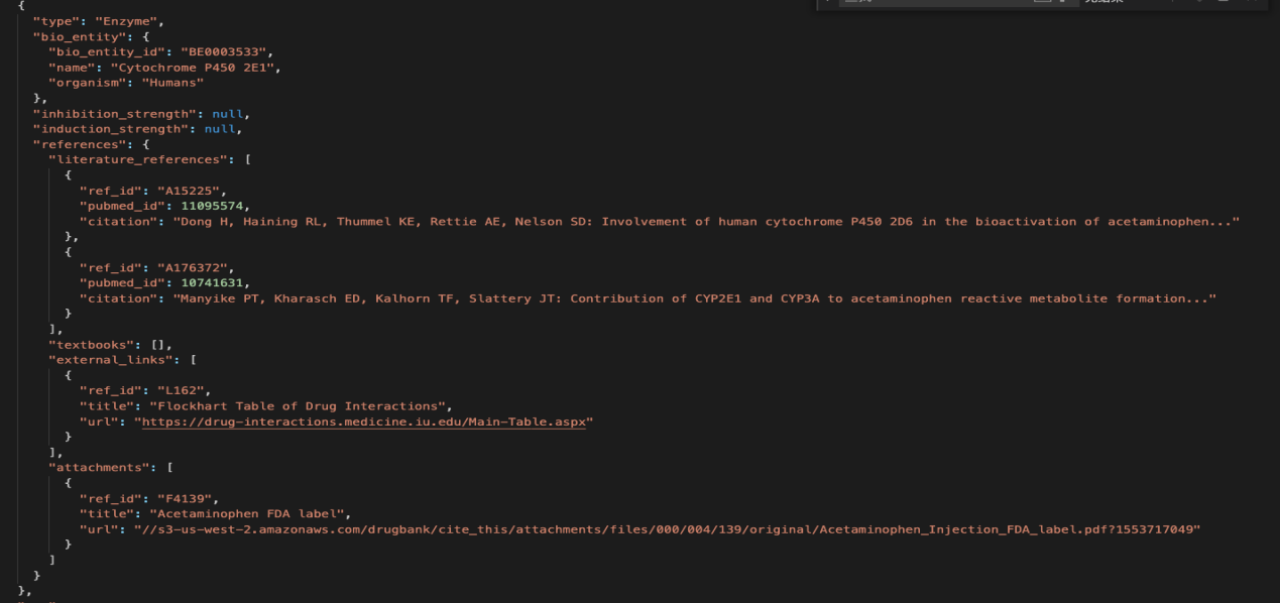
这个仅仅是跑出来的中间某一次的数据,其中对drug与疾病相关联的内容是Enzyme(酶的种类)、Carrier(运载体的种类)等具体信息,由于在计算这类中间隐含的相关变量时,需要将酶的特征向量与Carrier特征向量,我们需要根据最终得到的id进行集中编码,通过on-hot编码与初始化一个向量空间,去做映射,经由梯度下降去优化这个向量空间,最终我们需要得到的效果是这些中间变量能够起到drug与disease的中间相互关联的作用。
最终我们的由爬虫爬取然后再整合后需要是这样的一个结构:
{drugId:String,
drugName:String,
ingredient(成分):array,
therapeutic categories(治疗类别):array,
bond:{carrier:[id,name]}, {Enzyme:[id,name]}}
下面是对应的爬取到的例子信息:
{drugId: "02236871",
drugName: "(extra Strength) Acetaminophen, Caffeine & 8mg Codeine Phosphate Caplets",
ingredients: [{name: "Codeine",drugbank_id: "DB00318",},
{name: "Acetaminophen",drugbank_id: "DB00316", }],
therapeutic_categories: [{drugbank_id: "DBCAT000003",name: "Enzyme Inhibitors",},
{drugbank_id: "DBCAT000132",name: "Respiratory System Agents",}],
bond: [{"type": "Carrier","bio_entity_id": "BE0000530","name": "Serum albumin"},
{"type": "Enzyme","bio_entity_id": "BE0003533","name": "Cytochrome P450 2E1"}]}.
爬虫总结:之前也有想过直接使用javascript直接调用ajax封装好的axios包进行调用接口,直接去爬取对应的接口,然后通过KOA的nodejs去将这个javascript的接口直接部署到服务器使其自动爬取,但是因为浏览器的原因,javascript在文件管理方面没有python那么开放,不能自由随意地去读写文件,这使得爬取到的信息的保存等方式不能够实现,然后就直接去使用python进行了。
(2)模型构造模块:
- drug-other information-drug的模型很符合异构图的架构,异构图能够的多节点的构架图很符合我们这个数据,需要多定义几个节点类型,组建一个对应的drug-info1-drug,drug-info2-drug,drug-info3-drug...在通过数爬取到各类药物检索网站(https://www.drugfuture.com/fda/、https://drugbank.com等)得到我们最终的数据集。通过人工去将某些药物所能够治疗的疾病删去(随机)来构造对应的训练集与测试集的x,将完全匹配没有做删除的数据集作为训练集与测试集的y。
- 异构图的基本构架:异构图是一种特殊的异构信息网络,包含了多种类型的边和节点。例如,图1中的演员节点的特征可能包括性别,年龄和国籍,而电影节点的特征可能包括情节,演员等。边的不同类型可以体现在电影与导演的拍摄关系演员与电影的角色扮演关系。
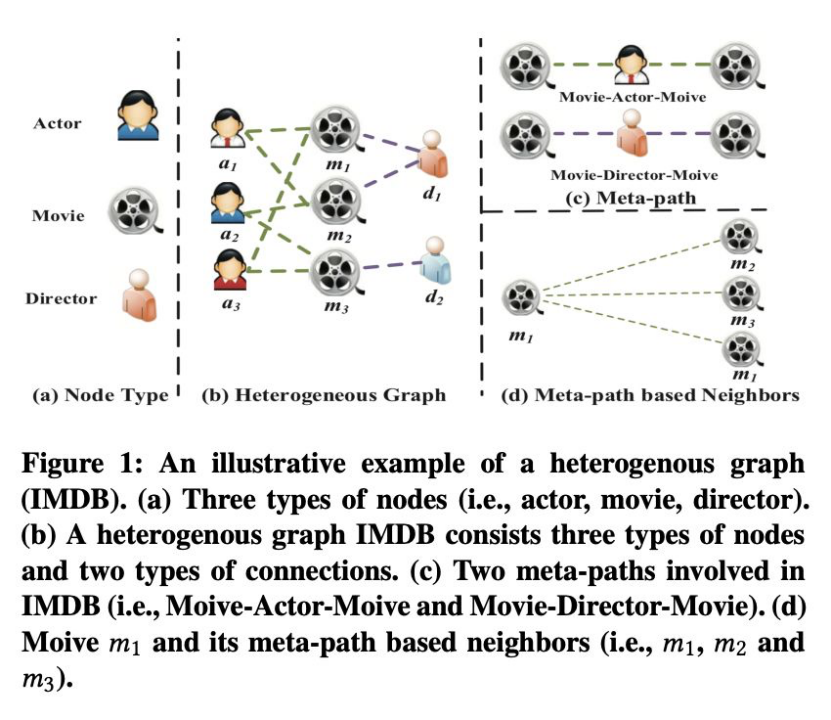
Figure 1: An illustrative example of a heterogenous graph(IMDB). (a) Three types of nodes (i.e., actor, movie, director).(b) A heterogenous graph IMDB consists three types of nodes and two types of connections. (c) Two meta-paths involved inIMDB (i.e., Moive-Actor-Moive and Movie-Director-Movie). (d)Moive m1 and its meta-path based neighbors (i.e., m1, m2 and m3).
我们将这个构架放到我们收集到的药物信息中,呈现方式如下图所视:

对于上图中的meta-path的构造可以为1.治疗类别-成分-治疗类别、2.药物-成分-药物,也可以在二元的meta-path结构上构造多元的meta-path:药物-成分-治疗类别-成分-药物,如此的多元meta-path。
我们可以通过一些邻接矩阵的乘法,来获得通过元路径获得药物的邻居,或许在这一个层面来得到一种药物的替代品,通过最终的判别函数,可以给对得到的邻居节点做一个打分,最终选出对应某几个得分最高的药物,
下面我们加入attention机制对异构图的这个模型进行多权重地学习,使得模型了解每个元路径的重要性,并为其分配适当的权重,我们可以通过一个deep Walk的类似的算法,构造词袋模型,作为最终回归结果的关系选择。如下图所示,先通过对基于节点的meta-path级的attention分配给不同连接边的权重,然后再通过一个语料级的attention对meta-path分配权重。
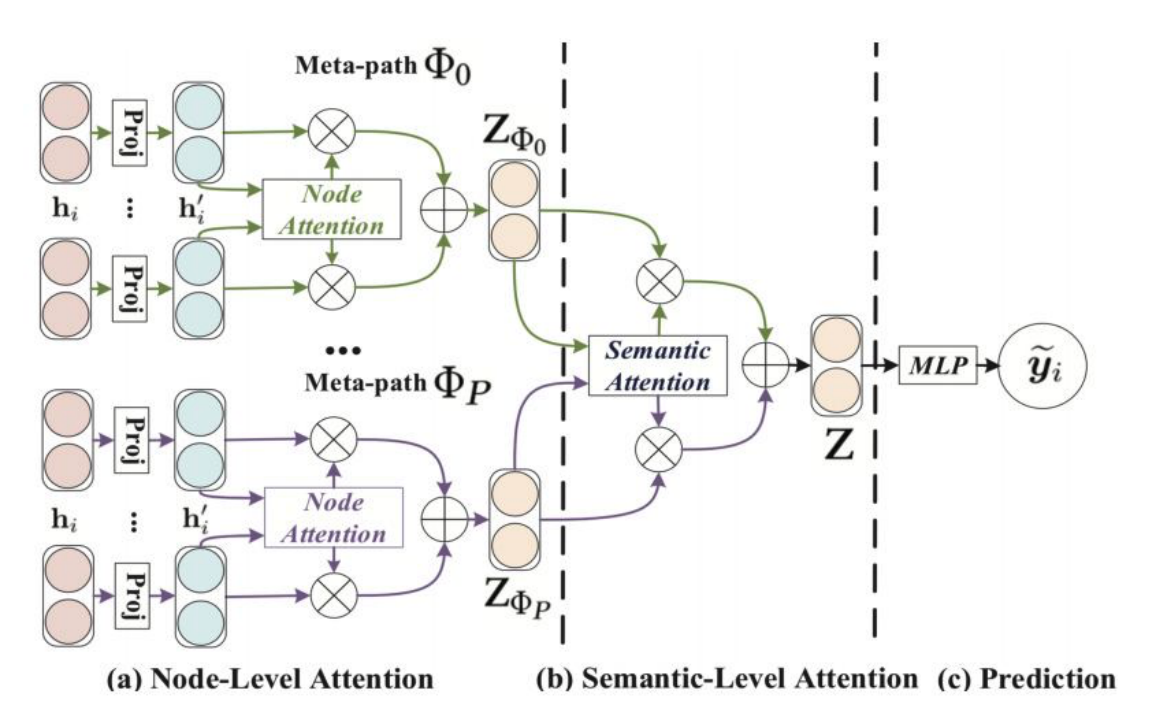
对于这部分的实现,可以通过pytorch中的transformers的包导入对应的attention机制,不过,介于本attention是基于异构图网络来构造的,所以现有的包工具可能不是太适配,于是自己通过矩阵与矩阵之间的运算,加上全连接、LSTM等现有的模块,通过pytorch组装而成。
下面是对于这部分的核心代码:
# 这一部分是对节点的embedding的初始化
node_feature = self.set_wnfeature(graph)
#这一步是对边的权重的初始化
edge_feature = self.n_feature_proj(self.set_snfeature(graph))
node_state = node_feature
#这一步是根据图的节点上面进行attention来赋予边上的权重
#这里的核心公式有:
edge_state = self.word2sent(graph, node_feature, edge_feature)
#这一步是根据图的meta-path进行利用attention提取没一个node的embedding
for i in range(self._n_iter):
node_state = self.sent2word(graph, node_state, edge_state)
edge_state = self.word2sent(graph, node_state, edge_state
#这一步的核心公式有:
result = self.wh(edge_state)
#其中对应的self.word2sent()函数是异构图论文实现attention函数,具体实现通过多头堆叠不同的单头注意力矩阵,单头的注意力机制代码
class GATLayer(nn.Module):
def __init__(self, in_dim, out_dim, feat_embed_size):
super().__init__()
self.fc = nn.Linear(in_dim, out_dim, bias=False)
self.feat_fc = nn.Linear(feat_embed_size, out_dim)
#这一行attn_fc的作用是对三个需要关注对embedding矩阵运算最终输出对应整个元路径对权重
self.attn_fc = nn.Linear(3 * out_dim, 1, bias=False)
#本处代码是对边进行注意力矩阵权重的计算
def edge_attention(self, edges):
#本行代码在于将已有的embedding特征向量提取出来
dfeat = self.feat_fc(edges.data["tfidfembed"]) # [edge_num, out_dim]
#整合两个对应的边构造关联矩阵权重的维度(两个embedding cat到一起可以对两个embedding的不同特征分别求对应的关联程度矩阵)
z2 = torch.cat([edges.src['z'], edges.dst['z'], dfeat], dim=1) # [edge_num, 3 * out_dim]
#通过atten_fc基础函数进行权重的计算
wa = F.leaky_relu(self.attn_fc(z2)) # [edge_num, 1]
#基于元路径的节点对(i,j)j 对 i 的重要性:
return {'e': wa}
def message_func(self, edges):
return {'z': edges.src['z'], 'e': edges.data['e']}
def reduce_func(self, nodes):
alpha = F.softmax(nodes.mailbox['e'], dim=1)
h = torch.sum(alpha * nodes.mailbox['z'], dim=1)
return {'sh': h}
def forward(self, g, h):
wnode_id = g.filter_nodes(lambda nodes: nodes.data["unit"] == 0)
snode_id = g.filter_nodes(lambda nodes: nodes.data["unit"] == 1)
swedge_id = g.filter_edges(lambda edges: (edges.src["unit"] == 1) & (edges.dst["unit"] == 0))
z = self.fc(h)
g.nodes[snode_id].data['z'] = z
g.apply_edges(self.edge_attention, edges=swedge_id)
g.pull(wnode_id, self.message_func, self.reduce_func)
g.ndata.pop('z')
h = g.ndata.pop('sh')
return h[wnode_id]代码实现难度,主要在于将药物drug与drug衍生出的各类信息通过tokenize进行表征embedding的初始化的构建,与节点拟合的代码实现上面。因为模型的数据集的数量不够大,而且模型迁移过程中还有一定的不太完全适配drug-info-drug的情况(简单的来说drug的衍生信息通过爬虫爬取到的重复度太小,还需要融合其他特征进行进一步的训练)
(3)对比复现模块:
其他找到的做的工作相似的论文复现:
《deepDR: a network-based deep learning approach to in silico drug repositioning》这篇论文想法基本上跟我所做的实验目标是一致的,不过这篇论文使用的融合特征有很多,在看了这篇论文的实验情况之后,也对自己的实验与数据做了很多的调整。
这篇论文中的模型构架是加入了drug-drug,drug-protein,drugideEffect、drugsimChemicalnet等众多在生物结构与信息上面十分重要的特征,然后经过一系列的融合模型与推理模型,最终得到对应疾病相关治疗药物。本篇论文中的模型架构如下图所示:
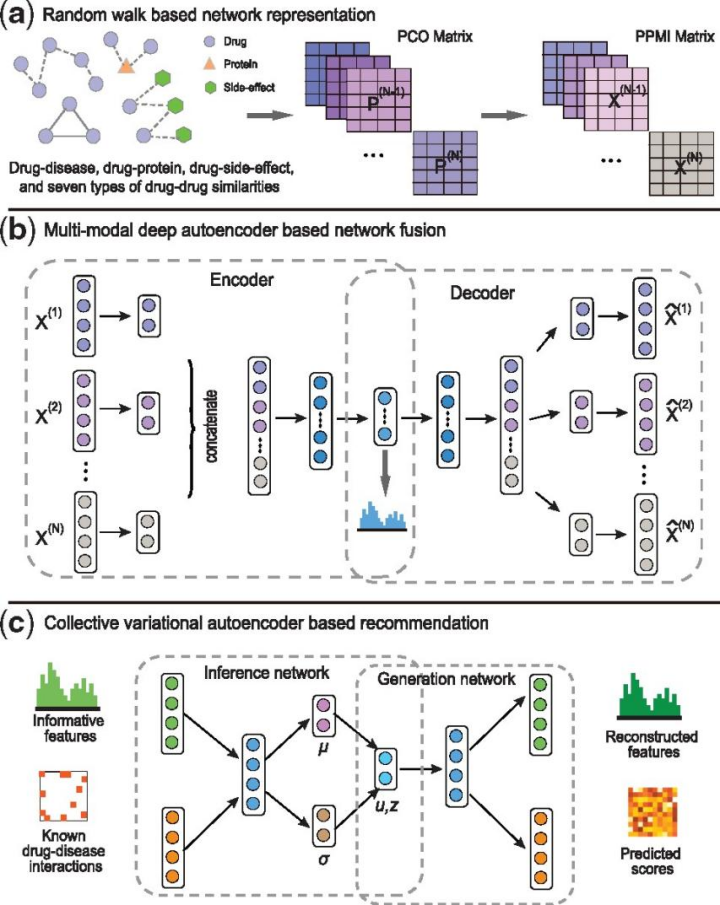
可以看出,该模型也是运用的图模型的知识,然后获得药物疾病与其他info的embedding以后通过卷积图卷积神经网络将各个现有的特征矩阵进行融合,经过多个深度学习的模型然后通过encoder和decoder去优化embedding在向量空间上面的空间聚集程度。在看了这个模型的论文以后,发现可以在我的模型基础之上进行进一步的开拓,将我所用的异构图attention机制输出的embedding不直接进行分类,可以后续加上几层全连接或者LSTM等加深网络的深度,然后融入残差神经网络的架构防止梯度消失的情况,进行进一步分析以后得出的特征空间,可以融合再进一步去融合全剧特征与局部特征,可以更好地去显示出药物的潜在特征与其他药物之间的相似性,进而挖掘疾病对应药物中的相互关联。
(4)后端服务管理
另外,对于本次试验来说,还做了可视化处理,通过web前端React与web后端Springboot技术搭建了一整套模型的预测与展示界面
通过MVC的开发架构,合理去调度python模型的预测过程(预测阶段不会太慢,所以可以实时给前端进行返回结果),首先通过controller接收到请求,然后调用相应的service进行整合服务,service去调用对应的python代码,最终跑出来的结果通过entity实体类返回给对应的接口,封装成结果的data传回前端
后端服务管理:用Springboot写了一套管理python代码预测调用,并部署到阿里云服务器上面。
使用MVC的构架,有利于后期的拓展与维护,这样可以有效加强系统的健壮性。
系统截图
python模型端:

后端:
数据量
数据量大小为20000个drug与其他相关信息的共同关系组,但是药物与疾病的总类都有一定的限制性,不能很好发挥模型的最大性能。
每一次进行predict的时候建议将预测结果选择前20个进行呈现,因为前20个是比较有可能被选中的药物,差不多置信度都能达到百分之八九十,超过前二十的部分的数据可能没有很高的置信度,甚至仅仅为0.001的级别。可能是在数据集中构建成meta-path的时候仅仅在某一环中出现了一次,没有引起注意力机制很大的权重分配。
性能评价
对于模型的性能,在目前通过爬虫与其他脚本构造出来的数据集来说,因为在构造数据集时,随机抹去了很多药物的关联此数据作为x,被抹除的药物作为y,从而使得药物在预测时能够保证有准确的回归过程,通过50epoch的训练,可以达到precision: 0.74,accuracy:0.75的水平。看了一下对应从谷歌学术上面检索到的论文的精确度,还是有一点差距,但是在模型深度方面,如果再叠加更深的模型的话,应该可以有更深的提升,并且在以后的参数量细化方面,从这个角度,可以组成一个新的transformer的新架构,或许可以在性能上面有更大的提升。






















 3443
3443











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








