文章题目
原文链接:
https://elifesciences.org/articles/48994
Self-assembling manifolds in single-cell RNA sequencing data
文章目的
这篇文章主要是想实现细胞降维聚类
We present the self-assembling manifold (SAM) algorithm, an iterative soft feature selection strategy to quantify gene relevance and improve dimensionality reduction. We demonstrate its advantages over other state-of-the-art methods with experimental validation in identifying novel stem cell populations of Schistosoma mansoni, a prevalent parasite that infects hundreds of millions of people.
这个是它摘要中的一段话,使用软feature selection的策略来定量基因之间的相关性和降维。并且它能够找到其他方法找不到的cell type。降维和聚类是紧密联系再一起的。
算法实现
计算gene weigth
- 首先我们输入这个软件的应该是一个行是cell,列是gene的matrix
- 然后得到knn graph matrix, 进行矩阵乘法。得到新的gene expression 的matrix。
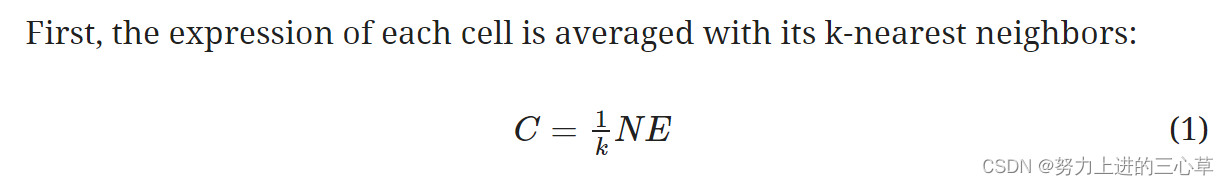
- 求每一个基因的平均值和方差,还有Fano factor, Fano factor就是方差/平均值,它能够表示出数据的离散程度,并且忽略平均值的影响。这就是为了防止某些表达很高的基因在后面的计算过程中占很重要的作用。作者想让在不同细胞中表达不同的基因占很重要的作用,而不是表达高的基因占重要作用。
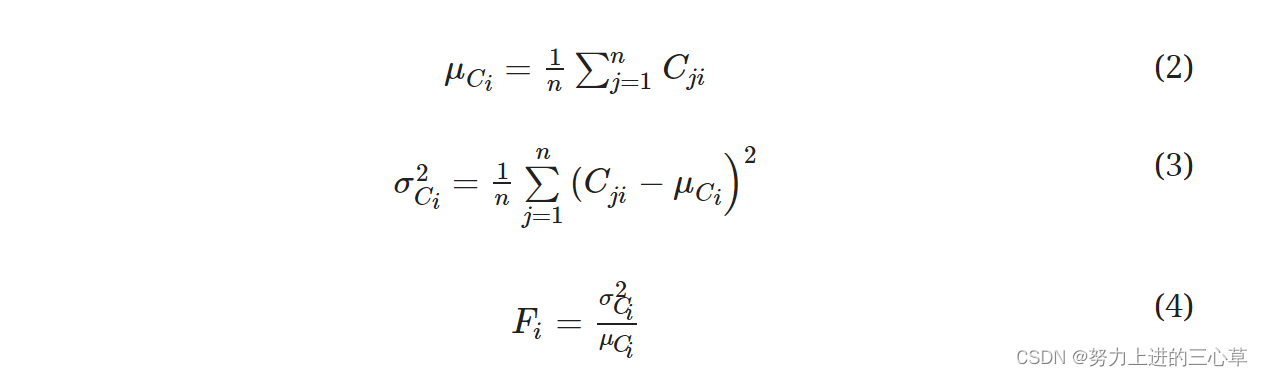
接着对得到的F进行归一化,也就是让F最后分布在0到1之间,作者做这件事的方法是:它选择了F值最大的50个值,然后求平均值(z),然后如果F值大于z的话,就让它等于z,最后拿每一个F值都除以z,这样的话,最后的值就可以在[0,1]之间了。
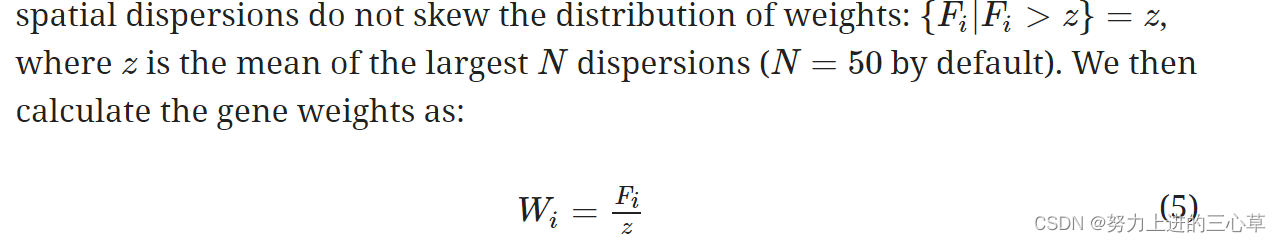
rescale 表达矩阵
- 首先对最开始用户输入的表达矩阵E进行标准化,得到E拔。
- 对标准化之后的矩阵,每一列基因都乘以这个基因相应的W值。(这里是对角矩阵和一个正常矩阵的乘法)
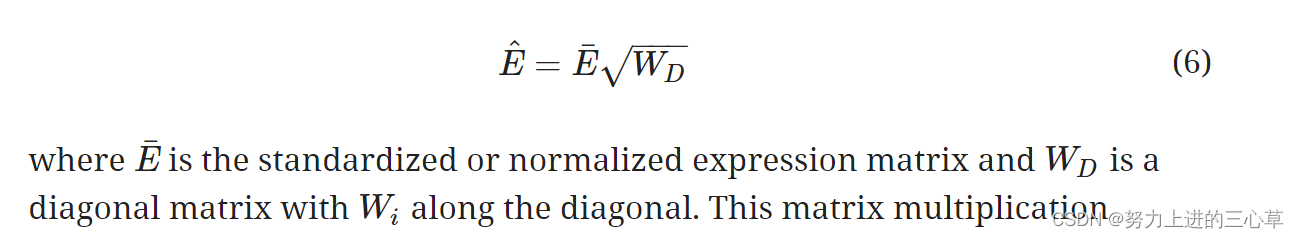
更新knn graph
- 使用PCA进行降维
PCA进行降维的标准操作步骤,第一步是进行去中心化,也就是按列,先分别求出每一个基因的平均值,然后用每个细胞中的该基因值-这个平均值。
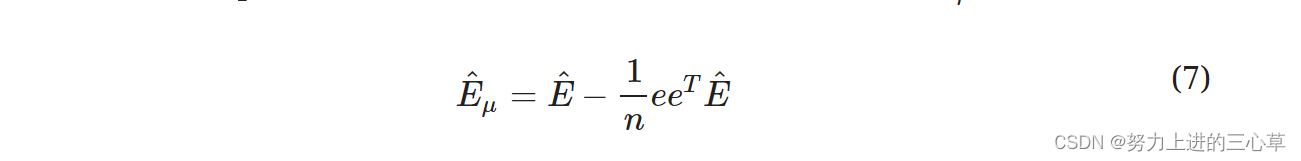
对表达矩阵进行PCA,实际就是得到表达矩阵的协方差矩阵的PC和特征值。协方差矩阵的PC和特征值其实就是我们PCA最后要求的。
具体的推到过程如下 (注意,下面有一个错误:我们得到的S2/(n-1)是奇异值,而我们最终要得到特征值,而特征值是奇异值开根号。 √ 就是单纯的根号,给对角线上的值开根号,没别的意思。

接着作者又对新得到的PC矩阵进行scale, 用特征值乘以相应的PC。也就是想让不那么重要的PC值的权重变小一些,因为作者要用所有的PC来计算细胞之间的距离,所以这一步是重要的。平时我们只取前面几个PC是因为PCA已经根据特征值的大小对我们的PC进行了排序,所以不用乘以特征值。
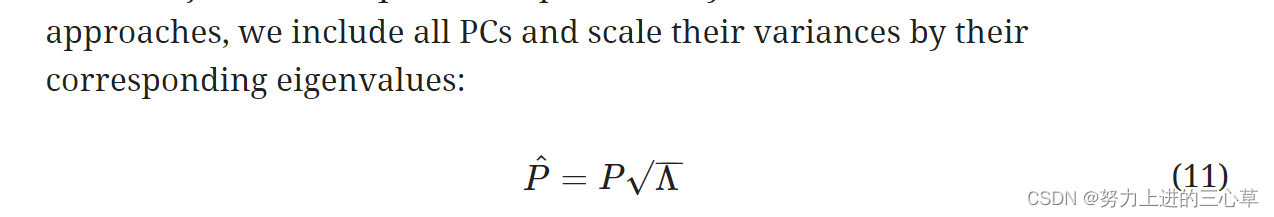
- 根据新得到的PC矩阵计算细胞与细胞之间的距离 (欧式距离,或者Pearson’s correlation distance)
- 使用新得到的距离定义每一个细胞的邻居,得到新的kNN matrix
- 最后迭代上面的过程,直到收敛,收敛的标准为:这一轮得到的基因的weight值和上一轮的基因的weight值的差 (所有基因的差的和)小于5x10-3

启发
总的来说,这篇文章的整个思路还是比较简单的。我们给机器输入一个cellxgene的matrix,我们最终的目的就是想根据每一个基因在细胞中的表达情况把同一类的细胞聚在一起。那当然是表达谱越相同的细胞越应该聚在一起。但出现了一个问题,就是这些基因太多了,我们不可能参考所有基因的在不同细胞中的表达情况吧,因此我们就想到了先降维再聚类。所以基本的框架就是先用PCA降维,然后用kNN聚类。但是作者的创新点在于
- 它想通过x gene weight来校正最开始的gene expression matrix(如果在不同的细胞中,某一个基因的表达值差异很大,那么这个值就大,如果在不同的细胞中,某一个基因的表达值差异很小,那么这个值就小)
- 它在进行PCA降维之后,并没有选择前几个PC,它用这些PCx相应的特征值,从而使用了所有的PC来进行后面的kNN聚类求细胞之间的距离。
但是这样运算难道不会很慢吗?
总之这应该是自己完全看懂公式和思路的第一篇方法的文章。学到了很多与矩阵有关系的知识。希望自己继续加油!
在组会上讲了这篇文章,尽管英文水平还有待提高,presentation的水平也有待提高。但是我在慢慢进步啊。使用的ppt在这里,更加详细的画图展示了整个过程。
把ppt放到这里了,可以作为参考
https://cowtransfer.com/s/c4c7a0424f2b40 点击链接查看 [ elife_SAM算法.pptx ] ,或访问奶牛快传 cowtransfer.com 输入传输口令 g5zu77 查看;
参考:
https://builtin.com/data-science/step-step-explanation-principal-component-analysis
https://zhuanlan.zhihu.com/p/331930951
https://blog.clairvoyantsoft.com/eigen-decomposition-and-pca-c50f4ca15501
https://www.samlau.me/test-textbook/ch/19/pca_svd.html
https://stats.stackexchange.com/questions/134282/relationship-between-svd-and-pca-how-to-use-svd-to-perform-pca (最有价值)






















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








