Title
3D CNN-BASED SOMA SEGMENTATION FROM BRAIN IMAGES AT SINGLE-NEURON RESOLUTION
基于3D-CNN从单细胞分辨率脑图像中分割细胞体
发表信息:ICIP 2018(International Conference on Image Processing)
Author: 中国科学技术大学 刘东*和毕国强团队
Abstract
- 意义:神经细胞分割对于大体脑图像的自动分析来说是一项很重要的任务。
- 以前的方法:以前的神经细胞分割方法依赖于手工提取图像特征,但是在解决高分辨率,低信噪比的脑图像上存在困难。
- CNN在自然图像分割上取得了显著成功,但是CNN需要准确的标注数据用于训练,这对大体积的脑图像来说是很难达到的。
- 本文的方法:提出了一种弱监督学习策略来解决训练数据不准确的问题,并采用3D-CNN对脑图像进行自动的细胞体分割。
- 结果:在我们自己收集的单细胞分辨率脑图像上测试了我们的方法,结果显示:基于3D-CNN的方法显著优于传统方法。
1. Introduction
Para1:开发高分辨率脑图像自动分析算法成为当务之急
- 生物显微成像技术的发展使得单细胞分辨率的脑成像成为可能,极大地促进了对脑神经形态和结构的观察。例如:我们现在能够收集物理分辨率为0.5m^3/vox的小鼠大脑图像,这可以清楚地识别每个神经元。
- 由于它们的高分辨率,这样的大脑图像体积巨大(在我们的设置下,一只老鼠的大脑产生大约3 TB的图像)。手动观察和处理这些图像是不可能的。
Para2:神经细胞分割是一项被研究已久的重要任务,但仍然是神经科学领域的一大挑战
- 重要性:从脑图像中分割神经元是一项重要的分析任务,是神经元形态分析、神经元计数、脑功能图谱等诸多后续任务的实现技术。
- 挑战性:不同脑功能区的神经元在大小、形态、强度或密度上存在很大的差异;
成像过程可能会引入畸变或噪声,从而产生低信噪比的图像;
以往的成像技术只能产生神经元无法分辨的低分辨率图像。
以往的研究多采用手工制作的图像特征进行分割,而对于差异较大的脑图像,这些特征并不具有鲁棒性。
Para3:考虑将CNN应用于脑图像上
- CNN的成功:CNN在自然图像方面取得了显著的成功,在生物医学图像中也取得了一定的成功
- 应用CNN的困难:使用CNN进行大脑图像处理,有一个显著的不同:CNN通常需要大量准确标记的数据来训练,但是手动标记大脑图像中的神经元既费时又费力。
Para4:本文采用的方法和取得的结果
- 提出了一种弱监督学习策略(从自己收集的小鼠大脑图像中构建了一个弱标释的数据集)来解决与CNN相关的训练数据问题
- 采用3D CNN结构,即深度监督网络(deep Supervised Network, DSN)对大脑图像进行细胞体分割
- 结果表明,我们的方法明显优于以前的方法(NGPS),消融研究验证了弱监督学习策略
2. Related work: 神经元检测或分割的方法
Para1: 传统方法
- 基于传统的图像处理算法,如分水岭算法、能量函数法、均值漂移聚类算法等
- 传统算法依赖于手工制作特征和精心调参,通常在高信噪比的情况下才能表现良好。
Para2: CNN的发展
- 近年来,CNN在自然图像的分类、分割和检测方面取得巨大成功。
- CNN在自动学习最具鉴别性的特征方面具有优势,特别是在低信噪比的情况下。
Para3: CNN应用于生物医学图像分析:针对三维图像,提出了一系列的深度学习模型
- 基于3D-CNN的脑微出血检测、基于CNN的神经元分割、结合LSTM的联合序列学习进行分割、基于3D自编码器对细胞组织进行分割等
- MICCAI 2017中,有两种3D全卷积结构,即Deeply Supervised Network (DSN) 和DenseVoxNet, 性能都可以与最先进的方法相媲美甚至更好。
- 在本文中,我们采用了DSN的网络结构来解决胞体分割问题,并设计了一种新的有效的训练策略
注:MICCAI是由国际医学图像计算和计算机辅助干预协会(Medical Image Computing and Computer Assisted Intervention Society) 举办,跨医学影像计算(MIC)和计算机辅助介入 (CAI) 两个领域的综合性学术会议,是该领域的顶级会议
3. Proposed Method

Figure 2 shows an example 3D patch, where the voxels inside the white sphere are foreground, and the others inside the yellow cube are regarded ambiguous.
3.1 Training Data
- 以前基于CNN的图像分割:像素级的标注(胞体大小、形状和密度不同,这样标注耗时耗力)。
- 本文的策略:简化标注。将每个soma标记为一个球体,即每个soma与它的中心(三维坐标)和直径的标签相关联。
- 这样做的问题:由于标注不准确,如果将球体内部的所有体素设置为前景(soma),将球体外部的体素设置为背景(non-soma),那么CNN在训练的时候可能会被误导。特别是somas边界的体素无法被CNN正确识别。
- 改进:将标签转化成体素级的标注。如果一个体素在一个球体中,那么它属于前景; 如果它离任何球体足够远,它就属于背景;否则,它是不明确的。对于不明确的体素,在训练过程中“不关心”分割结果是否正确,即在计算损失时将这些体素排除在外。
3.2. Network Structure Analysis
2D-CNN
- 以往的研究也使用CNN对体积数据进行生物医学分割问题的研究,但其模型基于2D-CNN,分别从每张图像中提取特征,然后进行融合。
- 2D-CNN可能无法充分利用图像之间的三维信息和关系。
3D-CNN
- 根据我们的经验,由于somas的大小、形态和强度不同,我们需要在不同的3D图像视图中区分somas。
- 在三维上提取特征对体积图像更有益。因此,我们采用了3D-CNN: DSN。
DSN
- DSN包括卷积层、池化层、反卷积层,全部采用3D 形式。
- 卷积层和池化层作为特征提取器,反卷积层和softmax层将特征立方体上采样到与输入相同的大小.
- 在DSN中,为了避免梯度消失,在较浅的层上还有两个分支。
3.3. Training Strategy
Para1: 内存限制
- 问题:训练3D CNN面临内存限制(特征立方体相对于输入尺寸是巨大的)
- 方法:将3D脑图像裁剪成有冗余的小立方体,使用尽可能大的立方体避免将一个soma分割到两个立方体。训练时,batch size 设为1。
Para2:胞体稀疏性,数据不平衡
- 问题:脑图像中的soma十分稀疏,非soma区域要大得多,导致数据不平衡问题,影响CNN的训练效果。
- 使用数据平衡技术:对于每个立方体,随机选择一定比例的背景体素作为non-soma,其余的背景体素标记为“do not care”,即不考虑计算损失。此外,只有那些包含超过2000个soma体素的立方体才作为训练数据保存。
Para3: 数据扩增
- 问题:成像过程,数据集中的3D图像在不同维度上并不一致。特别是,在一个维度上体素插值过,而在其他维度上没有。
- 方法:对数据集进行随机翻转
4. Experiments
4.1 Implementation
Para1: 训练集和测试集处理
- 原始体积数据:512*869*1024, 16bit。手工标注soma:数量查过7000个,半径从小于4um到30um不等。
- 将原始数据裁剪成191个160*160*160的立方体(30um的冗余)。随机选择52个立方体用于测试,剩下的用于训练。
- 为了平衡soma和non-soma体素的比例,随机保留non-soma 体素使其数量是soma 的十倍,其余的背景体素在训练中不考虑。
- 常见的预处理,即对图像减去均值除以标准差。
Para2: 模型设置
- DSN模型由Caffe及其matlab接口实现。所有实验都在GeForce GTX 1080Ti上完成的。
- 随机初始化:均值为0,方差为0.01的高斯分布。优化方法:随机梯度下降法。基准学习率:0.001,并根据“poly policy”下降(第iter次迭代时,学习率乘以(1-iter/max_iter)^power, max_iter=15000,power=0.9)
4.2 Test Protocal
- 使用经过15,000次迭代训练的最终模型来测试DSN。
- 每个测试图像也被裁剪成与训练过程中相同的大小。通过对重叠区域进行简单的平均,对立方体的分割结果进行融合。简单的后处理来移除那些太小的检测区域,因为它们不太可能是somas。
- 测试NGPS: 所有测试图像的测试参数相同:二值化阈值为6.0,最小半径为4.0。这些参数都经过了仔细的调整,以确保NGPS在该数据上获得最佳结果。NGPS检测somas为球体,因此输出每个检测到的soma的中心和半径
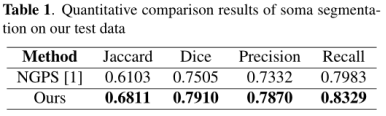
- 使用四个常用的度量来定量地评估提出来的方法和NGPS的性能:Jaccard、Dice、Precision和Recall
4.3 Results
where R denotes segmentation result and G denotes ground truth. It is worth noting that the ground truth is also labeled with the sphere approximation, i.e. each soma is recorded as a sphere.
Fig. 3. Visual comparison of the segmentation results. From top to bottom: the original image with ground-truth labels, each soma is denoted by a white sphere, yellow circles indicate some difficult cases; the segmentation result by our method; the segmentation result by NGPS. In the segmentation results, green circles indicate false positive, yellow circles indicate false negative (i.e. missing).
- 两种方法的定量结果如表1所示。可以看出,我们的方法明显优于NGPS。
- 对分割结果进行了可视化检查。图3显示了一些示例。我们可以看到,我们的方法在稀疏和稠密情况下都优于NGPS。
- 例如(a)和(b)中,胞体的密度太大,难以识别,但我们仍然可以观察到我们的模型分割出的胞体更接近于ground truth。(a)在NGPS的结果中存在一些误报。在(c)、(d)和(e)中,胞体非常稀疏,NGPS很难在边界或低信噪比区域检测胞体。NGPS可能将轴突、树突或其他组织误认为是胞体,从而导致假阳性。相反,我们的方法在这种情况下可以很好地工作。
4.4 Ablation Study
- 为了验证我们提出的训练策略的有效性,我们进行了消融研究,结果如表2所示。
- 平衡是指是否进行数据平衡,即在训练过程中控制非soma体素的数量。转置是指是否进行随机转置作为数据扩充。
- 从结果中我们可以看到,这两种操作都提高了我们的模型在Jaccard和Dice方面的性能。值得注意的是,当不使用数据平衡时,由于CNN倾向于将体素识别为non-soma并避免假阳性,因此准确率可能会非常高,但召回率会非常低。因此,使用数据平衡可以更好地权衡精确性和召回率,从而获得更好的性能。
5. Conclusion
- 在这篇文章中,我们提出了一种基于3D-CNN的方法来对大脑图像进行高分辨率的胞体分割。
- 将采集到的数据用简化的标签标注为弱监督,实验证明经过训练的CNN仍然可以预测胞体的具体表面。这种标注方式节省了大量的劳动和时间。与传统的NGPS方法相比,我们的模型在数据集上的表现明显更好。
- 该方法可用于神经元计数、脑功能定位、神经元重建等。
- 未来的工作将研究用于胞体分割的其他网络结构,并将重点放在困难的密集胞体案例上。







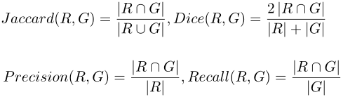















 2万+
2万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








