目录
一、引言
在机器学习和数据科学的领域中,支持向量机(Support Vector Machine, SVM)是一种广泛使用的分类算法。其独特的原理和强大的性能使得SVM在诸多领域中都有出色的表现。然而,对于初学者而言,SVM的复杂性和抽象性往往让人难以捉摸。本文将通过数据可视化的方式,深入浅出地解析SVM的工作原理和应用,帮助读者更好地理解并掌握这一强大的分类工具。
二、支持向量机的基本原理
SVM的核心思想是通过找到一个决策超平面,将不同类别的数据分隔开来。这个决策超平面不仅要能将数据分开,还要使得数据点到超平面的距离最大化,即所谓的“最大间隔”。这样,SVM就能够对新的数据点进行准确的分类。
在二维空间中,这个决策超平面就是一条直线;在三维空间中,它是一个平面;而在更高维度的空间中,则是一个超平面。为了找到这个最优的超平面,SVM引入了“支持向量”的概念。支持向量是那些距离决策超平面最近的数据点,它们决定了超平面的位置和方向。
三、数据可视化在SVM中的应用
数据可视化是一种强大的工具,它可以帮助我们直观地理解SVM的工作原理。通过绘制数据点和决策超平面的图形,我们可以清晰地看到SVM是如何将数据分开的。
四、SVM的数据可视化绘图方法
1.sin正弦函数绘制
这里采用Matplotlib模块来对数据可视化,用pip install matplotlib 命令安装该模块,先绘制一个sin函数图形。
from pylab import *
import numpy as np
X = np. Linspace(-np. pi, np. pi, 256,endpoint=True)
S = np. sin(X)
#产生绘图数据X,S,为x轴和y轴一一对应的数据
plot ( X,S)
#以默认的形式绘制数据,plot 第1个参数为x轴坐标数据,第2个参数为y轴坐标数据
show ( )运行结果如图:

plot(X,S)为画图函数,X为x轴坐标数组,S为对应的y轴坐标数组,用默认的方法画出图形。
show()函数把前面用plot函数绘制的图像显示到屏幕上。
2.实例化默认值
(1)改变颜色和线宽
(2)设定限定
(3)设置坐标刻度和坐标标签
(4)移动脊柱
(5)注释要点和添加图例
3.图的布局
首先尝试一下图的布局。从以下代码就能明白 subplot 函数中3个参数的含义:(2,1,1)的意思是图的分布是2行1列,而本次绘制占用第一个位置
from pylab import * subplot (2,1,1)
#产生两行一列的子图,先设置第1个子图
xticks([]) , yticks([])
text (0. 5,0. 5, 'subplot (2,1,1)', ha='center', va='center', size = 24, alpha = . 5)
subplot ( 2,1,2)
#设置第2个子图
xticks []) , yticks [ ])
text (0. 5,0. 5, 'subplot (2,1,2)', ha='center', va='center', size = 24, alpha = . 5)
show ()这里用subplot函数绘制参数子图,subplot(2,2,1)表示产生一个两行两列的4个子图,现在对第一个子图操作。
效果如图下:
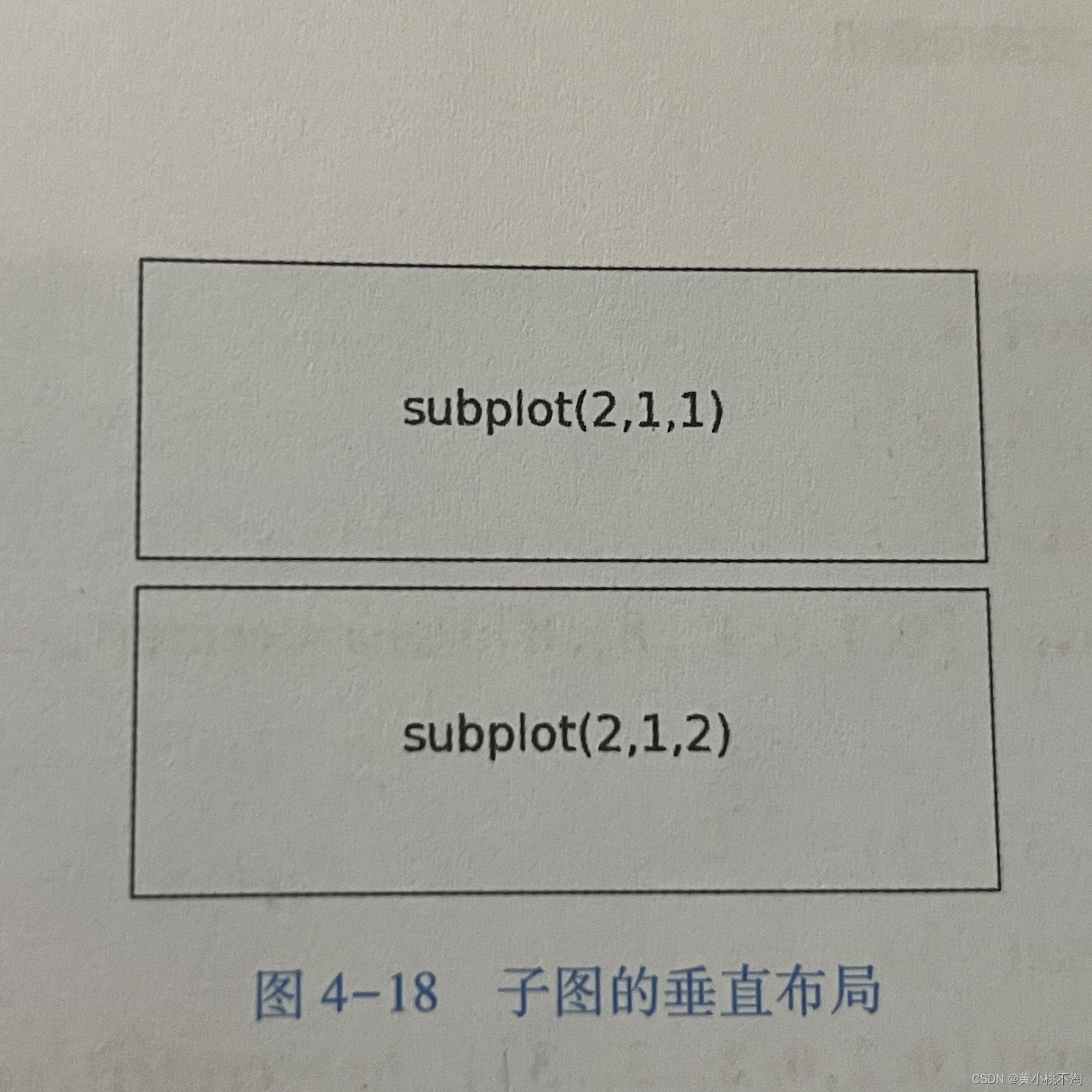
text (0. 5,0. 5, 'subplot(2,1,1)', ha='center', va= 'center', size = 24, alpha =. 5), 函数是绘制文本,前2个参数为坐标,第3个参数为绘制的内容。
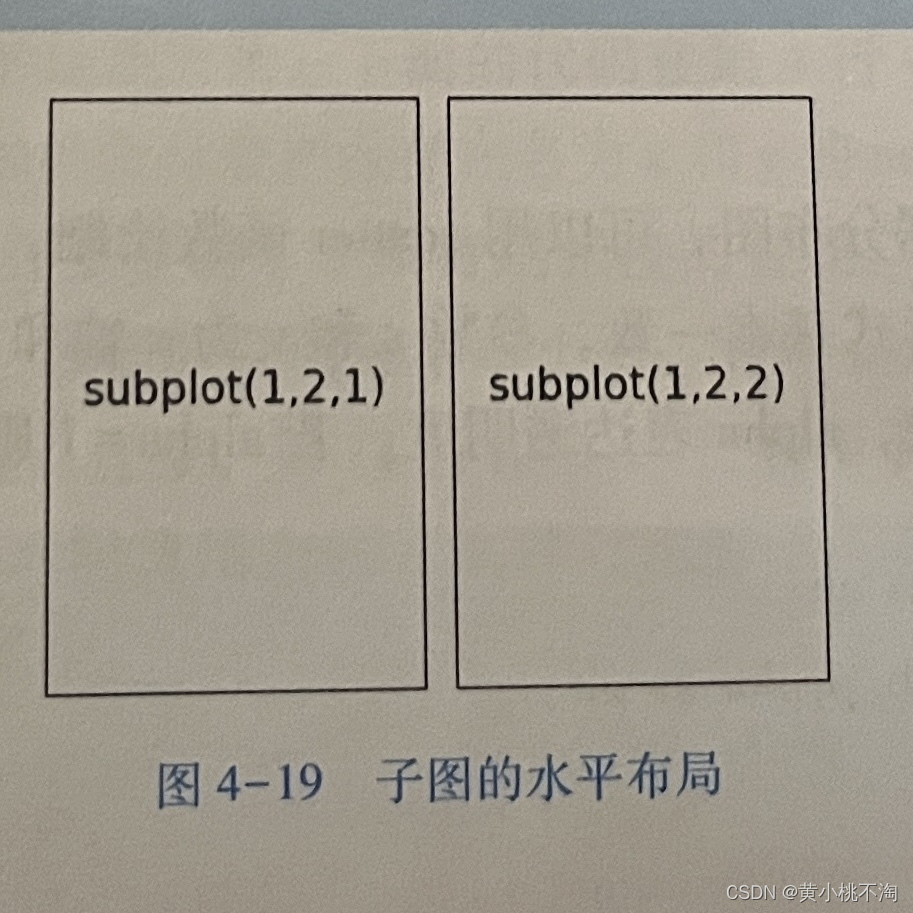
与此相似,以下代码是制作1行2列的图
from pylab import *
subplot (1,2,1)
#产生一行两列的子图,设置第1个子图,第1个参数为多少行,第2个参数为多少列,第3个为当前子图
xticks ([]) , yticks ( [])
text (0. 5,0. 5, 'subplot (1,2,1)',ha='center', va= 'center', size = 24, alpha =. 5)
subplot ( 1,2,2)
xticks ([]) , yticks ( [] )
text(0.5,0.5, subplot(1,2,2)',ha='center, va='center,size =24,alpha=.5)
show ()运行结果图下
4.坐标轴
坐标轴和子图功能类似,不过它可以放在图像的任意位置。
from pylab import *
axes ( [0. 1,0. 1,8,.8])
xticks([]) , yticks([])
text (0. 6,0. 6, 'axes ( [0. 1,0. 1,08,8] )',
', ha='center', va= 'center', size = 20 , alpha
=.5)
axes ( [0. 2,0. 2,.3,.3])
xticks([]), yticks ([])
text (0. 5,0. 5,'axes ( [0. 2,0. 2,•3, 3] )',ha='center', va ='center', size = 16, alpha
=.5)
show ()效果如下:

5.散点图
散点图是最常见数据分布图,可以用 scalter函数绘制,该函数需要引用 matplotio
库,与绘图函数的调用形式基本一致,参数x和y为轴和y轴的坐标向量,s和c为形状大小参数和颜色参数,alpha 表达透明度,若 alpha=1 则表示完全不透明。
import numpy as np
import matplotlib. pyplot as plt
import time
np. random. seed (int (time. time() ))
n = 128
X = np. random. normal (0,1,n)
Y = np. random. normal (0,1,n)
T = np. arctan2 (Y,X)
#产生绘制数据,T为颜色
plt. scatter (X, Y, s=75, c=T, alpha =. 5) plt. xlim (-1.5, 1. 5) , plt. xticks([])
plt. ylim (-1. 5,1.5) , plt. yticks ( [])
plt. show ()散点图效果如图下:
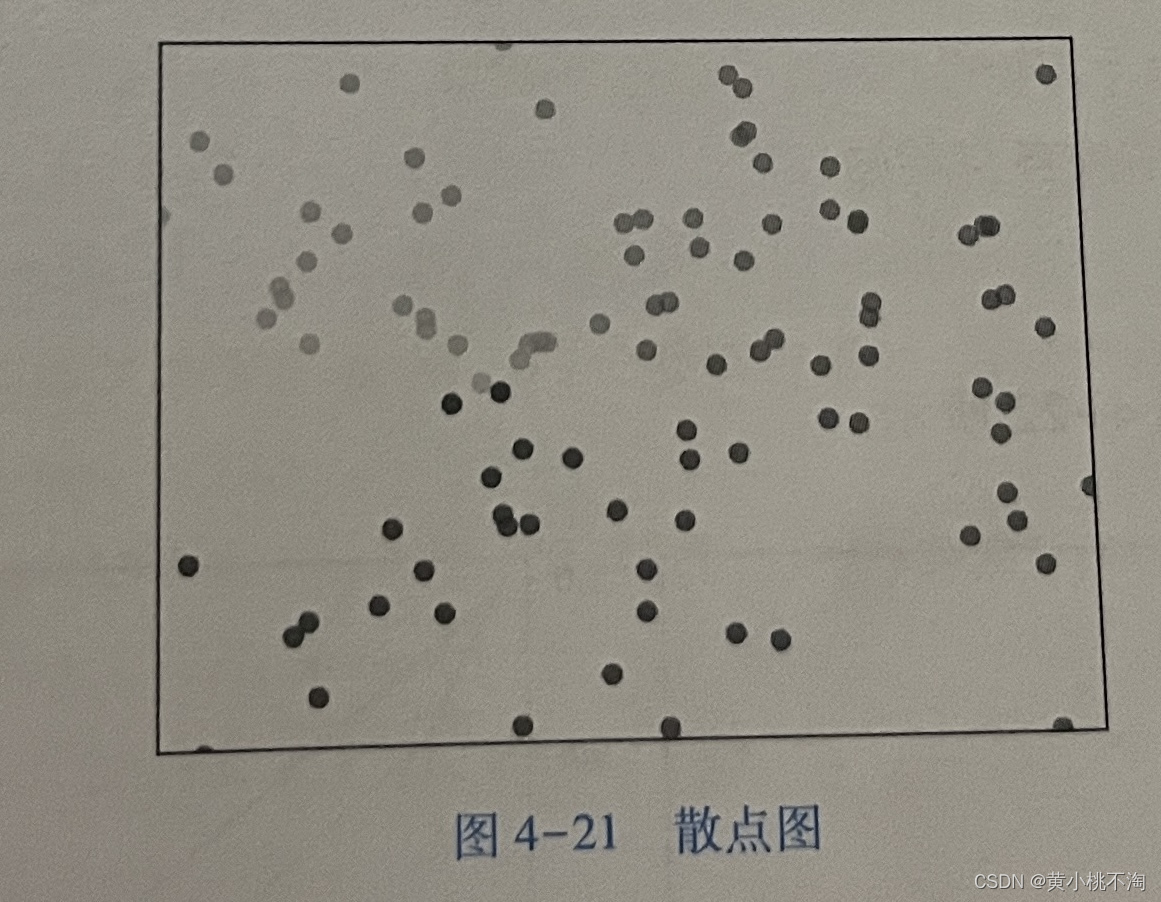
plt. scatter(X, Y,s=75,c=T,alpha=.5),scatter 函数用于绘制点,X和Y为要绘制点的对应数列,s为形状大小,°为颜色列表,alphe 值在0到1之间,默认是 None。
6.折线图
折线图也是一种常用的绘图方法。下面的代码在第4行~第7行建立了6个随机的点坐标,其中x轴和y轴的坐标分别保存在名为x和y的 numpy 数组中,然后在第8行~第12行用5种不同的线型绘制了5条首尾相接的直线。Pplot函数的主要参数是x轴和y轴坐标向量和线型,线型的说明见本章小结。利用 plot 绘图时可以传入一个完整向量,plot 方法会自动逐点连接,第13行和第14 行利用子图演示了该方法。另外,代码中给出了两个子图的不同使用方法,这种对比演示了在绘图方法上,subplot 与pyplot 具有同样的绘图性能。
import numpy as np
import matplotlib.pyplot as plt
import time
np.random.seed(int(time.time()))
N = 6
x = np. random. rand ( N)
y = np. random. rand( N)
plt. subplot ( 1,2,1). plot (x[0:2] ,y[0:2], "b--")
plt. subplot ( 1,2,1). plot (x[1:3] ,y[1:3], "go--")
plt. subplot (1,2,1). plot(x[2:4],y[2:4],”rー")
plt. subplot ( 1,2,1). plot (x[3:5] ,y[3:5],"cd-.")
plt. subplot ( 1,2,1). plot (x[4:6] ,y[4:6"m:")
plt_s2 = plt. subplot ( 1,2,2)
plt_s2. plot (x,y, "b-")
#在两个子图内各自绘画
plt.show()代码运行结果如图:
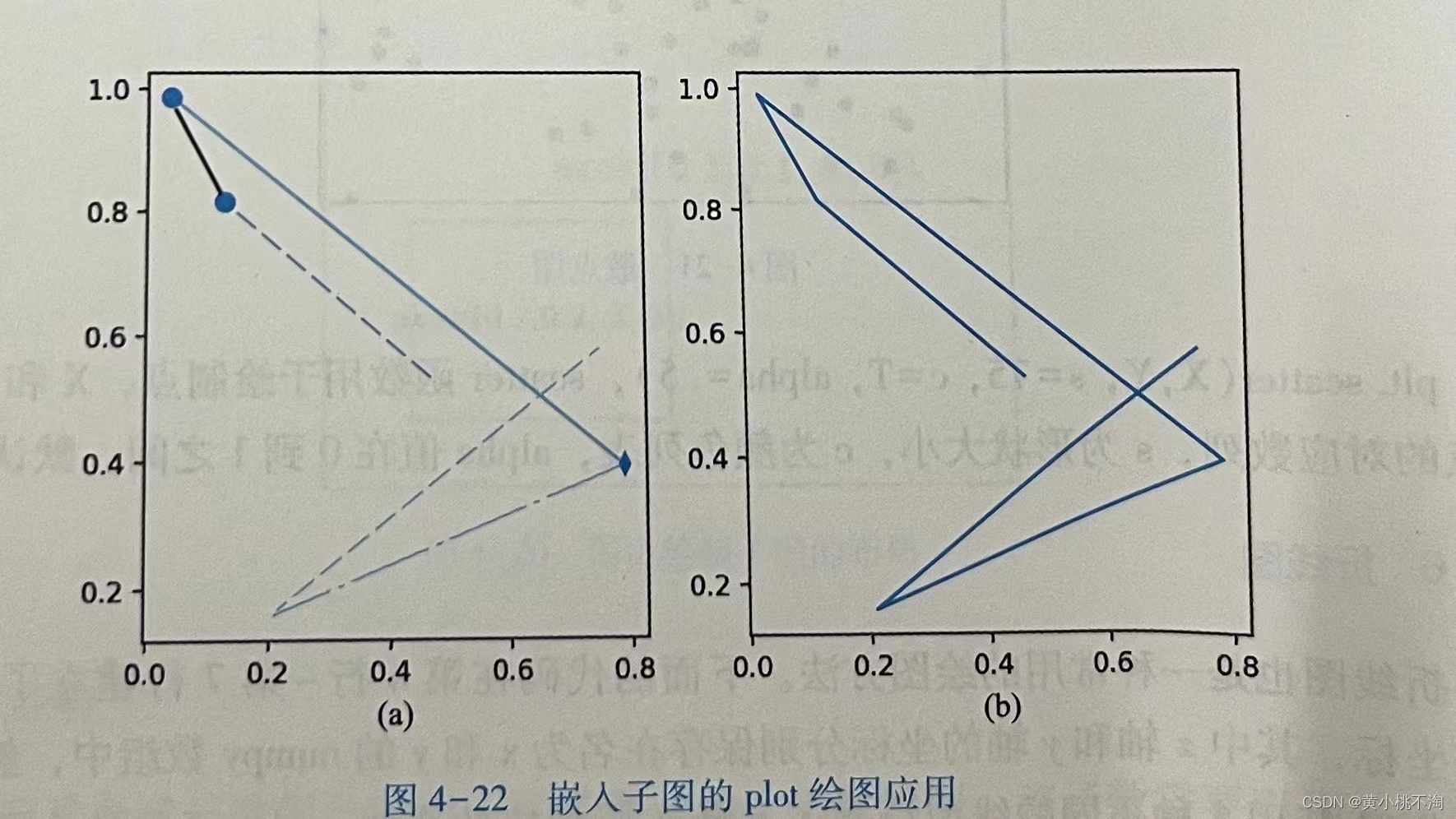
plt. subplot(1,2,1).plot(x[ 0:2],y[0:2],"b--")表示在1行2列的子图里操作第1个子图,并绘制一条线,线的颜色和线型采用了简写的方式,“b--”表示蓝色虚线.
五、总结
本文通过数据可视化的方式,深入浅出地解析了SVM的工作原理和应用。通过绘制数据点和决策超平面的图形,我们可以直观地看到SVM是如何将数据分开的,并理解其背后的原理。同时,我们还探讨了SVM的优缺点及应用场景,希望能够帮助读者更好地掌握这一强大的分类工具。
本文的文章链接:写文章-CSDN创作中心





















 6119
6119











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








