作为一名熟练的机器学习调包侠和调参侠,面临的一个很大的问题就是机器学习模型有很多超参数需要人工去调参,超参数的设置很大程度上影响着模型最后预测结果的好坏。特征工程和调参是机器学习算法运用中最耗费人工的2个部分。
那么如何利用遗传算法进行把调参变得“高大上”呢?这其实已经触碰到了时下最火热的AutoML(自动化机器学习)的领域了:自动化调参,不需要人工。
本文的案例将使用svm算法做iris鸢尾花分类,我们的任务就是优化SVM中的两个超参数C和Gamma。
这个问题的写法跟之前的单目标优化问题的写法很不一样,因为它不只是涉及决策变量C和Gamma,还涉及训练特征data_train。假设我们的预测值是:
那么我们的目标函数是最大化模型的预测结果的分类准确度:
一、geatpy数据结构
怎么利用geatpy做自动化调参呢?我们需要先了解geatpy的Phen、objV等数据结构长啥样,才能理解代码怎么写。当然这部分也可以不看直接看第二部分,但是会比较难理解第二部分中的实践代码。
1、种群染色体Chrom
Geatpy中,种群染色体是一个ap.array类型的二维矩阵,一般用Chrom命名,每一行对应一个个体的一条染色体。
我们一般把种群的规模(即种群的个体数)用Nind命名;把种群个体的染色体长度用Lind命名,则Chrom
的结构如下所示:

2、种群表现型Phen
种群表现型的数据结构跟种群染色体基本一致,也是numpy的array类型。我们一般用Phen来命名。是种群染色体矩阵Chrom经过解码后得到的基因表现型矩阵,每一行对应一个个体,每一列对应一个决策变量。若用N var表示变量的个数,则种群表现型矩阵Phen的结构如下图:

3、目标函数ObjV
目标函数是一个np.array,也就是每一个样本的分类结果的交叉验证得分。
Geatpy采用np.array类型矩阵来存储种群的目标函数值。一般命名为ObjV,每一行对应每一个个体,因此它拥有与Chrom相同的行数;每一列对应一个目标函数。

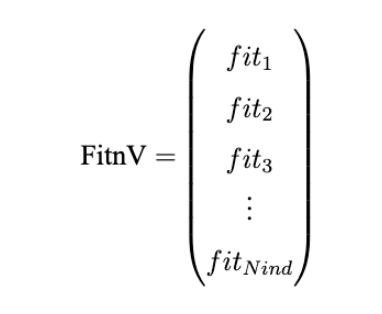
4、个体适应度FitnV
Geatpy采用列向量来存储种群个体适应度。一般命名为F itnV,它同样是np.array类型,每一行对应种群矩阵的每一个个体。因此它拥有与Chrom相同的行数。
5、违反约束程度CV
CV(Constraint Violation Value)来存储








 本文介绍了如何利用遗传算法(geatpy)自动调优sklearn的SVM模型,以提高鸢尾花分类的准确性。通过理解geatpy的数据结构,包括Chrom、Phen、ObjV等,并在实践中应用遗传算法,最终找到最优的C=31.07733和Gamma=0.0039,使训练集分类准确率达到97.78%,测试集达到96.67%。
本文介绍了如何利用遗传算法(geatpy)自动调优sklearn的SVM模型,以提高鸢尾花分类的准确性。通过理解geatpy的数据结构,包括Chrom、Phen、ObjV等,并在实践中应用遗传算法,最终找到最优的C=31.07733和Gamma=0.0039,使训练集分类准确率达到97.78%,测试集达到96.67%。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








