
今天给大家介绍的文章是“Tensor Decomposition with Relational Constraints for Predicting Multiple Types of MicroRNA-disease Associations”,这篇文章是华中农业大学章文教授团队的研究成果。该论文年初出现在arXiv网站,6月初被Briefings in Bioinformatics杂志接收。作者创新性地将miRNA-disease-type三元组表示为一个张量,引入张量分解的方法来预测多种类型的miRNA-disease的关联,并进一步提出了一种新的张量分解方法——关联约束张量分解法(TDRC)。实验证明了该方法与现有的两种张量分解法相比具有很好的性能和更高的效率。
1.研究背景
MicroRNA(miRNA)是一种小型的非编码RNA,在与人类疾病相关的多种生物学过程中发挥着重要作用, miRNA的异常表达也与多种人类疾病相关。识别与miRNA相关的潜在关联有助于理解miRNA相关疾病的分子机制,帮助开发新的疗法。大多数现有的计算方法主要预测是否存在miRNA-disease的关联,如已有研究使用随机游走和矩阵分解模型等来预测潜在关联的存在。从下图1中可以看出,miRNA与疾病的关联有多种类型,要研究与miRNA失调有关的疾病的发病机制,不仅要探究miRNA与疾病的相关性而且还需要了解其具体类型。张量是一个多维数组,三维张量通常用于三联体数据分析,如多关系网络、推荐系统和知识图谱等。一个miRNA-disease-type可以自然地建模为一个三元组,研究的目标是分解张量来探索一些未被观察到的三元组。张量分解是一种常用的张量补全方法,它将一个张量分解为几个小张量的乘积,从而得到它的逼近,可以很好地克服上述局限性。此外,现有的一些张量分解方法仅利用了关联信息,无法很好地深入捕捉miRNAs与疾病之间的相关性。
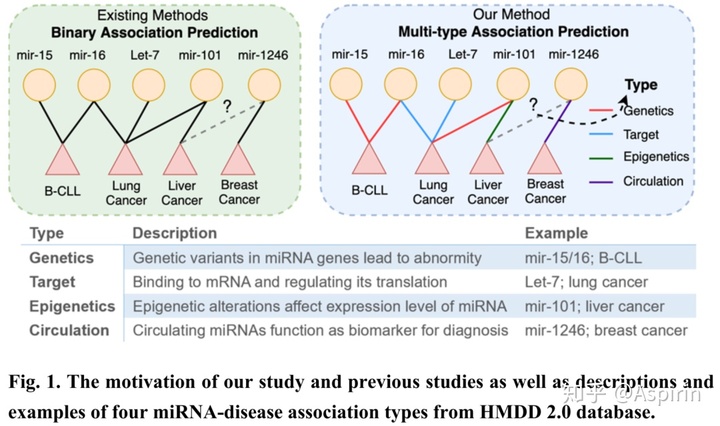
图1 以前的研究和该论文的研究对比
2.主要贡献
(1)引入张量分解方法来研究多型miRNA-disease关联预测任务,并与最近的基线方法相比取得了不错的改进;
(2)研究了现有张量分解方法在研究问题上的有效性,并提出了TDRC方法以整合辅助生物信息作为约束来进一步提高性能;
(3)利用乘子的交替方向法(ADMM)框架,为TDRC模型提供了一种高效的优化算法,并利用共轭梯度(CG)方法避免在ADMM内部迭代时计算逆矩阵,以降低时间复杂度;
(4)实验结果表明,该算法具有良好的鲁棒性和有效性。
3.方法
本节首先介绍了最常用的CANDECOMP/PARAFAC (CP)分解,然后描述了将生物辅助信息整合到CP分解框架中的TDRC方法,最后提出了一种求解TDRC目标函数的有效优化方法。
3.1 CP分解
CP分解是一种最常见的张量分解形式。已知miRNA-disease型张量
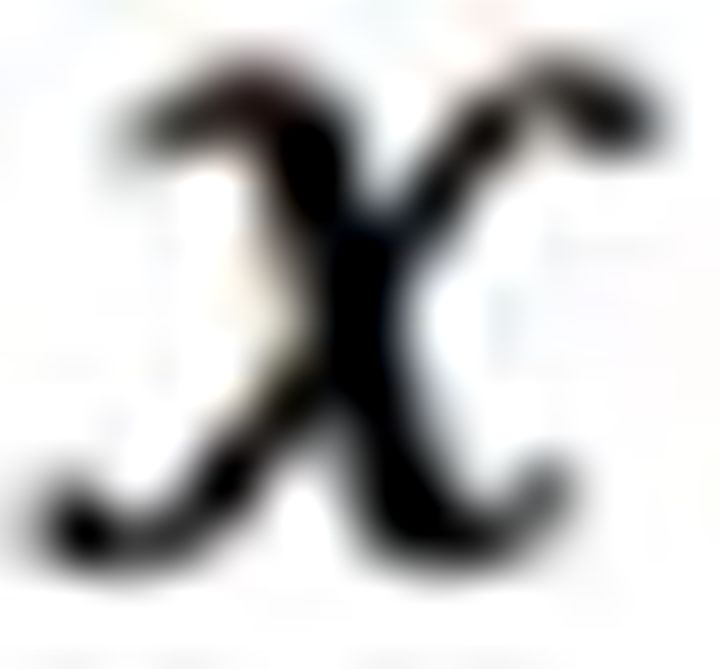
,CP分解模型可表示为以下优化问题:

其中,
表示张量的范数,C,P,F是相对于miRNA、疾病和类型模式的因子矩阵,通常被认为是对应模式的潜在表征,

是其的重构张量。
3.2 TDRC方法
标准的CP模型只利用关联信息。作者进一步提出关联约束张量分解法(TDRC)方法,将相似性作为约束纳入CP模型。TDRC的整个模型体系结构如图2所示。
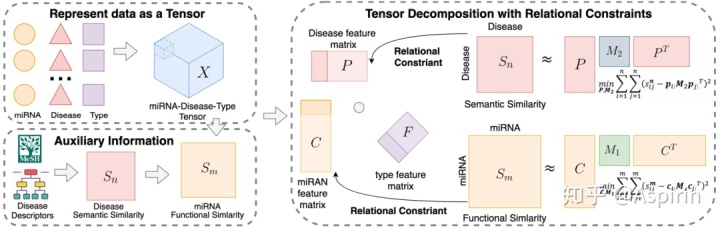
图2 用张量表示数据以及TDRC的模型体系结构引入投影矩阵和,分别将疾病(P)和类型模式(C)的因子矩阵,转换为疾病语义相似矩阵和miRNA-miRNA功能相似矩阵,根据正则化,得到TDRC的目标函数:

作者使用交替更新的方式来优化目标函数,会交替更新投影矩阵和,相似矩阵和,求解优化目标函数方法见算法1。

4.实验
4.1数据库
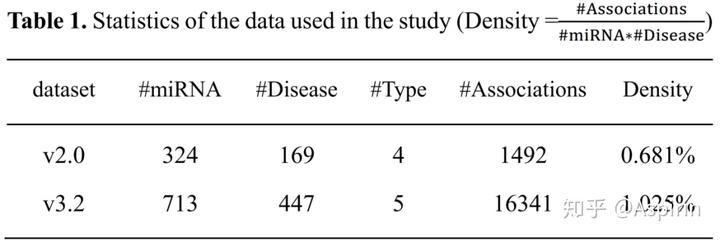
人类MiRNA疾病数据库(HMDD)是一个含有经过实验验证的人类miRNA-disease关联的数据库。HMDD v2.0根据遗传学、表观遗传学、循环和miRNA靶点相互作用的证据,这些关联被分为四种类型。HMDD v3.0已经发布,论文使用的是2019年3月27日发布的最新版本v3.2,包含了五种不同类型的miRNA-disease关联。为了获得稠密的数据,论文删除了所有类型中涉及总关联少于两种的miRNAs(疾病),获得的数据中对两个数据集的统计结果如表1所示。研究者同时下载了Medical Subject Heading数据库,包括了医学主题标题中的疾病描述,可用于计算疾病语义相似度。表1 本研究中使用的数据统计
4.2对比试验
为了全面研究模型在预测多种miRNA-disease相关性方面的表现,作者考虑了两种类型的10折交叉验证:和,选择了的方式,即将至少有一种关联的miRNA-disease对随机分为10个大小相等的集合,每轮使用一个子集进行测试,其余9个子集作为训练集。对于测试集中的每对miRNA-disease,都对其所有关联类型的预测进行排序,计算出最高的precision、recall和F1测度。选择NLPMMDA,两种张量分解方法CP和TFAI作为基线方法,实验对比结果如表2,可以看到在HMDD v3.2和HMDD v2.0上,TDRC的性能明显优于与其他方法相比有了明显的改进。表2 各种方法效果对比(1)

表2 各种方法效果对比(2)

作者对张量分解方法进行了时间效率分析,使用HMDD v3.2中的整个的张量和预先计算的相似性作为输入,在同样条件下做了对比试验,结果如表3所示,可以看到,TDRC相比其他张量分解方法具有高效性。表3不同张量分解方法的平均运行时间(20次)

作者进一步评估张量分解方法预测未观察到的miRNA-disease三元组的实际能力。在HMDD v2.0数据集中使用所有已知的四种miRNA-disease关联类型来构建所有模型,然后得到那些未知的miRNA-disease三元组的预测分数,对与特定疾病相关的miRNA配对进行排序,并从HMDD v3.2中找到了前20位预测的结果。图3为TDRC方法的15种选定疾病的结果,可以看到,对于某些疾病,前20名的预测精度不低于50%,表明张量分解方法在预测疾病相关miRNA及其相关类型方面具有很大的潜力。
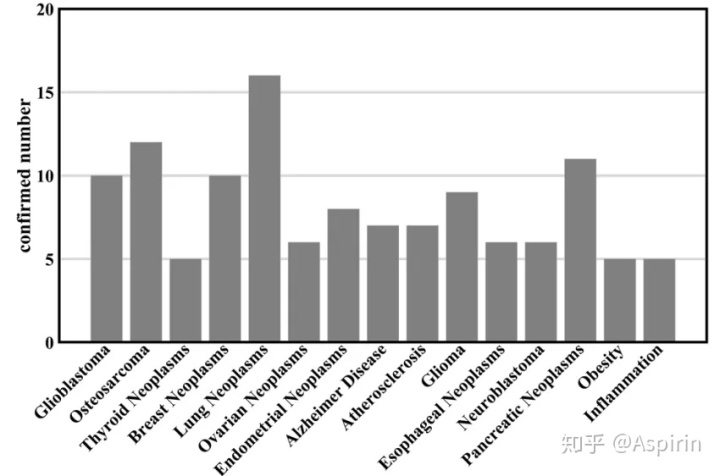
图3 TDRC基于HMDD v2.0对15种流行疾病的前20个预测中,在基于HMDD v3.2确认的miRNA-disease三元组的数量5总结预测多种miRNA-disease关联有助于理解与miRNA失调相关的人类疾病的发病机制。在这项研究中,该论文介绍了一系列张量分解方法来预测未观察到的miRNA-disease-type三元组关联。此外,还提出了一种新的基于张量分解的方法,称为TDRC,它将关系约束引入到张量分解模型中,集成了miRNA-miRNA相似性和疾病相似性。利用ADMM框架,作者提供了一种高效率的优化算法,并利用共轭梯度(CG)方法避免在ADMM内部迭代时计算逆矩阵,以降低时间复杂度。实验结果表明,张量分解方法优于基线方法,并且有较强的鲁棒性和较高的效率。在未来的工作中,将讨论更多张量分解形式,如Tucker分解,同时也适用于基于张量的模型,比如药物靶点-疾病三元关联和多关联的药物-药物相互作用。
参考资料
https://arxiv.org/abs/1911.05584








 华中农业大学章文教授团队利用张量分解方法研究多型miRNA-disease关联预测,提出TDRC方法,结合miRNA与疾病相似性约束,提高预测性能和效率。实验表明,TDRC在预测多种miRNA-disease关联上优于现有方法,具有良好的鲁棒性和效率。
华中农业大学章文教授团队利用张量分解方法研究多型miRNA-disease关联预测,提出TDRC方法,结合miRNA与疾病相似性约束,提高预测性能和效率。实验表明,TDRC在预测多种miRNA-disease关联上优于现有方法,具有良好的鲁棒性和效率。
















 1095
1095

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








