K-means聚类分析算法原理、改进及实现
对聚类算法简单了解可参考
https://zhuanlan.zhihu.com/p/43651469
K-means算法是一种无监督分类算法,将划分出来的类簇中点的均值当作该类簇的中心点(质心),其可以在不确定划分规则的前提下通过对数据集合不断迭代的方法对数据集进行划分,自动计算并更新每个类簇的中心点。
其思想可参考
https://www.cnblogs.com/txx120/p/11487674.html
sklearn库中含K-menas方法,python实现过程:
#-*- coding: utf-8 -*-
import pandas as pd
from sklearn.cluster import KMeans
#参数初始化
inputfile = 'E:\\arithmetic\\Kmeans.xlsx'
outputfile = 'E:\\arithmetic\\data_type.xls' #保存结果的文件名
k = 4 #聚类的类别
iteration = 500 #聚类最大循环次数
data = pd.read_excel(inputfile, index_col = 'CZLDWDM') #读取数据
#data_zs = 1.0*(data - data.mean())/data.std() #数据标准化
data_zs=data
model = KMeans(n_clusters = k, max_iter = iteration) #分为k类, 并发数4
model.fit(data_zs) #开始聚类
#简单打印结果
r1 = pd.Series(model.labels_).value_counts() #统计各个类别的数目
r2 = pd.DataFrame(model.cluster_centers_) #找出聚类中心
r = pd.concat([r2, r1], axis = 1) #横向连接(0是纵向), 得到聚类中心对应的类别下的数目
r.columns = list(data.columns) + [u'类别数目'] #重命名表头
print(r)
#详细输出原始数据及其类别
r = pd.concat([data, pd.Series(model.labels_, index = data.index)], axis = 1) #详细
# 输出每个样本对应的类别
r.columns = list(data.columns) + [u'聚类类别'] #重命名表头
r.to_excel(outputfile) #保存结果
print(r)
具有优点:可解释强,已于实现,适用于多维数据
缺点:需要提前确定k值;初始聚类中心选择是随机的,对后续结果影响较大。
针对上两个问题的改进方式
一、确定k值
目前较多的两种方式,主要是手肘法和轮廓系数法
(1)手肘法,利用样本聚类误差平方和SSE(sum of the squared errors)

其中,K是聚类数量,p是样本,m_k 是第k个聚类的中心点,SSE越大,则样本聚合程度越高。
其基本思想是:当k小于真实聚类数时,由于k的增大会大幅增加每个簇的聚合程度,故SSE的下降幅度会很大,而当k到达真实聚类数时,再增加k所得到的聚合程度回报会迅速变小,所以SSE的下降幅度会骤减,然后随着k值的继续增大而趋于平缓,这个最先趋于平缓的点就是合适的K值。由下图可看出,最佳k值应选择5。但该方法在实际应用中往往不是很如意。

sklearn库中含K-menas方法,其用python实现过程如下:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
#导入数据
df=pd.read_csv(r'E:\\小论文Arithmetic\\Kmeans-k.csv',header=None,encoding='gbk')
#df=pd.read_csv(r'E:\data analysis\test\cluster.txt',header=None,sep='\s+') #空格分隔符
print(df.head())
x=df.iloc[:,:-1] #iloc 按数据位置进行数据索引,获取行,列
y=df.iloc[:,-1] #按行索引,切片作为索引
#手肘法看k值
d=[]
for i in range(1,11): #k取值1~11,做kmeans聚类,看不同k值对应的簇内误差平方和
km=KMeans(n_clusters=i,init='k-means++',n_init=10,max_iter=300,random_state=0) #n_clusters要分成的簇数,init初始化质心, 种子点选取的第二种方法 计算该类中所有样本的平均值n_init选择质心种子次数 max_iter:每次迭代的最大次数 random_state: 随机生成器的种子 ,和初始化中心有关
km.fit(x) #开始聚类
d.append(km.inertia_) #inertia簇内误差平方和
plt.plot(range(1,11),d,marker='o')
plt.xlabel('number of clusters')
plt.ylabel('distortions')
plt.show()
更详细解释请参考:
https://www.cnblogs.com/kemaswill/archive/2013/01/26/2877434.html
(2)轮廓系数法
某个样本点x_i 的轮廓系数定义如下:

其中,a是x_i与同簇的其他样本的平均距离,称为凝聚度,b是x_i与最近簇中所有样本的平均距离,称为分离度。而最近簇的定义是:
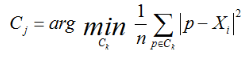
其中p是某个簇C_k中的样本。就是用x_i到某个簇所有样本平均距离作为衡量该点到该簇的距离后,选择离x_i最近的一个簇作为最近簇。
求出所有样本的轮廓系数后再求平均值就得到了平均轮廓系数。平均轮廓系数的取值范围为[-1,1],且簇内样本的距离越近,簇间样本距离越远,平均轮廓系数越大,聚类效果越好。
二、初始聚类中心选择
(1)、初始的聚类中心之间的相互距离要尽可能的远
K-means++算法
https://www.cnblogs.com/wang2825/articles/8696830.html
(2)、 选用层次聚类或者Canopy算法进行初始聚类,然后利用这些类簇的中心点作为KMeans算法初始类簇中心点。
另外,针对聚类算法最后的结果可进行评判
戴维森堡丁指数(DBI),其计算类内距离之和与类外距离之比,来优化k值的选择
具体公式理解可参考下面博客:
http://blog.sina.com.cn/s/blog_65c8baf901016flh.html
python实现过程:
import math
import pandas as pd
import os
import numpy as np
cluster_data = pd.read_csv(r'E:\\小论文Arithmetic\\DBI-cluster.csv',header=None,encoding='gbk')
cluster_data_array = np.array(cluster_data)
data_data = pd.read_csv(r'E:\\小论文Arithmetic\\DBI-data.csv',header=None,encoding='gbk')
data_data_array = np.array(data_data)
# print(cluster_data)
# print("---------------------------")
# print(cluster_data_array)
# 求两个点的欧式距离
def VD(v1, v2):
s = 0
for i in range(1, 3): #这个簇质心的维数为9啊,可改为3,相当于传过来的点也是数组形式
s += (v1[i] - v2[i]) ** 2
return s ** 0.5
# 求两个簇质心的距离
def Compute_decent(center1, center2):
return VD(center1, center2)
# 求簇i内任两点距离的和 这个计算,两两点直接应该没有重复
def Compute_Ci(x, clusterIndex):
Ci = 0
for i in range(x.shape[0] - 1): #应该是循环所有的点,除最后一个(j要用)
if x[i][0] == clusterIndex: #x[i][0]表示该点属于哪个簇,判断x[i]是否是要计算的簇
for j in range(i + 1, x.shape[0]):
if x[j][0] == clusterIndex: #x[j]也是该簇
Ci += VD(x[i], x[j]) #计算欧氏距离
return Ci
# 取Ci的均值
def Ci_avg(x, ClusterIndex):
clusterNum = 0
for i in range(x.shape[0]):
if x[i][0] == ClusterIndex:
clusterNum += 1
n = clusterNum * (clusterNum-1) / 2
return Compute_Ci(x, ClusterIndex) / n
# 计算max后面那个式子
def Compute_Mij(avgCi, avgCj, denominator):
numerator = avgCi + avgCj
if denominator == 0:
print('error! denominator is 0')
return numerator / denominator
# nc就是聚类数目,由于测试的数据量相对较大,用array把值存储下来,避免重复计算
def Compute_DBI(x, y, nc):
# s--final result;
# avgCi--1*nc list stores avgCi
# decentList--nc*nc list stores decent
s = 0
avgCi = np.zeros(nc) #返回一个行长度为12的数组,numpy.zeros(shape,dtype=float,order = 'C')给定形状和类型的新数组,用0填充 ,默认float 64;‘C’‘F’为行列顺序存储方式,默认为行
decentList = np.zeros((nc, nc)) #返回一个行和列长度都为12(可改为5)的数组,应该是记录两两质心点之间的欧氏距离
for i in range(nc):
for j in range(nc):
decentList[i][j] = Compute_decent(y[i], y[j]) #传过去两个质心点
print('decentList store over')
for i in range(nc):
avgCi[i] = Ci_avg(x, i + 1) #得到簇i内任两点距离的和,x是所有样本点数组,i是从0开始,而簇的种类是从1开始
print(avgCi[i])
print('avgCi', i + 1, 'store over')
for i in range(nc):
print('epoc is:', i + 1)
m = 0
for j in range(i + 1, nc):
temp = Compute_Mij(avgCi[i], avgCi[j], decentList[i][j])
if m < temp:
m = temp
print('epoc', i + 1, 'over')
s += m
return s / nc
print(Compute_DBI(data_data_array, cluster_data_array, 5))
























 1495
1495











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








