一 环境准备
项目地址
https://github.com/clara-parabricks/GenomeWorks
软件说明写的很清楚,要满足gcc,cmake,python,cuda的版本要求。
使用了docker构建了基础环境,镜像模板为centos7,gcc4.8.5,cuda10.2,cmake2.8。除了cuda满足了要求其他都不满足,经过一系列升级后,满足了版本要求。
#升级GCC
gcc 升级采用了源码安装,官网下载的太慢可以使用镜像网站下载。
gcc 下载地址http://ftp.gnu.org/gnu/gcc/gcc-7.2.0/gcc-7.2.0.tar.gz
安装过程可以参考下面的博客,类似的
https://blog.csdn.net/wangji163163/article/details/49305165?utm_source=blogxgwz0
与博客类似在下载 contrib/download_prerequisites的时候也遇到了国外网站访问太慢的问题,所以使用迅雷下载进行离线安装。
打开contrib/download_prerequisites找里面些的对应的版本去http://mirrors.nju.edu.cn/gnu/下载,进行手工安装后升级成功。要注意的是安装前置依赖时候,安装目录把版本号去掉。博客写的很清楚。
升级gcc成功#
#升级python版本
升级python打算使用conda进行python的版本管理,所以手贱把系统原生的python2.7卸载掉了。装上conda后base环境自带python3.7,但是yum命令用不了了,查询后发现yum依赖python2.7。开始想用conda创建一个python2.7的环境使用yum但是提示缺少包。最后没法办重装了yum和python2.7.
参考博客 https://blog.csdn.net/DawsonCheng/article/details/90515869
需要的包下载地址 http://mirrors.163.com/centos/7/os/x86_64/Packages/
python-2.7.5-34.el7.x86_64.rpm
python-chardet-2.2.1-1.el7_1.noarch.rpm
python-devel-2.7.5-34.el7.x86_64.rpm
python-iniparse-0.4-9.el7.noarch.rpm
python-kitchen-1.1.1-5.el7.noarch.rpm
python-libs-2.7.5-34.el7.x86_64.rpm
python-pycurl-7.19.0-17.el7.x86_64.rpm
python-setuptools-0.9.8-4.el7.noarch.rpm
python-urlgrabber-3.10-7.el7.noarch.rpm
rpm-python-4.11.3-17.el7.x86_64.rpm
yum-3.4.3-132.el7.centos.0.1.noarch.rpm
yum-metadata-parser-1.1.4-10.el7.x86_64.rpm
yum-plugin-aliases-1.1.31-34.el7.noarch.rpm
yum-plugin-fastestmirror-1.1.31-34.el7.noarch.rpm
yum-plugin-protectbase-1.1.31-34.el7.noarch.rpm
yum-updateonboot-1.1.31-34.el7.noarch.rpm
yum-utils-1.1.31-34.el7.noarch.rpm
将上面的包放到一个目录下 进入目录 执行 rpm -ivh --force *.rpm --nodeps
修复了yum后,conda依然不影响使用base环境是3.7.python升级完毕#
其他的工具升级比较简单,百度以下很多资料不再赘述。
二 按照项目说明进行CMAKE过程出现的问题
1.缺少htslib,这个库是用来读取一些生物信息领域特殊文件格式的库。
https://github.com/samtools/htslib
该库需要源码安装。
安装中发现需要以下依赖包:
zlib
需要先安装zlib 源码安装 wget http://www.zlib.net/zlib-1.2.11.tar.gz
bzlib
yum install bzip2-devel.x86_64
lzma
yum install -y xz-devel
curl/curl
yum install libcurl-dev libcurl-devel
安装上面依赖包以后cmake htslib以及cmake install通过。
2 对GenomeWorks进行安装,按照项目说明操作后,cmake过程中报错如下:
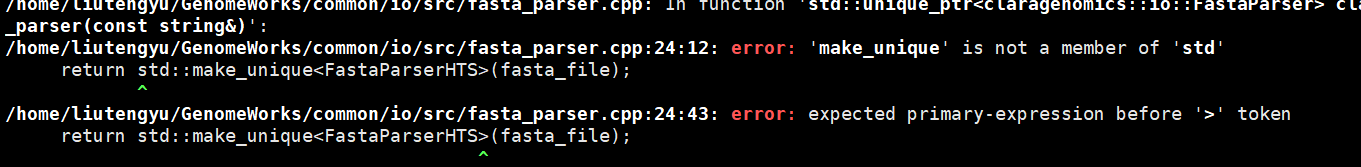
在std命名空间中没有这个成员,这个是出在cmake过程中,使用C++11标准,而应该采用C++14标准。如何使用C++14呢,我在报错的.cpp的CMakeLists显示的指定了C++14。

再次进行cmake报错变了
再次查找问题后,确定了我使用的编译器没有用gcc7.2而是用了gcc4.8.5

再次试验了下,果然系统中存在两个gcc,我应该使用的是带local的这个gcc,但是程序查找是按顺序的,所以先找到了低版本的gcc。
如何显示的指定cmake的编译器呢
-DCMAKE_C_COMPILER=/path/to/your/gcc -DCMAKE_CXX_COMPILER=/path/to/your/g++ 对CMAKE加这两个参数,显示指定gcc/g++路径。
解决完以上问题,按照官网的说明安装成功。






















 400
400











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








