程序如下:
load yeastdata.mat
%6400个数据,数据集相当大,许多信息对应于在实验中没有显示任何有趣变化的基因。为了更容易地找到有趣的基因,首先要做的是通过去除那些没有任何兴趣的表达资料来减少数据的大小。有6400个表达资料。你可以使用一些技术来将其减少到包含最重要基因的子集。
emptySpots = strcmp(‘EMPTY’,genes);% strcmp函数,并从数据集中删除索引命令
yeastvalues(emptySpots,:) = [];
genes(emptySpots) = [];
numel(genes)
nanIndices = any(isnan(yeastvalues),2);% 功能isnan用于识别缺失数据和索引命令的基因,用于删除缺少数据的基因。
yeastvalues(nanIndices,:) = [];
genes(nanIndices) = [];
numel(genes)
mask = genevarfilter(yeastvalues);% genevarfilter函数可以过滤出随时间变化的小变异基因。该函数返回与变量基因相同大小的逻辑数组,它们与方差的匹配值相对应,而方差大于第10百分位数和0,与阈值以下的0相对应。
% Use the mask as an index into the values to remove the filtered genes.
yeastvalues = yeastvalues(mask,:);% genelowvalfilter移除具有非常低的绝对表达值的基因。注意,基因过滤器函数也可以自动计算过滤的数据和名称。
genes = genes(mask);% 遗传熵驱器去除具有低熵的基因
numel(genes)
[mask, yeastvalues, genes] = …
genelowvalfilter(yeastvalues,genes,‘absval’,log2(3));
numel(genes)
[mask, yeastvalues, genes] = …
geneentropyfilter(yeastvalues,genes,‘prctile’,15);
numel(genes)
%如果产生新的数据,则可以使用两个设置变量将mapstd和processpca应用到其他数据中。
[x,std_settings] = mapstd(yeastvalues’); % Normalize data
[x,pca_settings] = processpca(x,0.15); % PCA
figure
scatter(x(1,:),x(2,:));
xlabel(‘First Principal Component’);
ylabel(‘Second Principal Component’);
title(‘Principal Component Scatter Plot’);
 net = selforgmap([5 3]);%生成一个5*3的神经元矩阵
net = selforgmap([5 3]);%生成一个5*3的神经元矩阵
view(net)
net = train(net,x);
nntraintool
figure
plotsompos(net,x);

y = net(x);
cluster_indices = vec2ind(y);
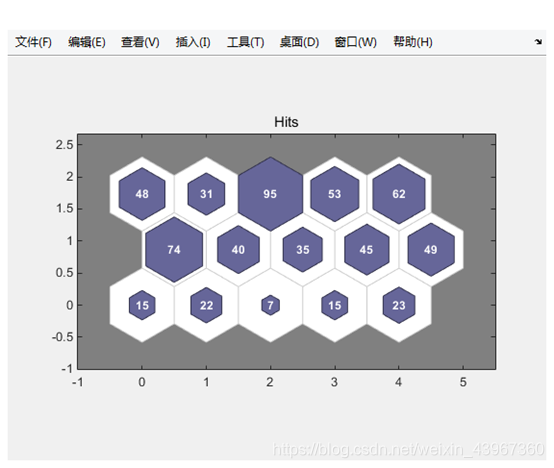
figure
plotsomhits(net,x);



























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








