相关资料
广州大学人工智能原理实验一:知识的表示与推理实验
广州大学人工智能原理实验二:八数码问题
广州大学人工智能原理实验三:产生式系统推理
广州大学人工智能原理实验四:TSP问题的遗传算法实现
广州大学人工智能原理实验五:基于汉诺塔的问题规约图实现
五份实验报告下载链接🔗
一、实验目的
本实验课程是计算机、智能、物联网等专业学生的一门专业课程,通过实验,帮助学生更好地掌握人工智能相关概念、技术、原理、应用等;通过实验提高学生编写实验报告、总结实验结果的能力;使学生对智能程序、智能算法等有比较深入的认识。要掌握的知识点如下:
1.掌握人工智能中涉及的相关概念、算法;
2.熟悉人工智能中的知识表示方法;
3.掌握问题表示、求解及编程实现;
4.熟悉和掌握遗传算法的基本概念和基本思想;
5.理解和掌握遗传算法的各个操作算子,能够用选定的编程语言设计简单的遗传优化系统;
6.通过实验培养学生利用遗传算法进行问题求解的基本技能。
二、基本要求
1.实验前,复习《人工智能》课程中的有关内容。
2.准备好实验数据。
3.程序可以在多个同学交流下讨论完成,但是实验报告独立完成。
4.该按照自己完成的部分进行。
三、实验软件
推荐使用C或C++(Visual studio等平台)(不限制语言使用,Java,matlab,Python等)。
四、实验内容:
以N个节点的TSP(旅行商问题)问题为例,应用遗传算法进行求解,求出问题的最优解。
1 旅行商问题
旅行商问题(Traveling Salesman Problem, TSP),又译为旅行推销员问题、货担郎问题,简称为TSP问题,是最基本的路线问题。假设有n个可直达的城市,一销售商从其中的某一城市出发,不重复地走完其余n-1个城市并回到原出发点,在所有可能的路径中求出路径长度最短的一条。
TSP问题是组合数学中一个古老而又困难的问题,也是一个典型的组合优化问题,现已归入NP完备问题类。NP问题用穷举法不能在有效时间内求解,所以只能使用启发式搜索。遗传算法是求解此类问题比较实用、有效的方法之一。
下面给出30个城市的位置信息:
| 城市编号 | 坐标 | 城市编号 | 坐标 | 城市编号 | 坐标 |
|---|---|---|---|---|---|
| 1 | (87,7) | 11 | (58,69) | 21 | (4,50) |
| 2 | (91,38) | 12 | (54,62) | 22 | (13,40) |
| 3 | (83,46) | 13 | (51,67) | 23 | (18,40) |
| 4 | (71,44) | 14 | (37,84) | 24 | (24,42) |
| 5 | (64,60) | 15 | (41,94) | 25 | (25,38) |
| 6 | (68,58) | 16 | (2,99) | 26 | (41,26) |
| 7 | (83,69) | 17 | (7,64) | 27 | (45,21) |
| 8 | (87,76) | 18 | (22,60) | 28 | (44,35) |
| 9 | (74,78) | 19 | (25,62) | 29 | (58,35) |
| 10 | (71,71) | 20 | (18,54) | 30 | (62,32) |
最优路径为:1 2 3 4 6 5 7 8 9 10 11 12 13 14 15 16 17 19 18 20 21 22 23 24 25 28 26 27 29 30
其路径长度为:424.869292
也可取前10个城市的坐标进行测试:
表2 Oliver TSP问题的10个城市位置坐标
| 城市编号 | 坐标 |
|---|---|
| 1 | (87,7) |
| 2 | (91,38) |
| 3 | (83,46) |
| 4 | (71,44) |
| 5 | (64,60) |
| 6 | (68,58) |
| 7 | (83,69) |
| 8 | (87,76) |
| 9 | (74,78) |
| 10 | (71,71) |
有人求得的最优路径为: 1 4 6 5 10 9 8 7 3 2 1
路径长度是166.541336
上述10个城市的求解中编号从0开始,把所有路径搜索完又返回到出发节点。
2 问题描述
应用遗传算法求解30/10个节点的TSP(旅行商问题)问题,求问题的最优解。
五、实验报告内容
(1)要求求出问题最优解,若得不出最优解,请分析原因;
一、取前10个城市测试:
参数设置:
| 交叉率 | 突变率 | 种群大小 | 进化代数 |
|---|---|---|---|
| 0.5 | 0.1 | 30 | 300 |
最优路径为:
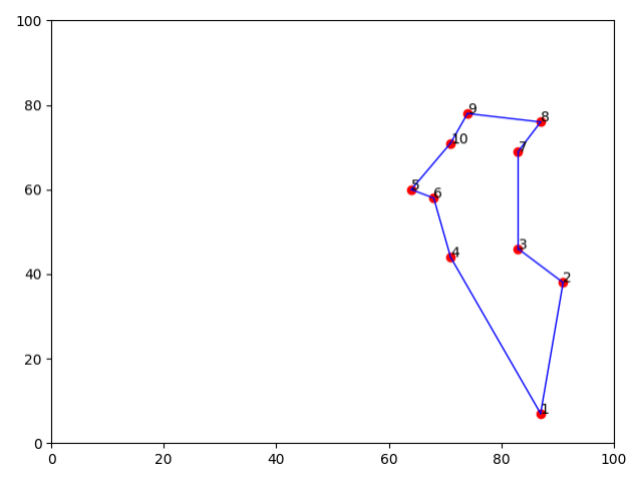
[ 1 2 3 7 8 9 10 5 6 4 1]
最优距离:166.54133557746266
二、取所有城市测试:
参数设置:
| 交叉率 | 突变率 | 种群大小 | 进化代数 |
|---|---|---|---|
| 0.6 | 0.3 | 200 | 1000 |
最优路径为: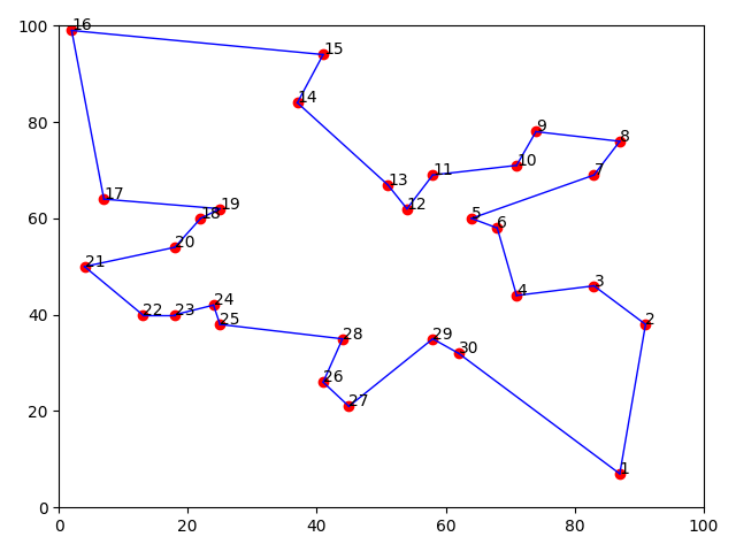
[13 12 11 10 9 8 7 5 6 4 3 2 1 30 29 27 26 28 25 24 23 22 21 20 18 19 17 16 15 14 13]
最优距离:424.8692923184516
(2)对实验中的几个算法控制参数进行仔细定义,并能通过实验选择参数的最佳值;
① 以所有城市测试为例,进行以下参数讨论;
② 每一条基因的定义为一条旅游路线,即每一条基因对应了30个城市数字下标,这30个数字每个都是有且只有一个;
③ 选择种群:优胜劣汰,对低于平均适应度的染色体进行交叉和突变,高于平均适应度的进行保留;
④ 交叉操作:选取两条父母染色体,对母亲染色体随机截取一段S,对父染色体在S上出现的数字剔除,然后拼接S,返回交叉后的染色体;
⑤ 突变操作:对染色体进行随机截取一段,进行翻转操作;
⑥ 种群进化:种群内每一根染色体都有可能进行交叉和突变,取决于交叉率和突变率。
⑦ 大的种群可以保证每一代里拥有的染色体更多,交叉和突变得到的新染色体更多,有助于跳出局部最优解,尽早获得全局最优解。
⑧ 为了快速得到实验结果,默认进化代数为500、种群大小为100,对交叉率和突变率进行消融实验
| pc\pm | 0.1 | 0.2 | 0.3 | 0.4 | 0.5 | 0.6 | 0.7 | 0.8 | 0.9 | 1.0 |
|---|---|---|---|---|---|---|---|---|---|---|
| 0.1 | 498.7 | 467.7 | 465.8 | 458.5 | 468.7 | 476 | 428.9 | 511.1 | 444 | 548.1 |
| 0.2 | 459.9 | 470.3 | 470.5 | 446.2 | 457.9 | 450.9 | 440.5 | 500.5 | 466.2 | 477.7 |
| 0.3 | 465.9 | 460.4 | 513.5 | 463.4 | 428.7 | 488.6 | 431.8 | 477.4 | 532.3 | 534.3 |
| 0.4 | 506 | 453.9 | 424.9 | 498.4 | 461.1 | 487.3 | 458 | 510.5 | 548.6 | 593.9 |
| 0.5 | 484.3 | 447.1 | 448.7 | 489.9 | 471.5 | 437 | 533.9 | 552.7 | 538 | 597.1 |
| 0.6 | 445.6 | 488 | 429.1 | 482.2 | 445.6 | 500.1 | 445 | 620.5 | 588.6 | 609.6 |
| 0.7 | 439.9 | 451.6 | 429.1 | 495.4 | 521.5 | 570 | 574.9 | 627.9 | 637.8 | 618.9 |
| 0.8 | 440.7 | 454.7 | 499.7 | 559.4 | 594.1 | 578.2 | 571.7 | 684.5 | 673.1 | 668.6 |
| 0.9 | 504.8 | 536.6 | 590.2 | 559.8 | 659.7 | 669.5 | 641.6 | 720.2 | 682.7 | 677.9 |
| 1.0 | 561.5 | 625 | 612.4 | 659.1 | 638.5 | 688.1 | 741.9 | 767.3 | 792.5 | 736.3 |
⑨ 可以看到pc取0.4,pm取0.3时,可取得全局最优解。但实验初始化具有随机性,参数的选择仅具有一定参考意义。
⑩ 解决办法:采用随机初始化种子,固定随机数的产生,进行多组消融实验,因花费时间可能过多,这里不再一一展开。
(3)要求界面显示每次迭代求出的局部最优解和最终求出的全局最优解。
一、取前10个城市测试:
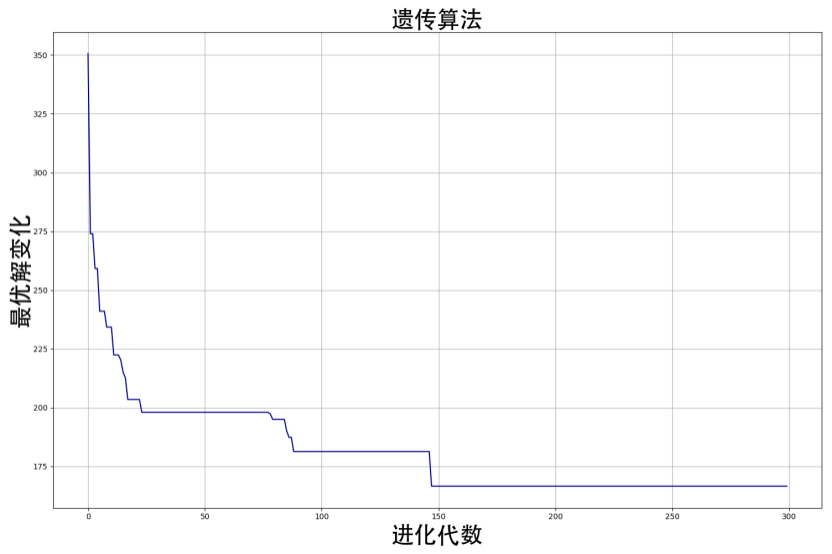
二、取所有城市测试:
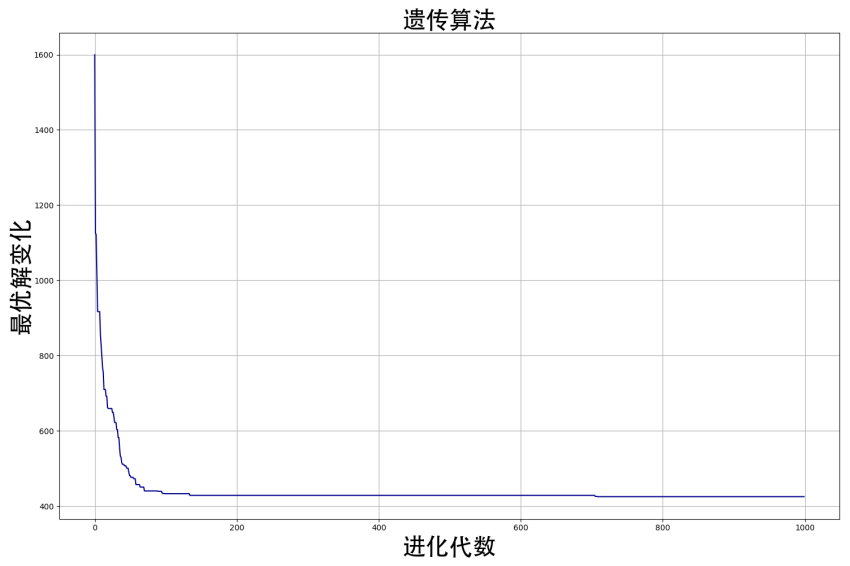
(4)实验结果讨论。
① 本次实验,前十城市还是所有城市,都可以成功寻得全局最优值,完成了实验的基本要求。
② 遗传算法的核心主要在于,种群淘汰、交叉与变异三个操作,因为这几个点的选择非常重要,所以要针对具体问题设计对应的操作,这样可以大大加快优化的时间。
③ 选择合适的操作之后,还要对参数进行设置,种群大小、交叉率、变异率,这三个参数可以直接影响是否能够跳出局部最优,寻得全局最优。
④ 在对于所有城市的求解过程中,可以发现全局最优解的获得往往要通过多次运行代码才能获得,因此对于该遗传算法应该仍有较大的优化空间。
⑤ 通过本次实验,可以发现一个种群存在着一些问题,就是每次保留的最优染色体都一直是由该种群产生的,很容易陷入局部最优解,或许可以采用多个种群解决这一问题。
⑥ 通过网上查阅,发现已经有人提出了多种群遗传算法MSGA,下面为多种群遗传算法流程简介图。
图一:多种群遗传算法流程图
⑦ 具体操作为:将初始种群展开成N个种群,然后同时进行进化(SGA,标准的遗传算法),然后在各个种群选出最优的,合成一个精华种群,然后反复迭代更新,可以设置一个早停参数M,避免运行时间过长。
六、实验代码
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from matplotlib.lines import Line2D
from matplotlib.font_manager import FontProperties
class YIchuan(object):
best_distance = -1 # 记录目前最优距离
best_gene = [] # 记录目前最优旅行方案
all_best_distance = [] #记录每一代最优距离
citys = np.array([]) # 城市数组
citys_name = np.array([])
population_size = 100 # 种群大小,每个种群含有多少条基因
cross_rate = 0.9 # 交叉率
change_rate = 0.1 # 突变率
population = np.array([]) # 种群数组
fitness = np.array([]) # 适应度数组
city_size = -1 # 标记城市数目
iter_num = 200 # 最大迭代次数
def __init__(self, cross_rate, change_rate, population_size, iter_num):
self.fitness = np.zeros(self.population_size)
self.cross_rate = cross_rate
self.change_rate = change_rate
self.population_size = population_size
self.iter_num = iter_num
self.fig, self.ax = plt.subplots()
self.plt = plt
def init(self):
TSP = self
TSP.load_city_data() # 加载城市数据
TSP.population = TSP.creat_population(TSP.population_size) # 创建种群
TSP.fitness = TSP.get_fitness(TSP.population) # 计算初始种群适应度
self.ax.axis([0, 100, 0, 100])
def creat_population(self, size):
"""
创建种群
:param size:种群大小
:return: 种群
"""
population = [] # 存储种群生成的基因
for i in range(size):
gene = np.arange(self.citys.shape[0])
np.random.shuffle(gene) # 打乱数组[0,...,city_size]
population.append(gene) # 加入种群
return np.array(population)
def get_fitness(self, population):
"""
获得适应度
:param population:种群
:return: 种群每条基因对应的适应度
"""
fitness = np.array([]) # 适应度记录数组
for i in range(population.shape[0]):
gene = population[i] # 取其中一条基因(编码解,个体)
dis = self.compute_distance(gene) # 计算此基因优劣(距离长短)
dis = self.best_distance / dis # 当前最优距离除以当前population[i](个体)距离;越近适应度越高,最优适应度为1
fitness = np.append(fitness, dis) # 保存适应度population[i]
return fitness
def select_population(self, population):
"""
选择种群,优胜劣汰
策略:低于平均的要替换
:param population: 种群
:return: 更改后的种群
"""
best_index = np.argmax(self.fitness)
ave = np.median(self.fitness, axis=0)
for i in range(self.population_size):
if i != best_index and self.fitness[i] < ave:
pi = self.cross(population[best_index], population[i]) #交叉
pi = self.change(pi) #变异
population[i, :] = pi[:]
return population
def cross(self, parent1, parent2):
"""
交叉
:param parent1: 父亲
:param parent2: 母亲
:return: 儿子基因
"""
if np.random.rand() > self.cross_rate:
return parent1
index1 = np.random.randint(0, self.city_size - 1)
index2 = np.random.randint(index1, self.city_size - 1)
tempgene = parent2[index1:index2] # 交叉的基因片段
newgene = []
xxx = 0
for g in parent1:
if xxx == index1:
newgene.extend(tempgene) # 插入基因片段
if g not in tempgene:
newgene.append(g)
xxx += 1
newGene = np.array(newgene)
return newGene
def reverse_gene(self, gene, i, j):
"""
翻转i到j位置的基因
:param gene: 基因
:param i: 第i个位置
:param j: 第j个位置
:return: 翻转后的基因
"""
if i >= j:
return gene
if j > self.city_size - 1:
return gene
parent1 = np.copy(gene)
tempgene = parent1[i:j]
newgene = []
p1len = 0
for g in parent1:
if p1len == i:
newgene.extend(tempgene[::-1]) # 插入基因片段
if g not in tempgene:
newgene.append(g)
p1len += 1
return np.array(newgene)
def change(self, gene):
"""
突变,主要使用翻转
:param gene: 基因
:return: 突变后的基因
"""
if np.random.rand() > self.change_rate:
return gene
index1 = np.random.randint(0, self.city_size - 1)
index2 = np.random.randint(index1, self.city_size - 1)
new_gene = self.reverse_gene(gene, index1, index2) #翻转
return new_gene
def evolution(self):
"""
迭代进化种群
:return: None
"""
for i in range(self.iter_num):
best_index = np.argmax(self.fitness)
worst_f_index = np.argmin(self.fitness)
local_best_genee = self.population[best_index]
local_best_distance = self.compute_distance(local_best_genee)
if i == 0: #第一代记录最优基因和最短距离
self.best_gene = local_best_genee
self.best_distance = self.compute_distance(local_best_genee)
if local_best_distance < self.best_distance:
self.best_distance = local_best_distance # 记录最优值
self.best_gene = local_best_genee # 记录最个体基因
else:
self.population[worst_f_index] = self.best_gene #替换掉最差的基因
print('代数:%d 最优距离:%s' % (i, self.best_distance))
self.all_best_distance.append(self.best_distance)
self.population = self.select_population(self.population) # 选择淘汰种群
self.fitness = self.get_fitness(self.population) # 计算种群适应度
for j in range(self.population_size):
k = np.random.randint(0, self.population_size - 1)
if j != k:
self.population[j] = self.cross(self.population[j], self.population[k]) # 交叉种群中第j,k个体的基因
self.population[j] = self.change(self.population[j]) # 突变种群中第j个体的基因
def load_city_data(self, file='city.csv', delm=';',head = 30):
# 加载实验数据
data = pd.read_csv(file, delimiter=delm, header=None).values
self.citys = data[:head, 1:]
self.citys_name = data[:head, 0]
self.city_size = data[:head].shape[0]
def compute_distance(self, gen):
# 计算该基因的总距离
distance = 0.0
for i in range(-1, len(self.citys) - 1):
index1, index2 = gen[i], gen[i + 1]
city1, city2 = self.citys[index1], self.citys[index2]
distance += np.sqrt((city1[0] - city2[0]) ** 2 + (city1[1] - city2[1]) ** 2)
return distance
def compute_oushi_distance(self, city1, city2):
# 计算两个地点之间的欧氏距离
dis = np.sqrt((city1[0] - city2[0]) ** 2 + (city1[1] - city2[1]) ** 2)
return dis
def draw_line(self, from_city, to_city):
"""
连线
:param from_city: 城市来源
:param to_city: 目的城市
:return: none
"""
line1 = [(from_city[0], from_city[1]), (to_city[0], to_city[1])]
(line1_xs, line1_ys) = zip(*line1)
self.ax.add_line(Line2D(line1_xs, line1_ys, linewidth=1, color='blue'))
def draw_citys_way(self, gene):
'''
根据一条基因绘制一条旅行路线
:param gene:
:return:none
'''
num = gene.shape[0]
self.ax.axis([0, 100, 0, 100])
for i in range(num):
if i < num - 1:
best_i = self.best_gene[i]
next_best_i = self.best_gene[i + 1]
best_icity = self.citys[best_i]
next_best_icity = self.citys[next_best_i]
self.draw_line(best_icity, next_best_icity)
start = self.citys[self.best_gene[0]]
end = self.citys[self.best_gene[-1]]
self.draw_line(end, start)
def draw_citys_name(self, gen, size=5):
'''
根据一条基因gen绘制对应城市名称
:param gen:
:param size: text size
:return:
'''
m = gen.shape[0]
self.ax.axis([0, 100, 0, 100])
for i in range(m):
c = gen[i]
best_icity = TSP.citys[c]
self.ax.text(best_icity[0], best_icity[1], TSP.citys_name[c], fontsize=10)
def draw(self):
"""
绘制最终结果
:return: none
"""
self.ax.plot(self.citys[:, 0], self.citys[:, 1], 'ro')
self.draw_citys_name(self.population[0], 8)
self.draw_citys_way(self.best_gene)
plt.show()
def draw_dis(selfs):
"""
绘制最优距离曲线
:return: none
"""
x = [i for i in range(len(selfs.all_best_distance))]
y = selfs.all_best_distance
fig, ax = plt.subplots(figsize=(15, 10))
font = FontProperties(fname=r"/System/Library/Fonts/STHeiti Medium.ttc", size=20)
ax.plot(x, y, color='darkblue', linestyle='-')
plt.xlabel("进化代数", FontProperties=font, size=30) # X轴标签
plt.ylabel("最优解变化", FontProperties=font, size=30) # Y轴标签
plt.title("遗传算法", FontProperties=font, size=30)
plt.grid()
plt.show()
if __name__ == '__main__':
"""消融实验
number = np.ones([10,10])
for i in range(10):
for j in range(10):
TSP = YIchuan(0.1*(i+1), 0.1*(j+1), 100, 500) # 交叉率、突变率、种群大小、进化代数
TSP.init()
TSP.evolution()
TSP.draw()
TSP.draw_dis()
print("最优路径为:")
print(np.append(TSP.best_gene, TSP.best_gene[0]) + 1)
number[i,j] = TSP.best_distance
number = pd.DataFrame(number)
number.to_csv('out.csv')
"""
TSP = YIchuan(0.4, 0.3, 200, 1000) # 交叉率、突变率、种群大小、进化代数
TSP.init()
TSP.evolution()
TSP.draw()
TSP.draw_dis()
print("最优路径为:")
print(np.append(TSP.best_gene,TSP.best_gene[0])+1)























 6327
6327











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










