SPM计算脑指标
通过自己编写matlab脚本调用spm函数计算脑指标
数据地址👆
(11 封私信 / 80 条消息) zll - 知乎 (zhihu.com)
先进行数据预处理
part1 spm中fmri数据预处理 - 知乎 (zhihu.com)
一文看懂如何用 Python 查看三维数据 (nii.gz格式) 的各种图像参数_.nii.gz-CSDN博客
fmri--只有影像学数据,怎么拿到采集参数TR、slice num - 哔哩哔哩 (bilibili.com)
SPM系列教程-02:预处理之时间校正slice-timing correction哔哩哔哩bilibili
dicom转换到nifti
这一步可以用spm中的dicom import,由于我这里数据就是nii的,所以略过

batch添加对应module
slicetiming

选择上面的temporal里的slice timing

选择session并在里面选中对应文件夹

这里可以先在matlab里面运行fmridata=spm_vol('xxxxx.nii')把对应的数据拿进去看struct里有多少个,就说明一个nii最多会有多少,也可以输一个很大的数字,比如10000(1:1000),这样就会自动截取到最大的数
这里截图没截全,逗号后面是有个1的,需要在frames指定区间才能把一个nii里的所有都显示出来

按下回车后可以看到最大是590

在filter里面输入正则表达式,这里我们先只选中所有run-1的文件

右键会有select ALL,点击把所有文件选择进来

可以看到session1此时已经包含了第一个run的所有文件,下面两个session一样选择之后两个run的所有文件
下面slice number和TR TA的确认一般如果是dicom原始文件的话可以从dicominfo('xxx')来查看dicom文件包含的信息
但是现在文件初始已经是nifti格式,不过是在openneuro网站下载的,在下载的时候可以看到文件都有附带对应的json文件,json文件里一般会把信息写清楚。
{
"ConversionSoftware": "dicm2nii.m 20181126",
"SeriesNumber": 3,
"SeriesDescription": "fMRI_Run_1",
"ImageType": ["ORIGINAL", "PRIMARY", "M", "MB", "ND", "MOSAIC"],
"Modality": "MR",
"AcquisitionDateTime": "20180921141748.780000",
"TotalReadoutTime": 0.041399172,
"SliceTiming": [
0,
0.685,
0.3925,
0.0975,
0.7825,
0.49,
0.195,
0.88,
0.5875,
0.2925,
0,
0.685,
0.3925,
0.0975,
0.7825,
0.49,
0.195,
0.88,
0.5875,
0.2925,
0,
0.685,
0.3925,
0.0975,
0.7825,
0.49,
0.195,
0.88,
0.5875,
0.2925,
0,
0.685,
0.3925,
0.0975,
0.7825,
0.49,
0.195,
0.88,
0.5875,
0.2925 ],
"RepetitionTime": 1,
"PhaseEncodingDirection": "j-",
"EffectiveEchoSpacing": 0.000599988,
"EchoTime1": 0.035,
"deltaTE": 34,
"EchoTime2": 0.001,
"SliceThickness": 2.73,
"FlipAngle": 62,
"Manufacturer": "SIEMENS",
"SoftwareVersion": "syngo MR E11",
"MRAcquisitionType": "2D",
"InstitutionName": "R",
"InstitutionAddress": "Medical Center Dr 5,Columbia,SC,US,29203",
"DeviceSerialNumber": "67060",
"ScanningSequence": "EP",
"SequenceVariant": "SK\\SS",
"ScanOptions": "FS",
"SequenceName": "epfid2d1_70",
"TaskName":"affect"
}
可以看到slicetiming里有几行,就是扫描了几个slice
repetitionTime就是TR,这个文件的TR是1
当然如果只有nii文件的时候也没关系,可以在matlab里把nii的数据用spm_vol读进来然后直接看

这里可以看到前面的frames是590.是一个四维的数据,第四维就是slice number就是40,跟前面json文件里数的也一样
TR可以通过fmri_data(1).private.timing.tspace

得到,跟刚刚json文件里的repetition time也是一样的
而TA就是TR-TR/slice number(1-1/40),直接填进去就可以,也可以先不填直接写0.只要后面slice order写好了,spm会自己计算的
在json文件中可以看到这个数据是一个多通道收集的数据,因为他同时出现了四个0,说明在最开始他有四个通道同步进行
这里可以直接把json的slice timing复制到spm里面
0,
0.685,
0.3925,
0.0975,
0.7825,
0.49,
0.195,
0.88,
0.5875,
0.2925,
0,
0.685,
0.3925,
0.0975,
0.7825,
0.49,
0.195,
0.88,
0.5875,
0.2925,
0,
0.685,
0.3925,
0.0975,
0.7825,
0.49,
0.195,
0.88,
0.5875,
0.2925,
0,
0.685,
0.3925,
0.0975,
0.7825,
0.49,
0.195,
0.88,
0.5875,
0.2925

reference slice就是之后在进行realign头动矫正的时候需要以哪个slice为基准,填1就是以第一个slice为基准
filename prefix就是生成之后的文件以什么为前缀
在创建完这个batch之后可以把这个batch的matlab文件保存下来,之后再想运行slice timing可以用图形界面重新点到batch里面把这个文件加载进来,修改对应的参数
如果不用改参数也可以直接把这个文件load进来直接run,这样就不用点到图形界面进去了
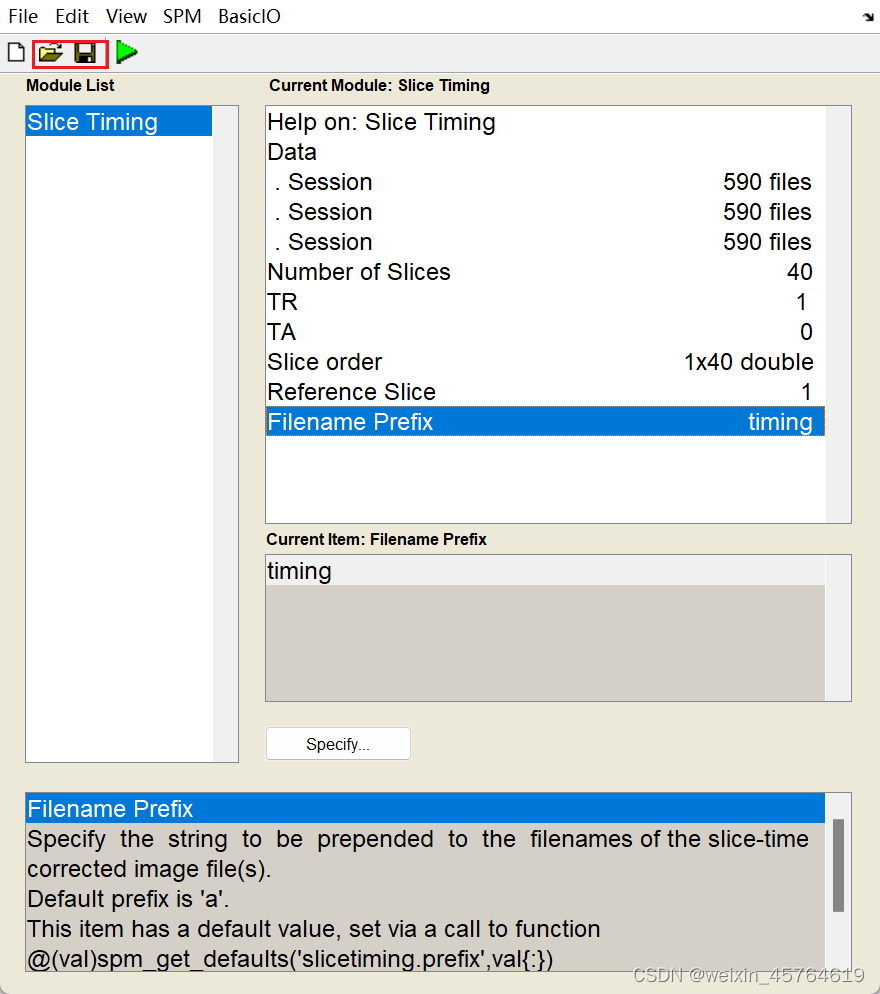
左边是load,右边是保存
比如设置成slice timing.mat保存下来
然后用xx=load('slice timing.mat')加载进来,可以看里面包含的数据结构
可以再次在batch里面load这个文件,也可以直接
spm_jobman('run',xx(1).matlabbatch)
























 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








