论文标题: A multi-sequences MRI deep framework study pplied to glioma classfication
翻译: 多序列MRI深度框架研究在胶质瘤分类中的应用
摘要
胶质瘤是最重要的中枢神经系统肿瘤之一,在男性和女性最常见的癌症中排名第15位。磁共振成像(MRI)是医学专家诊断神经胶质瘤的常用工具。根据病理的严重程度,从MRI中选择一组多序列。我们提出的方法旨在创建一个计算机辅助系统,能够帮助更多的专家诊断脑胶质瘤。我们提出了一种基于卷积神经网络框架和迁移学习技术的监督学习机制。我们的研究更侧重于不同预训练深度学习模型在不同MRI序列下的表现。我们强调了这种模型- mri序列对的最佳组合,以用于我们对健康脑与胶质瘤脑进行分类的特定任务。此外,我们还提出对提取的深层特征进行视觉分析,以研究MRI序列与模型的存在关系。这种可解释性分析为医学专家理解模型所作的诊断提供了一些提示。我们的研究是基于著名的BraTS数据集,包括多序列图像和专家诊断。
介绍
世界卫生组织已经定义了各种诊断标准,允许对神经胶质瘤进行分类。根据胶质瘤的形态学评价、增殖指数、治疗反应和患者的生存时间,将胶质瘤分为四个级别。因此,I级为良性肿瘤,II级为相对非恶性肿瘤,III级为低级别恶性肿瘤。IV级用于恶性程度最高的肿瘤,患者的生存希望在6 - 12个月之间。
本文的一个科学贡献是提出了一个完整的框架,该框架使用五种有效且众所周知的CNN模型来研究不同成像序列在特定任务中的性能。我们评估了最常见的MRI采集序列的相关性。我们的研究还强调了为特定任务选择和组合最佳序列以减少处理和分析时间的重要性。我们还研究了CNN模型提取的深层特征。深度特征有助于解释和理解深度学习方法及其性能。重要的是,虽然我们提出的框架最初是为神经胶质瘤检测设计的,但它可以应用于其他计算机辅助医疗任务。在ADNI数据集上的一些实验表明,我们的框架对另一项任务(即检测患有阿尔茨海默病的患者)具有适应性。
我们在三个基准数据集上的详尽实验证明了正确选择序列和深度学习网络对于神经胶质瘤病理和阿尔茨海默病任务的重要性。结果强调了在选择序列的数量方面效率和准确性之间可能存在的权衡。本文的一个科学贡献是提出了一个完整的框架,该框架使用五种有效且众所周知的CNN模型来研究不同成像序列在特定任务中的性能。我们评估了最常见的MRI采集序列的相关性。我们的研究还强调了为特定任务选择和组合最佳序列以减少处理和分析时间的重要性。我们还研究了CNN模型提取的深层特征。深度特征有助于解释和理解深度学习方法及其性能。重要的是,虽然我们提出的框架最初是为神经胶质瘤检测设计的,但它可以应用于其他计算机辅助医疗任务。在ADNI数据集上的一些实验表明,我们的框架对另一项任务(即检测患有阿尔茨海默病的患者)具有适应性。
我们在三个基准数据集上的详尽实验证明了正确选择序列和深度学习网络对于神经胶质瘤病理和阿尔茨海默病任务的重要性。结果强调了在选择序列的数量方面效率和准确性之间可能存在的权衡。
请注意,本文是我们初步研究的延伸,在Brats 2020和ADNI数据集上获得了新的实验结果,显示了我们的框架对不同任务的适应性。我们还纳入了一项多序列融合研究,包括突触权重和深度模型相关分析。我们还使用更多的可视化技术扩展了对深层特征的分析。
材料和方法
在本节中,描述了我们研究胶质瘤分类的管道。图1说明了分析流程,从大脑的MRI数据库开始检测胶质瘤。对患者头部进行三维高分辨率图像剥离,提取二维切片和三维立方体序列。接下来,我们将这些不同的图像提取到图像归一化的预处理步骤。我们随后应用了数据增强技术,然后根据它们的维度将它们输入到2D和3D预训练的神经网络中。最后,采用迁移学习方法对预训练的卷积神经网络进行增量拟合,完成神经胶质瘤分类任务。在此步骤之后,我们对模态和网络组合的性能进行了详尽的分析。同时对深层特征进行提取和分析,进行可解释性分析。对特征映射的研究可以更好地理解网络用于学习的特征。
数据
在医学和医学研究中,MRI已成为检测胶质瘤的重要诊断手段。通过这种光学成像方式,序列采集的几种方式被用来突出肿瘤的不同组织。典型的肿瘤组织有水肿、核心坏死和边缘活动。对于水肿的检测,可以使用T2 FLAIR(流体衰减反转恢复)序列。同样,对于肿瘤核心(通常是坏死组织)的检测,可以使用T1ce序列(T1序列加上注射钆作为造影剂)。
我们选择了BraTS挑战2018和2020数据库其中包含不同的序列用于我们的研究。BraTS数据库(多模式脑肿瘤分割)是由CBICA(生物医学图像计算和分析中心)与MICCAI(医学图像计算和计算机辅助干预协会)合作制作的数据库。Brats 2018数据库包含从300多名患者收集的MRI大脑数据横切面扫描的在横切面扫描的数据格式为NIFTI文件(.nii.gz),包含四个不同的序列,如下所列:
- T1序列:纵向松弛
- T1ce (T1-对比度增强)序列:纵向松弛加造影剂(钆)序列
- T2序列:横向松弛
- T2 FLAIR(流体衰减反转恢复)序列:横向松弛加反转恢复(流体衰减)序列
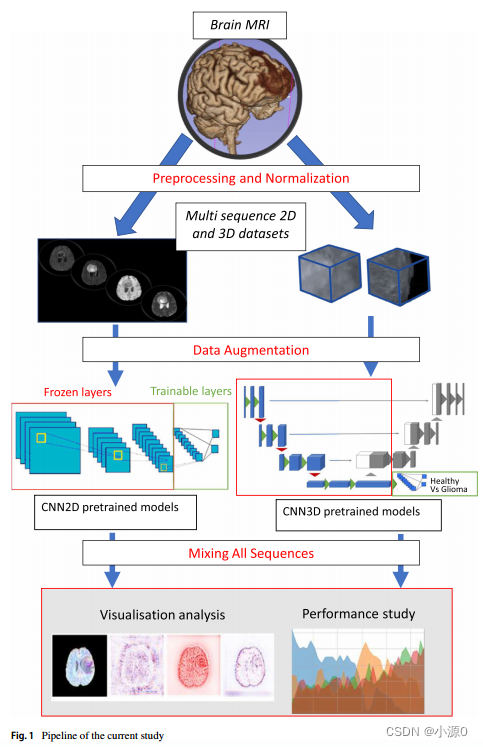
图2显示了高级别胶质瘤患者的四种类型的序列扫描。
该MRI数据库来自19个不同的机构,包含不同级别的胶质瘤。肿瘤的分割和标记是事先由经过批准和经验丰富的神经放射学家按照相同的协议手工完成的。根据BraTS 2018数据库的描述,这些数据代表了210名高级别胶质瘤患者和75名低级别胶质瘤患者。然而,根据当地神经放射学家和WHO的标准,如Dequidt等[18,19],254例患者为高级别患者,31例为低级别患者。因此,最终的LGG与HGG的比值为0.109,如作者报告的那样,这使得研究与这种类型的数据库的平衡非常复杂。因此,对于我们的研究和胶质瘤的研究,我们使用深度神经网络对胶质瘤的存在与否进行分类和预测。
因此,存在两种输出:只有健康的脑组织和存在肿瘤的脑组织。这些数据被分为训练集、验证集和测试集。然后,在最终被cnn使用之前,它们经历了标准化、准备和数据增强的预处理步骤。
为了进行研究,我们还选择了BraTS 2020数据集。这个数据库在几个方面与以前的数据库不同。只保留了12- 13届BraTS的图片和注释。其他数据被删除,因为它们包含了术前和术后的混合分析。神经放射学家重新评估了所有原始TCIA胶质瘤标本。因此,所有术前扫描仪都由专家注释并包含在BraTS数据集2020中。我们的研究总共对369例患者的数据进行了实验,其中259例为高级别胶质瘤,110例为低级别胶质瘤。
此外,与BraTS 2018相比,BraTS 2020增加了84个MRI序列,即增加了29.6%的新数据。
最后,为了概括研究,我们在另一个小数据集:ADNI上执行了工作流程。我们选择了两个亚组,每组20例患者,包括10例健康和10例严重患有阿尔茨海默病为每位患者选择三种不同的MRI序列(即MPRAGETSE, Double-TSE和T2-Proton Density)。
预处理
在选定的数据库上训练神经网络之前,将NIFTI文件(MRI)转换为图像数据,以应对CNN输入。一般来说,Keras库接受许多图像格式(例如,PNG, JPG, BMP, PPM或TIFF)用于训练神经网络。
在我们的研究中,我们使用PNG格式,因为它具有无损压缩1。此外,实验图像必须包含三个与RGB通道基底相对应的通道。
这个要求是由于所研究的网络只能接受RGB格式的图像,而需要一些最小像素大小的图像。
Ahmad等人已经将这种预处理MRI数据的想法用于深度迁移学习的肌肉分割。此外,在数据归一化之前,所有大脑的强度缩放(IS)仅用于大脑的健康区域。IS方法在孙晓飞等人中有描述。
对于每个病人,我们得到他们的横切面,神经网络将用它来训练。根据专家的分割,对每个横切面进行分类并标记是否有胶质瘤。只有背景值(黑色)的切片被排除;它们代表了没有数据存在的颅盒的外部界限。这些切片被转换为PNG格式,以匹配二维神经网络的输入格式。因此,将图像转换为PNG将体素的深度从16位减少到8位。因此,信息可能会丢失。本研究的目的之一是找出这种降维中包含的信息是否足以用于从标准图像中大量学习的cnn。因此,使用迁移学习技术对这些图像进行训练可以显著减少计算机资源和处理时间。这个程序面对的是一个三维网络的研究,保留在16位格式。由于数据集很大(50,812片),我们使用三向保留方法将数据分成三个不同的子集(即训练集、验证集和测试集)。
数据之前被平衡和划分,70%用于训练,15%用于验证,15%用于测试。请注意,这种分裂不是随机进行的,而是对患者而不是对切片进行的。这样,我们就可以确保训练集、验证集和测试集没有偏差。
数据增加
一般来说,为了从机器学习模型中获得良好的性能,我们需要与模型参数数量成比例的大量示例;以及模型必须执行的任务的复杂性。由于我们研究数据的稀缺性,有必要采用图像数据增强技术来扩大我们的数据集。此外,使用该技术不仅可以解开有限的数据,还可以帮助深度学习算法避免过拟合问题。尽管CNN可以是不变的(即图像可以放置在不同的方向,平移,视点,大小或照明),但医疗数据集仍然包含有限条件下的样本。相比之下,真实情况下的MRI可能存在多种条件,如不同的方向、姿态、尺度、亮度等。
在将训练数据输入神经网络之前,我们对原始MRI数据进行了几次转换。我们在这项工作中应用的所有必要的变换如下:
- 旋转40度;
- 纵向变形率可达0.2;
- 轴向变形率可达0.2
- 剪切效应的比例可达0.2
- 放大到0.2的比例
- 水平和/或垂直翻转
模型的实现
对于每个模型,根据它们在imageNet上的表现加载它们的突触权重。这些2D网络需要巨大的计算能力来进行训练,并且需要大量的时间来实现ILSVRC比赛的性能结果。这些不同的2D神经网络有1700万到3100万个可训练参数(高效网络b6有4300万个)。在一个小的MRI数据库中训练每个参数,并希望为这数百万个参数调整突触权重是不切实际的。
迁移学习的目标是在imageNet挑战的数百万张图像上为这些神经网络保留这些先前学习过的突触权重,因此,冻结层并使参数不可训练。这种方法背后的主要思想是,医学图像的特征,无论是来自核磁共振扫描还是其他方式,都与“标准”图像具有相同的特征。因此,形状,相邻像素之间的强度差异,颜色或灰色的阴影等,允许ILSVRC挑战的分类,可以在MRI图像中找到。
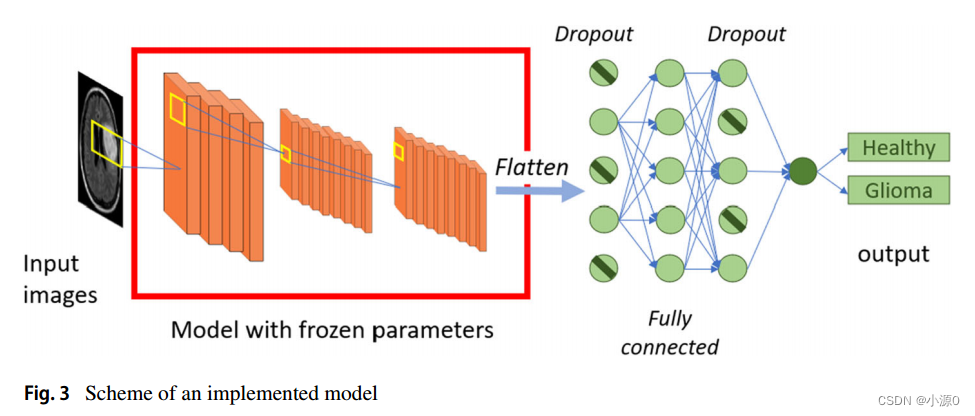
Tajbakhsh等人报道,迁移学习在注释很少的数据库的特定任务中显示出巨大的潜力。只有表示这些模型输出的瓶颈被更改为特定于任务的。因此,我们在每个研究模型中删除最后一层,并添加一个flatten层,将数据标准化为单个向量。这一层随后连接到一个小的神经元网络,形成完全连接的神经元。这种移除可能会引入0.5的dropout,这意味着只有50%的神经元在训练阶段通过了学习到的信息。
通过这样做,我们通过不允许神经元将信息传递到下一层来正则化深度神经网络。这种方法减少了过拟合的可能性,提高了泛化误差。最后,这些神经网络的输出连接到最后一层,这一层由研究过程中有多少类神经元组成。这里有两个神经元。每一个都代表一个分类输出,这意味着,大脑的一部分只代表健康组织,或者大脑的一部分有胶质瘤的存在。只有这最后几层是在MRI数据库的注释图像上训练的。我们可以训练的参数总数如表1所示,Adam 10-5是优化器。
dropout层允许一定比例的神经元停止训练,防止在每个epoch重新调整它们的突触权重。dropout层的主要目的是避免过拟合和增强网络的泛化能力。
没有这些退出层,InceptionV3、ResNet50和DenseNet模型在训练集上接近100%的准确率,但在验证集或测试集上完全随机,这是一个明显的过拟合问题。
全连接层将从卷积块中提取的特征重新组合以提出分类。这些层中的所有神经元都是相互连接的,并且有自己的重量。这种层的使用是微妙的,因为参数的数量相对于层中存在的神经元数量呈指数增长。因此,计算时间也相应增加。模型实现的总体方案如图3所示。





















 2万+
2万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








