今天给各位老铁们分享一篇于2024年10月17号发表在 Nature Machine Intelligence [18.8] 的文章:"Estimation of causal effects of genes on complex traits using a Bayesian-network-based framework applied to GWAS data",使用基于贝叶斯网络的框架估算基因对复杂性状的因果效应,并将其应用于 GWAS 数据。

DOI:10.1038/s42256-024-00906-7
| 摘要
复杂性状的遗传研究一直面临挑战,传统GWAS方法难以解释基因的直接和间接作用。本文提出了一个基于贝叶斯网络的基因-性状关联分析框架(BN-GWAS),用于量化基因对复杂性状的总和直接/间接因果效应。BN-GWAS结合GWAS数据和基因表达数据,能够灵活应用于不同数据集,并可拓展至多个性状的联合因果网络分析。通过模拟和大规模实证研究验证了该方法的准确性、特异性和精确度,尤其在基因选择方面表现出显著优势。作者应用BN-GWAS对52种性状和14种组织的分析揭示了这些性状的遗传结构,并发现了许多具有直接因果效应的核心基因。总而言之,BN-GWAS的优势包括能够量化基因的总效应和直接效应,避免传统基因分析中的混杂效应,并具有广泛的适用性。
| 所用数据
-
英国生物样本库(UKBB,UK Biobank):作者使用该数据集提供了个体表型和基因型数据,用于推断基因与复杂性状之间的因果网络。
-
GTEx RNA-seq数据:研究使用了GTEx V7版本的RNA测序数据,这些数据通过GTEx门户公开提供,GTEx数据用于推断组织特异性的基因表达和基因间因果关系。
-
Open Targets数据库:作者使用该数据库用于基因和复杂疾病关联的验证和功能注释。
| 研究内容
1. BN-GWAS框架
BN-GWAS 是一个用于识别基因与复杂性状之间的因果关系的框架,能够区分不同类型的因果基因(核心基因、外围基因和中介基因),并量化它们对性状的总因果效应以及直接/间接的因果效应。该框架结合了来自不同来源的 GWAS数据 和 基因表达 数据,以构建基因与性状之间的因果网络。简单来说,BN-GWAS框架的流程首先通过英国生物样本库(UKBB)的个体基因型和表型数据提取小于0.01的次等位基因频率(MAF)的SNPs,并使用PrediXcan工具获得组织特异性的顺式(cis)预测表达谱(步骤1)。随后,采用公开的转录表达定量特性位点(trans-eQTL)数据补充顺式预测的表达数据(步骤2),在调整群体结构后,使用PC-simple算法推断基因与性状的因果关系(步骤3)。接着,利用GTEx V7组织特异性RNA测序数据,经过混杂变量调整和基因筛选后,使用PC-algorithm推断基因-基因因果网络(步骤4)。最后,将推断出的基因-基因网络与基因-性状网络结合,使用基于惩罚的IDA和jointIDA方法估算不同基因对目标性状的总因果效应和直接因果效应,构建完整的基因-基因-性状因果网络(步骤5)。
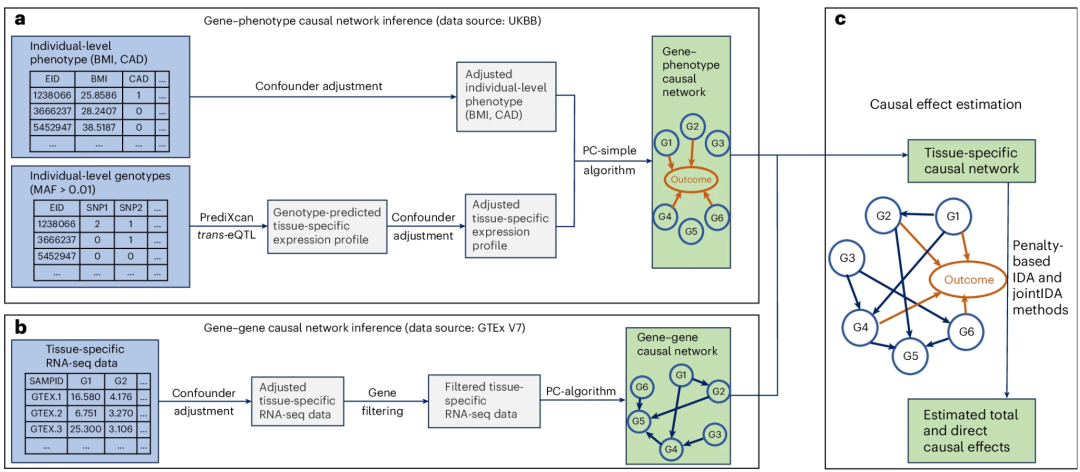
▲ BN-GWAS框架示意图。
2. BN-GWAS的应用展示
作者随后将BN-GWAS应用于英国生物样本库的52种性状,分析了这些性状在不同组织中的遗传机制。发现了大量具有因果作用的基因,并揭示了某些性状的复杂遗传结构。
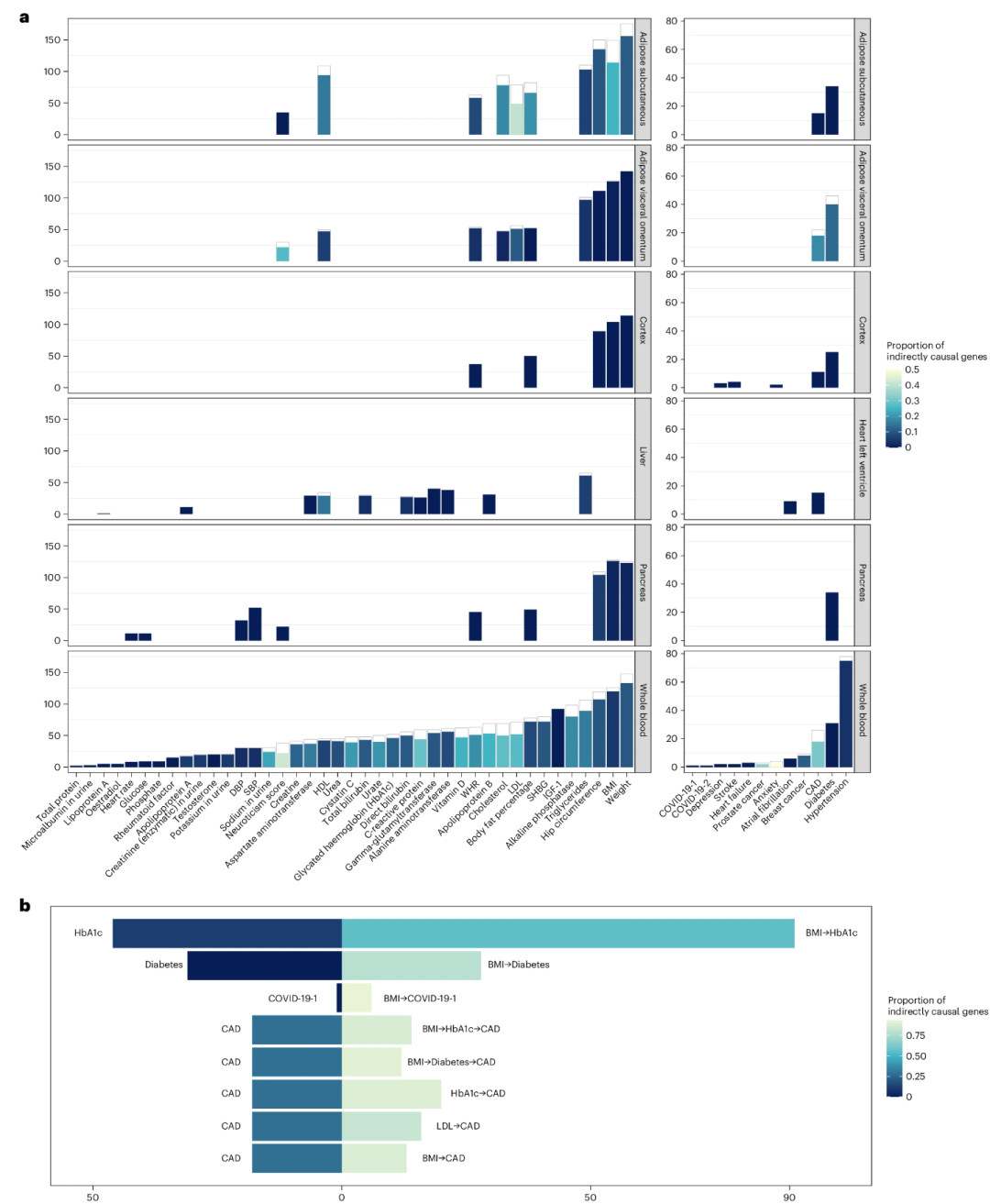
▲ BN-GWAS在不同组织中发现的与各种性状有因果关系的基因数量和比例:a. 单一性状分析中因果相关基因的检测结果。b. 多重性状分析中因果相关基因的检测结果。
基于基因间关联及其与疾病关联图的汇总方法
多疾病分析、直接间接因果基因挖掘
就分享到这了
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








