公共数据库挖掘
1. 国家基因组科学数据中心(NGDC):Home - National Genomics Data Center
2. EBI下载:ENA Browser
ENA是欧洲的生物测序数据库网站,在https://www.ebi.ac.uk/ena/browser/home 网址,输入文章提供数据的id。

3. GSA
GSA (Genome Sequence Archive)是2015年底,中科院北京基因组研究所生命与健康大数据中心开发的原始组学数据归档库。数据模型和数据格式遵照INSDC标准,在功能上等同于NCBI的SRA,EBI的ENA和DDBJ的DRA。

4. CNGBdb

5. NCBI 下载
到 NCBI SRA 数据库中搜索SRP编号,如 :SRP056687,就可以得到数据列表。

这个数据怎么下载呢?
有很多种操作,最方便的是在服务器上使用sra-tools 里的 prefetch命令。另外推荐使用 aspera,比FTP快多了,具体操作后边会介绍。
如何用aspera从NCBI上下载SRA数据
- 获取BioProject
- 获取到下载表单
- aspera下载数据
一、获取BioProject
点击SRA Run Selector 跳转并获取BioProject


二、获取到下载表单
通过在EBI上查询需要下载的list

一定注意在这个地方我通过aspera下载,所以在Read Files处需要勾选几个选项

如果想验证数据完整性,fastq md5也要勾上
下载TSV文件

获取到了TXV文件后我们需要对内容修改成我们需要的格式,首先文件格式是.TXT 使用EXCEL打开,处理成下图所示的文件
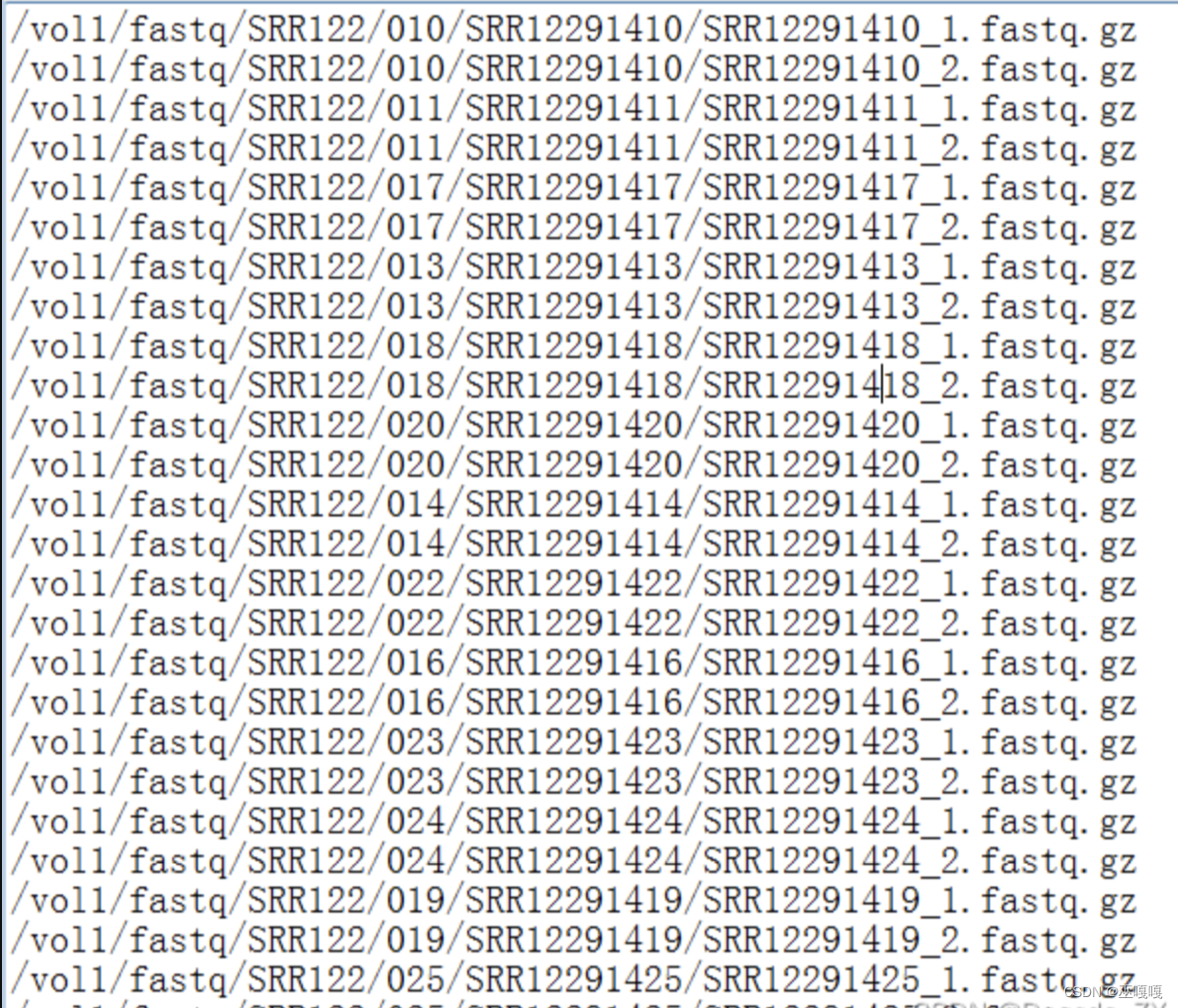
处理步骤如下:
1、打开下载的.TXT文件,图中红色方框中的数据是我们需要的数据,其他列如无需要可以删除。
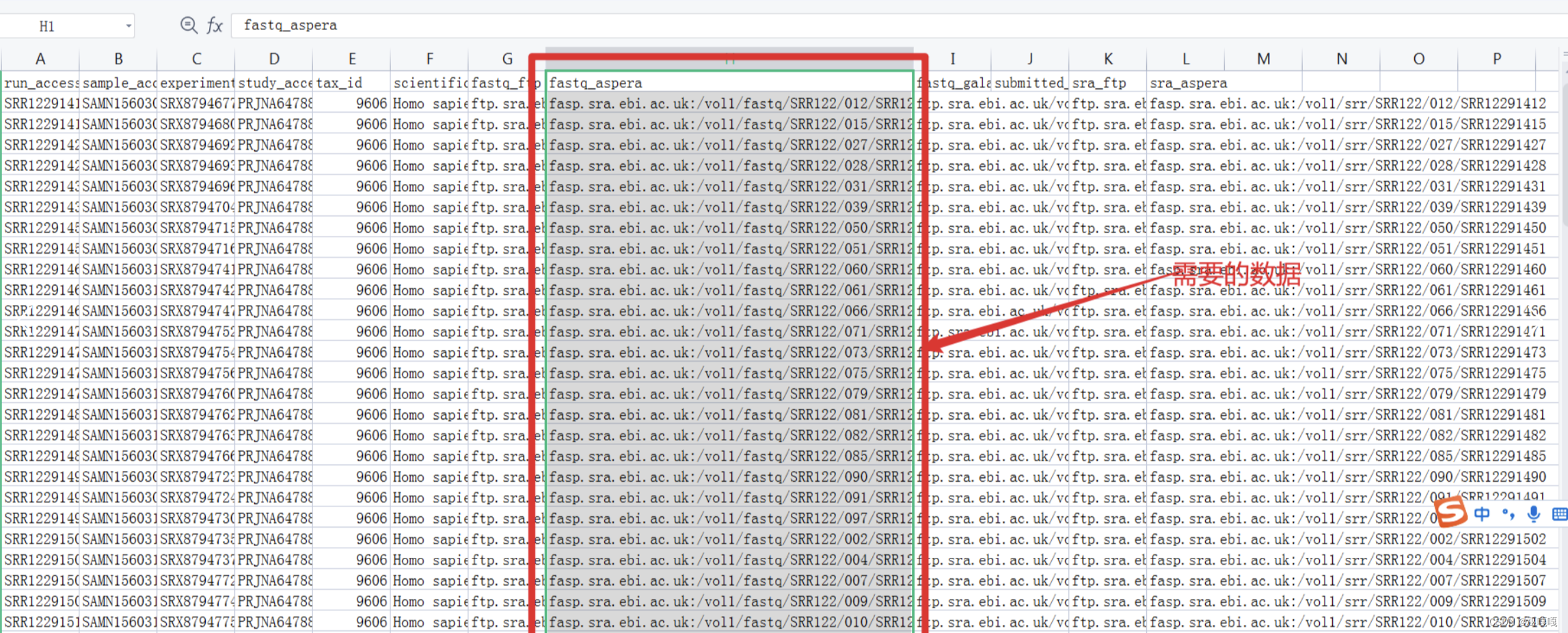
2、用excel将fasp.sra.ebi.ac.uk:直接替换成空


3、将该文件保存为txt格式,使用sed命令把;替换为换行符:sed -i.bak 's/;/\n/g' file_name.txt
三、aspera下载数据
- 简介:Aspera是一款高速传输软件,不受文件大小,网络条件等影响,速度比HTP和FTTP协议快数百倍。Windows和Linux系统均可下载使用。
1.下载Aspera-connec:wget https://download.asperasoft.com/download/sw/connect/3.6.2/aspera-connect-3.6.2.117442-linux-64.tar.gz
2.解压缩:tar zvxf aspera-connect-3.6.2.117442-linux-64.tar.gz
3.运行:sh aspera-connect-3.6.2.117442-linux-64.sh
(此时在home目录下会生成 `.aspera` 的隐藏文件,使用 ls -a 命令可查看)
4.添加环境变量:echo 'export PATH=~/.aspera/connect/bin:$PATH' >>~/.bashrc #正确的添加应该是 vim ~/.bashrc ,然后在最后加上export PATH=“~/.aspera/connect/bin:$PATH”,最后 source ~/.bashrc
5.使其生效:source ~/.bashrc
6.拷贝秘钥文件:cp ~/.aspera/connect/etc/asperaweb_id_dsa.openssh ~/
7.拷贝协议文件:sudo cp ~/.aspera/connect/etc/aspera-license /usr/local/bin/
批量下载数据;准备链接索引文件(sra_list.txt),运行以下代码:
ascp -T -i <path>//asperaweb_id_dsa.openssh -k 1 -l 200m --mode recv --host ftp-private.ncbi.nlm.nih.gov --user anonftp --file-list ./sra_list.txt ./
如需下载后台运行,可以使用 nohup &命令;如需前台转后台运行,查看往期文章:CSDN https://mp.csdn.net/mp_blog/creation/editor/133003205
https://mp.csdn.net/mp_blog/creation/editor/133003205
具体命令如:
nohup ascp -T -i /home/myname/.aspera/connect/etc/asperaweb_id_dsa.openssh -v -k 1 -P33001 -l 200m --mode recv --host fasp.sra.ebi.ac.uk --user era-fasp --file-list /data/myname/feather/datalist/YELLOW/YELLOW_SRR_LIST.txt /data/myname/feather/datalist/YELLOW &Aspera命令行工具的使用:ascp [参数] 目标文件 目的地址
ascp常用参数:
-T ---- 取消加密。若不添加此参数,可能会下载不了。
-i ---- 输入私钥,一般不要少。安装 aspera 后在目录 ~/.aspera/connect/etc/ 下有几个私钥, 使用 linux 服务器的时候一般使用 asperaweb_id_dsa.openssh 文件作为私钥。
-l string ----- 设置最大传输速度,比如设置为 200M 则表示最大传输速度为 200m/s。 若不设置该参数,则一般可达到10m/s的速度,而设置了,传输速度可以更高
--P 用于SSH身份验证的TCP端口,一般是33001
-k ---- 断点续传 ,一般设置为1
-v ---- 可以实时知道程序在做什么,方便查错
-Q --- 一般加上吧
--host=string --- ftp的host名(NCBI的为ftp-private.ncbi.nlm.nih.gov;EBI的为 fasp.sra.ebi.ac.uk)
--user=string --- 用户名(NCBI的为anonftp,EBI的为era-fasp)
--mode=string --- 选择模式,上传为 send,下载为 recv。
--file-list --- 批量下载SRA文件的路径



























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










