🎉 进入生物信息学的世界,与Rosalind一起探索吧!🧬
Rosalind是一个在线平台,专为学习和实践生物信息学而设计。该平台提供了一系列循序渐进的编程挑战,帮助用户从基础到高级掌握生物信息学知识。无论你是初学者还是专业人士,Rosalind都能为你提供适合的学习资源和实践机会。网址:https://rosalind.info
你是否想像专业人士一样分析DNA序列?这里有一个简单的任务来帮助你入门。
我们的第一个挑战:“计数DNA核苷酸” 📊
📝 任务说明: 给定一个DNA字符串,统计每种核苷酸(A、C、G、T)的出现次数。
示例:
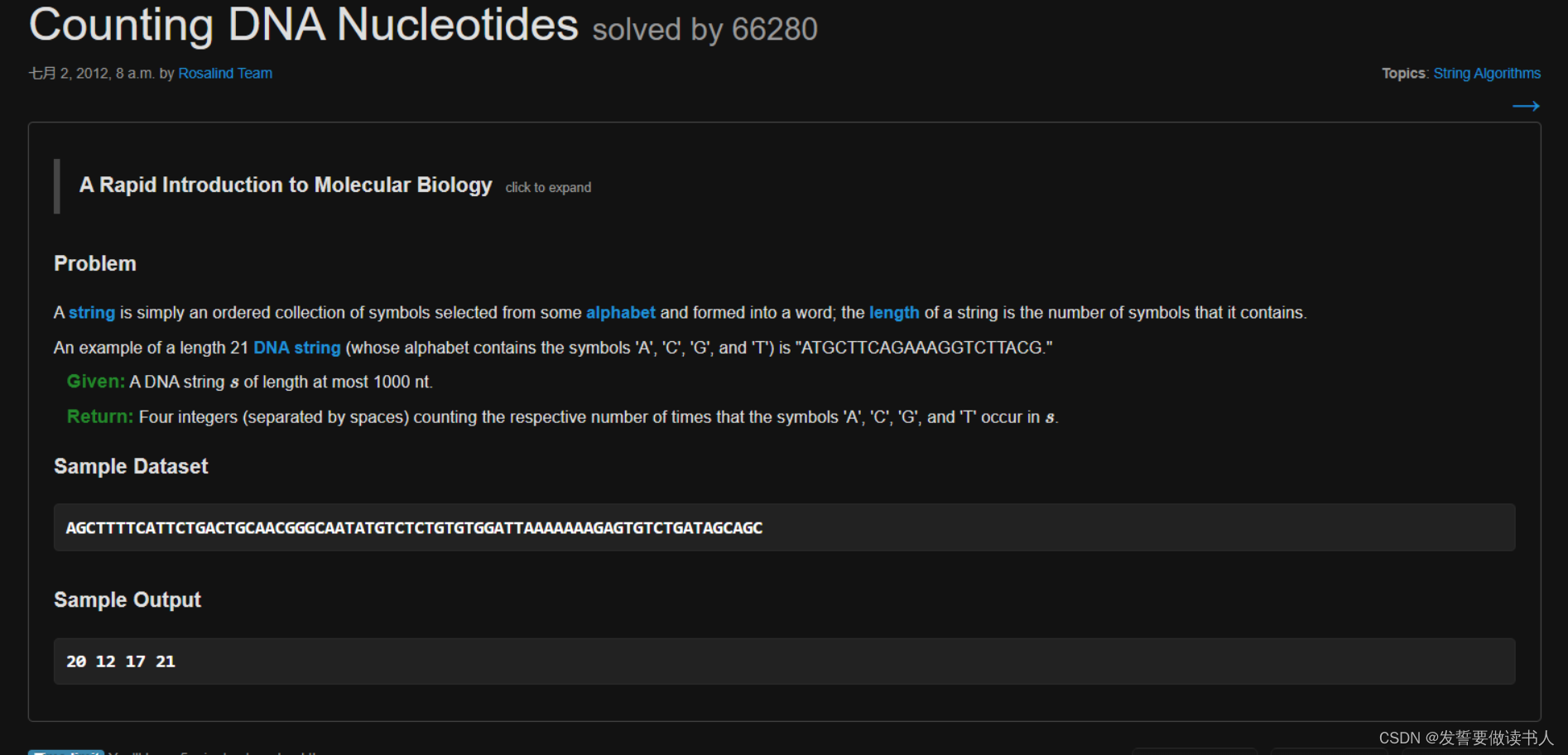
解答:
方法 1:使用循环和条件语句
我们可以通过遍历序列中的每个碱基,并使用条件语句来分别统计每种碱基的数量。这种方法简单直接,但在性能上可能不如其他方法高效。
start_time = time.time()
count_A = 0
count_C = 0
count_T = 0
count_G = 0
for base in Sequence:
if base == "A":
count_A += 1
elif base == "T":
count_T += 1
elif base == "C":
count_C += 1
elif base == "G":
count_G += 1
end_time = time.time()
elapsed_time = end_time - start_time
print(f"Method1 Runtime: 0.0624 seconds")
print(f"A:{count_A} C:{count_C} T:{count_T} G:{count_G}")
方法 2:使用字典存储计数结果
通过遍历序列中的每个碱基,并使用字典来存储每种碱基的数量。这种方法的效率较高,因为字典的查找和更新操作都是常数时间复杂度。
start_time = time.time()
base_dic = {}
for base in Sequence:
if base not in base_dic:
base_dic[base] = 1
else:
base_dic[base] += 1
end_time = time.time()
elapsed_time = end_time - start_time
print(f"Method2 Runtime: 0.0624 seconds")
print(base_dic)
方法 3:使用 collections.Counter
collections.Counter 是一个专门用于计数的字典子类,提供了简单高效的计数方法。
from collections import Counter
start_time = time.time()
base_counter = Counter(Sequence)
end_time = time.time()
elapsed_time = end_time - start_time
print(f"Method3 Runtime: 0.0624 seconds")
print(base_counter)
方法 4:使用字符串的 count 方法
Python字符串的 count 方法可以直接统计子字符串在字符串中出现的次数,这是最简单也是最直观的方法之一。
start_time = time.time()
count_A = Sequence.count("A")
count_C = Sequence.count("C")
count_T = Sequence.count("T")
count_G = Sequence.count("G")
end_time = time.time()
elapsed_time = end_time - start_time
print(f"Method4 Runtime: 0.0624 seconds")
print(f"A:{count_A} C:{count_C} T:{count_T} G:{count_G}")
方法 5:使用 Biopython 库
Biopython 是一个强大的生物信息学库,提供了许多便捷的工具来处理生物序列数据。我们可以使用 Biopython 的 Seq 对象来实现碱基计数。
from Bio.Seq import Seq
sequence = Seq(Sequence)
# 计数碱基
start_time = time.time()
count_A = sequence.count("A")
count_C = sequence.count("C")
count_T = sequence.count("T")
count_G = sequence.count("G")
end_time = time.time()
elapsed_time = end_time - start_time
print(f"Method5 Runtime: 0.0624 seconds")
print(f"A:{count_A} C:{count_C} T:{count_T} G:{count_G}")
比较
使用一个2,704,800个碱基的序列来进行测试,得到结果如下:
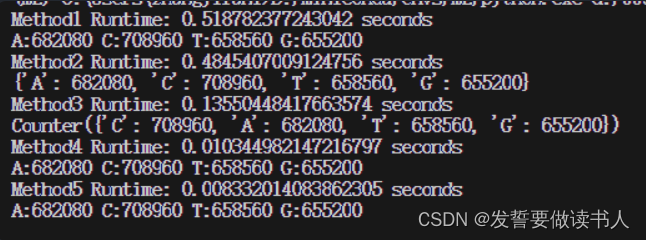
可以看到使用Biopython是最快的,其次是字符串的count方法…
纸上得来终觉浅,绝知此事要躬行。动手练练叭!
之后会坚持更新的,也是锻炼自己的毅力和编程能力,公众号BioYFan

























 252
252

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










