在医学影像的数据存储领域,是存在一定的行业标准的。X光、CT机器等等医疗器械等生产企业,会依据行业标准,对采集的数据进行规范化的存储。
这里面就包括了大名鼎鼎的 DICOM 3.0 协议,上述的摄影形式大部分也都是以这种形式进行存储和传播的。
但是呢,在医学领域进行数据处理的时候,经常会遇到除DICOM外其他的数据形式,比如常见的 .nii、.npy、.mha、.npz,还有就是常见的日常图像存储的 jpeg、png 等等格式。
本文就留作一个笔记本,主要记录上述医学影像的数据存储形式,以及数据间的转换,希望对初次接触的你,有所帮助。

具体介绍,可以参考这里:医疗影像图像格式![]() https://zhuanlan.zhihu.com/p/98551139
https://zhuanlan.zhihu.com/p/98551139
一、nii 3维数组,转2维图像显示、保存
大部分医学领域导出dicom格式,但是太复杂了。很多时候,将dicom转换为nifti(Neuroimaging Informatics Technology Initiative,神经影像信息技术倡议)格式,也就是nii格式进行存储。
nii格式的数据,可以直接在itk-snap软件中打开,直接拖动就可以了。这块我也在我博客的其他文章进行了展示,感兴趣的可以直接去我的主页查看。
这里我们就简单介绍下,存储为nii 3维度格式的文件,是如何拆分为我们容易查看的2维度图像进行保存和显示的。方便有这部分需求的伙伴,转图使用。
注意:如果图像是用作AI训练,普通的png图像和原始的nii存储的3维转2维数据都是可以使用的。一般都会在前处理阶段对数据做归一化操作,所以,我们验证的结果来看,影响不大。
1.1、展示1
这里读取的文件,是一个简单的标注好存储的标签文件,标注软件是上文提到的itk-snap。如果想要获取这个文件,建议找一个CT的dicom的文件,简单标注两个类别,保存下作为读取的Untitled.nii.gz。
完整代码如下:
from nibabel.viewers import OrthoSlicer3D
from nibabel import nifti1
import nibabel as nib
from matplotlib import pylab as plt
import numpy as np
import matplotlib
def show_nii():
# matplotlib.use('TkAgg')
# 需要查看的nii文件名文件名.nii或nii.gz
filename = r'E:\DCM-1\Untitled.nii.gz'
#filename = r'F:\lingjun2019\nodule\MedicalNet\data\MRBrainS18\images\75.nii.gz'
img = nib.load(filename)
# 打印文件信息
print(img)
print(img.dataobj.shape)
#shape不一定只有三个参数,打印出来看一下
width, height, queue = img.dataobj.shape
print(width,height,queue)
# # 显示3D图像
# OrthoSlicer3D(img.dataobj).show()
# # 计算看需要多少个位置来放切片图
interval = 1
x = int((queue/interval) ** 0.5) + 1
num = 1
# 按照10的步长,切片,显示2D图像
plt.figure(figsize=(12, 12))
for i in range(0, queue, interval):
img_arr = img.dataobj[:, :, i]
plt.subplot(x, x, num)
# plt.axis('off') # 去掉坐标轴
plt.title('num:'+str(i))
plt.imshow(img_arr, cmap='gray')
num += 1
plt.show()
if __name__ == "__main__":
show_nii()图像展示结果:一个很亮的,和一个不太亮的,是两个类别标签。其他全黑色的区域,表示没有标注内容。具体的数值我们后面会展示出来理解的。

1.2、展示2
下文 raw_data 文件夹内待读取的 .nii.gz 文件,存储在这里了,方便实验可以直接去下载获取:nii文件raw_data_nii.rar-深度学习文档类资源-CSDN下载![]() https://download.csdn.net/download/wsLJQian/59560970
https://download.csdn.net/download/wsLJQian/59560970
实现功能的完整代码,如下所示。大概步骤包括一下几点:
- load数据
- 截取需要关注的数组区域(这块根据你具体的任务进行设定,比如CT关注肺区,就设定除肺区外的为0)
- 归一化(nii数组的范围比较大,可能是-1000到1000,与图像0-255的范围不匹配)
- 逐层保存
代码如下:
import numpy as np
import nibabel as nib
import h5py
import os
from PIL import Image
img = nib.load(r"F:\TransUNet-main\raw_data\img\0066\DET0000101_avg.nii.gz")
label = nib.load(r"F:\TransUNet-main\raw_data\label\0066\DET0000101_avg_seg.nii.gz")
#Convert them to numpy format,
data = img.get_fdata()
label_data = label.get_fdata()
"""
Convert them to numpy format, clip the images within [-125, 275], normalize each 3D image to [0, 1],
and extract 2D slices from 3D volume for training cases while keeping the 3D volume in h5 format for testing cases.
"""
#clip the images within [-125, 275],
data_clipped = np.clip(data, -125, 275)
#normalize each 3D image to [0, 1], and
data_normalised = (data_clipped - (-125)) / (275 - (-125))
#extract 2D slices from 3D volume for training cases while
# e.g. slice 000
for i in range(data_clipped.shape[2]):
formattedi = "{:03d}".format(i)
slice000 = data_normalised[:,:,i]*255
np.savetxt(r"label.txt", label_data[:, :, 6], delimiter=',', fmt='%5s')
label_slice000 = label_data[:, :, i] * 255
print(slice000.shape, type(slice000))
image = Image.fromarray(slice000)
image = image.convert("L")
label = Image.fromarray(label_slice000)
label = label.convert("L")
image.save("./chestminist/" +"DET0000101_avg"+ str(i) + ".png")
label.save("./chestminist/" + "DET0000101_avg" + str(i) + "_label.png")更多问题,参考这里:Request preliminary data · Issue #24 · Beckschen/TransUNet · GitHub
展示结果:读懂上述代码,建议打开保存的 label.txt 文件,阅读像素信息,我们还是先看下第6层的像素信息(如果你第六层没有标注结果,看不到大于0的数)

保存的图像:

二、读取和保存 .nii 文件
2.1、读取和保存
ITK( Insight Segmentation and Registration Toolkit)是美国国家卫生院下属的国立医学图书馆开发的一款医学图像处理软件包,是一个开源的、跨平台的影像分析扩展软件工具。
读取和存储的操作代码如下:
import itk
dicom_directory = "此处为全部.dcm文件所在的文件夹或.nii单个序列文件"
reader = itk.imread(dicom_directory) # 读取nii文件
itk.imwrite(ct, r'F:\outputs\c.nii') # 保存nii文件
2.2、nii 文件直观展示
接下来,给出一个可能使用的场景:你预测了一个疾病,然后又想把它框定在一个范围内,比如肺区。
你就可以采用mask乘积的形式,去除掉不在肺区内的部分。因为
- 肺区内为1
- 肺区外为0。
主要代码如下:
import itk
def itk_saveNII():
img = itk.imread(r"F:\outputs\1.nii")
mask = itk.imread(r"F:\outputs\1_LungMask.nii")
queue, width, height = img.shape
print(width, height, queue)
masks = []
for i in range(queue):
img_arr = img[i, :, :]
mask_arr = mask[i, :, :]
# c = np.rot90(img_arr*mask_arr, k = 1)
# c = c[::-1]
c = img_arr*mask_arr
masks.append(c)
ctarr = np.stack(masks, axis=0)
ct = itk.image_from_array(ctarr)
itk.imwrite(ct, r'F:\outputs\c.nii')下面图做个展示,主要是使用 itk-snap 进行验证保存的数组,是否正确,如下所示:
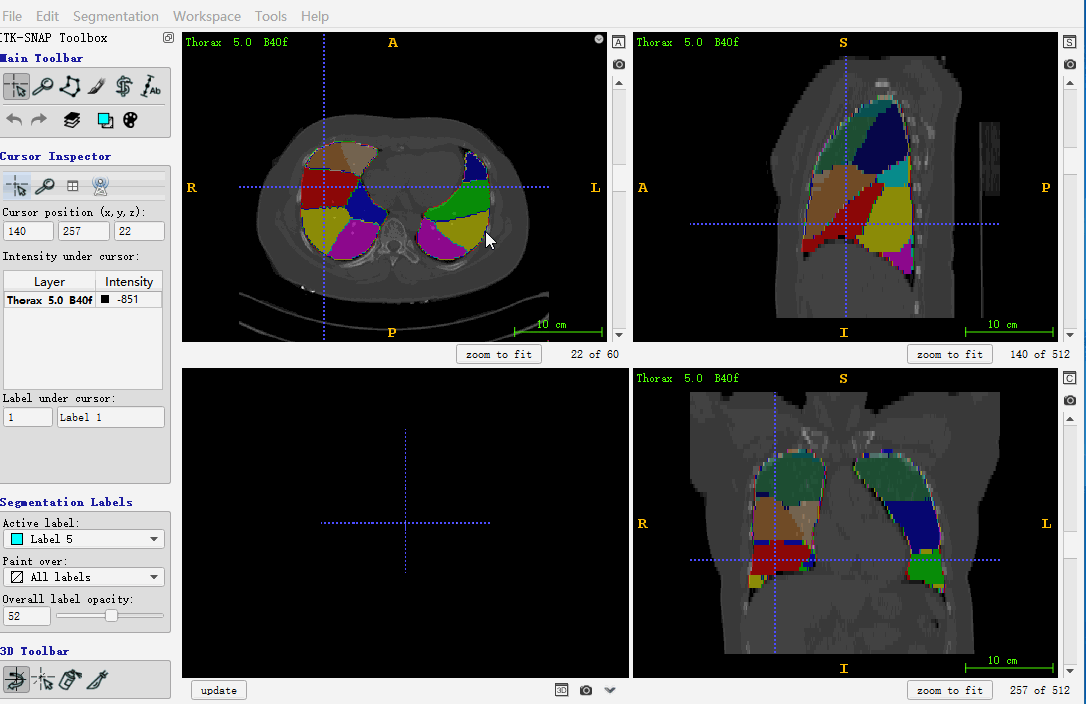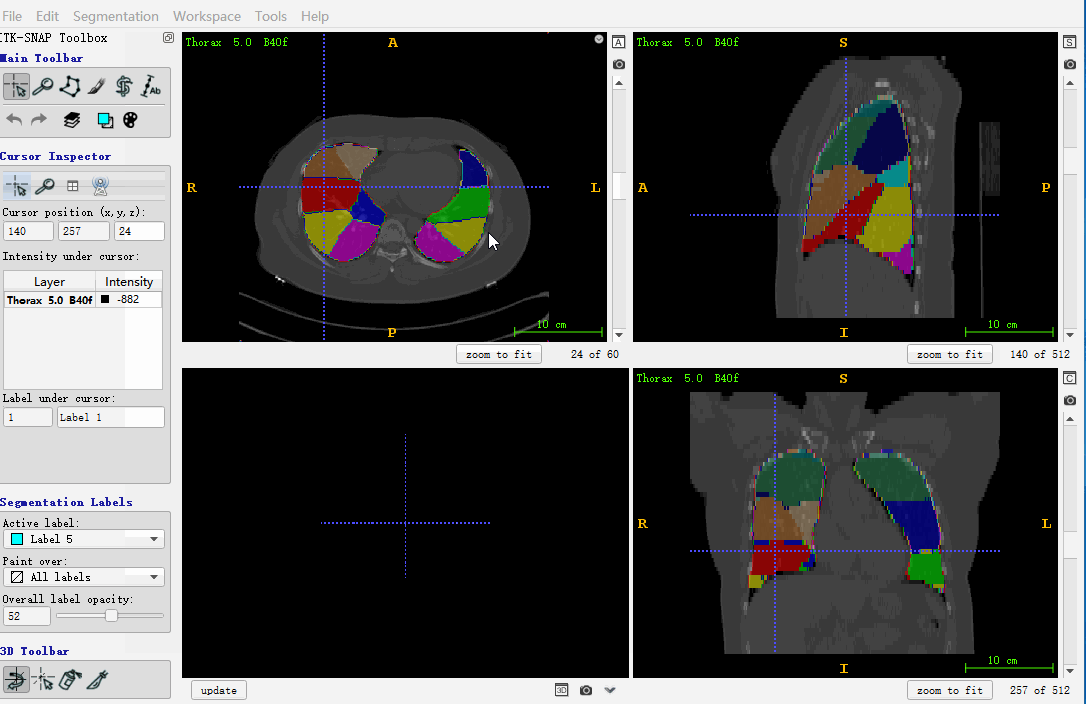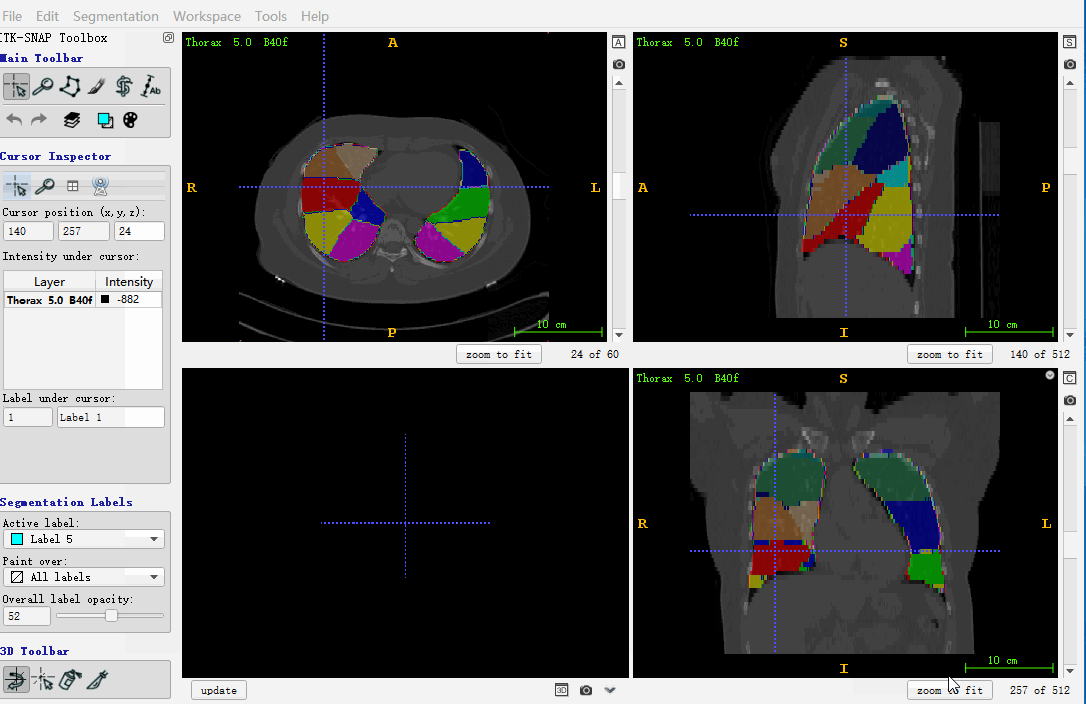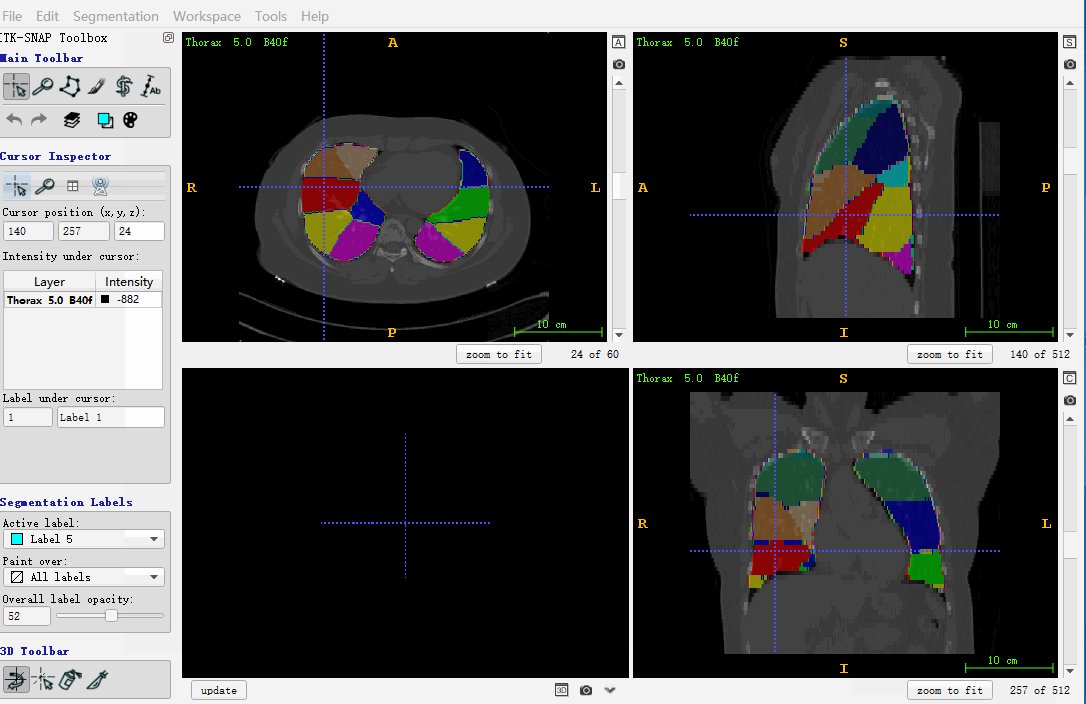
也可以采用 nibabel 进行打开,这里暂留笔记,不用着重看。
import itk
import nibabel as nib
def save_nii():
img = nib.load(r"F:\outputs\1.nii")
mask = nib.load(r"F:\outputs\1_LungMask.nii")
width, height, queue = img.dataobj.shape
print(width, height, queue)
masks = []
for i in range(queue):
img_arr = img.dataobj[:, :, i]
mask_arr = mask.dataobj[:, :, i]
c = np.rot90(img_arr*mask_arr, k = 1) # 逆时针旋转1次90度
c = c[::-1] # 左右镜像
masks.append(c)
ctarr = np.stack(masks, axis=0) # 按行堆叠数组
ct = itk.image_from_array(ctarr)
itk.imwrite(ct, r'F:\outputs\c.nii') # 保存nii文件主要内容是保存为nii格式的文件,顺便将两个 nii 文件进行如下操作:
- 乘积合并
- 旋转
- 左右镜像
2.3、SimpleITK 和 itk 库数组的差异
在日常操作中,发现上述两个库都是可以对nii文件进行操作的,但是两个库读取后的数组,存在差异,具体我们可以通过下面的介绍,进行理解。
先对两个库读取同一个nii文件如下:
import os
import SimpleITK as sitk
import itk
nii_path = os.path.join(r'./images', 'brain.nii.gz')
image = sitk.ReadImage(nii_path)
print('image size:', image.GetSize()) # depth、height、width
imgs = itk.array_from_image(itk.imread(nii_path))
print('img shape:', imgs.shape) # width、height、depth打印结果:
image size: (224, 256, 192)
img shape: (192, 256, 224)为什么两个库读取的结果,会有差异呢?
- 在nii文件中,像素的排列顺序是从头到尾按顺序排列的,即最外层是z轴方向,然后是y轴方向,最内层是x轴方向。
- 但是在读取nii文件时,有些库会将这个排列顺序调整为从内到外的顺序,即最内层是z轴方向,然后是y轴方向,最外层是x轴方向。
- 这是因为不同的库对于数组的存储方式不同,一些库使用C语言的方式进行存储,而C语言中数组的存储方式是从内到外的。
- 在SimpleITK中,采用的是C++的方式进行存储,因此读取出来的数组顺序是从内到外的;
- 而在itk中,采用的是C语言的方式进行存储,因此读取出来的数组顺序是从外到内的。
这个顺序的不同可能会导致一些问题,因此在使用不同的库读取nii文件时,需要注意数据的存储顺序。
三、npz 2 维数组转图像显示、保存
备注:下面用到的数据集是 Synapse multi-organ segmentation dataset。官方公开数据集下载地址的:Synapse | Sage Bionetworks,我就是这样下载到的,论文:TransUNet: Transformers Make Strong Encoders for Medical Image Segmentatihttps://arxiv.org/pdf/2102.04306.pdf![]() https://arxiv.org/pdf/2102.04306.pdf里面有讲到。下载慢的也可以在这里下载:https://download.csdn.net/download/wsLJQian/40704530
https://arxiv.org/pdf/2102.04306.pdf里面有讲到。下载慢的也可以在这里下载:https://download.csdn.net/download/wsLJQian/40704530![]() https://download.csdn.net/download/wsLJQian/40704530PS项目及数据:
https://download.csdn.net/download/wsLJQian/40704530PS项目及数据:
代码部分:
import numpy as np
from PIL import Image
import os
label_path=r'F:\project_TransUNet\data\Synapse\train_npz'
filename = "case0006_slice088.npz"
data = np.load(os.path.join(label_path, filename))
print(data.files)
x_train=data["image"]
label_train=data["label"]
print(x_train.shape, type(x_train))
print(label_train.shape, type(label_train))
np.savetxt(r"label.txt", label_train, delimiter=',', fmt='%5s')
image = Image.fromarray(x_train*255)
max_pix = np.amax(label_train)
label_train = label_train / max_pix # 归一化
label = Image.fromarray(label_train * 255)
image = image.convert("L")
label = label.convert("L")
print(image.size)
image.save(r"E:\temp\image/"+filename.replace('.npz', '.png'))
label.save(r"E:\temp\image/" + filename.replace('.npz', 'label.png'))保存的 tx t内容,会发现有2和5,思考2和5分别表示什么?这是标签文件存储的数组,那就是2是一个类,5是一个类。
同理,你也可以保存下图像的数组看看,看看它里面都是存储多大的数值。

保存的图像和标签内容如下所示:

打印内容:
['image', 'label']
(512, 512) <class 'numpy.ndarray'>
(512, 512) <class 'numpy.ndarray'>
(512, 512)保存结果:
进行归一化的目的是因为:在制作标签时候,不同的类采用不同的数字,替换相对应的像素点。第一类用1,第二类用2,依次类推。0类表示背景,这样一种mask图就将囊括了多目标分割
备注:完成数据我也不知道叫什么名字了,主要就是关于CT分割的数据集,标签都已经标注好了。
鉴于各位小伙伴在评论区的留言,我已经将目前我下载的数据上传到了CSDN,地址这里: https://download.csdn.net/download/wsLJQian/40704530![]() https://download.csdn.net/download/wsLJQian/40704530 有需要的自取哦。
https://download.csdn.net/download/wsLJQian/40704530 有需要的自取哦。
四、npy转图像保存
前面两个看到,学习懂了,其实其他的三维数组的读取和操作,都是大同小异,甚至的一样的。就是保存的后缀可能存在一丝的不同,.npy 文件的处理也一样。
import os
import cv2
import numpy as np
from matplotlib import pyplot as plt
import scipy.misc
from matplotlib.pyplot import cm
def load_npy():
npy_path = r"F:\datasets\test\bbox_mask\1.3.6.1.4.1.14519.5.2.1.6279.6001.100225287222365663678666836860.npy"
save_path = r"F:\datasets\test\mask"
label_array = np.load(npy_path)
print(type(label_array), label_array.shape)
for i in range(label_array.shape[-1]): # 循环数组 最大值为图片张数(我的是200张) 三维数组分别是:图片张数 水平尺寸 垂直尺寸 C,W,H
arr = label_array[:, :, i] # 获得第i张的单一数组
print(arr.shape)
# disp_to_img = scipy.misc.imresize(arr*255, [512, 512]) # 根据需要的尺寸进行修改
plt.imsave(os.path.join(save_path, "{}_disp.png".format(i)), arr, cmap=cm.gray) # 定义命名规则,保存图片为彩色模式
print('photo {} finished'.format(i))保存图像结果展示:

五、dicom 转 nii 数据处理(多进程批处理)
到这里就不同了,这里就聊到了医学影像中最常见到的数据形式:DICOM。在进行3维训练时候,有时候需要将医学dicom文件,转为 nii 三维数据形式保存。此处即是处理上述情况的数据转换:
特点:
- 根据 Instance number 排序的切片,与dicom查看器看到的顺序是一致的;
- nii.gz文件内包含dicom原始的一些信息,包括spacing等等
增加了多进程,代码如下:
import SimpleITK as sitk
import os
import pandas as pd
def _get_instance_number(dicom_path):
img_reader = sitk.ImageFileReader()
img_reader.SetFileName(dicom_path)
img_reader.LoadPrivateTagsOn()
img_reader.ReadImageInformation()
number_str = img_reader.GetMetaData('0020|0013') # 获取Instance number
return int(number_str)
def get_slice(dcm_path):
# 用这个函数获得按照Instance number排序的切片路径
reader = sitk.ImageSeriesReader()
dicom_names = reader.GetGDCMSeriesFileNames(dcm_path) # 获得切片路径,这个是按切片命名排序的
r = []
for name in dicom_names:
r.append({"instance_number": _get_instance_number(name), "dcm_name": name})
r = pd.DataFrame(r)
r = r.sort_values("instance_number") # 按照Instance number排序
r = tuple(r["dcm_name"]) # 获得按照Instance number排序的切片路径
return r
def dcm2nii(series_path, nii_dir):
dcms_path = os.path.join(series_path, 'dcm')
nii_path = os.path.join(nii_dir, series_path.split('/')[-1]+'.nii.gz')
print('dcms_path:', dcms_path, nii_path)
# 1.构建dicom序列文件阅读器,并执行(即将dicom序列文件“打包整合”)
reader = sitk.ImageSeriesReader()
dicom_names = get_slice(dcms_path)
reader.SetFileNames(dicom_names)
image2 = reader.Execute()
# 2.将整合后的数据转为array,并获取dicom文件基本信息
image_array = sitk.GetArrayFromImage(image2) # z, y, x
origin = image2.GetOrigin() # x, y, z
# print(origin)
spacing = image2.GetSpacing() # x, y, z
# print(spacing)
direction = image2.GetDirection() # x, y, z
# print(direction)
# 3.将array转为img,并保存为.nii.gz
image3 = sitk.GetImageFromArray(image_array) # 这些需要设置一下,不然z轴的空间信息可能会被压缩
image3.SetSpacing(spacing)
image3.SetDirection(direction)
image3.SetOrigin(origin)
sitk.WriteImage(image3, nii_path)
print('save end')
import os
import multiprocessing as mp
def collect_all_paths(checked_dir):
files_list = []
for ini_folder in checked_dir:
for patient in os.listdir(ini_folder):
dicom_dir = os.path.join(ini_folder, patient)
files_list.append(dicom_dir)
print(len(files_list), len(set(files_list)))
# assert len(files_list)==len(set(files_list))
return files_list
def get_path(queue, save_dir):
while True:
try:
idx, img_path = queue.get()
dcm2nii(img_path, save_dir)
except:
pass
def provide_path(queue, queue_idx_path):
while True:
idx, img_path = queue_idx_path.get()
queue.put((idx, img_path))
def run():
checked_dir = [r'./database-train/checked',
r'./database-train/checked2']
save_dir = r'./nii_file_all'
# 遍历文件夹下所有文件
files = collect_all_paths(checked_dir)
print('ok')
mp.set_start_method('spawn')
queue_img = mp.Queue(8)
queue_idx_path = mp.Queue(len(files))
[queue_idx_path.put(idx_path) for idx_path in enumerate(files)]
processes = list()
processes.extend([mp.Process(target=get_path, args=(queue_img, save_dir)) for _ in range(8)])
processes.extend([mp.Process(target=provide_path, args=(queue_img, queue_idx_path))])
[setattr(process, "daemon", True) for process in processes]
[process.start() for process in processes]
[process.join() for process in processes]
if __name__ == '__main__':
run()
这里是以CT结节的数据作为依据的,这个有公开的数据集,叫做 LIDC- IDRI,需要的可以去下载。如果自己有dicom的文件,到不妨先修改下代码,转一个试试。
转好了,如何验证转好的文件,是不是正确的呢?还是itk-snap这个标注软件,分别拖动dicom文件到 itk-snap 进行显示,再拖动转好的图,进行展示。
对比下就知道转图是否存在问题了。
六、png 转 nii 保存、显示
有时候拿到的图像并不是原始的图像,是经过一次处理的,已经是 png或jpeg 之类的图像。比如很多国外的开源医学数据,就是转好图的形式。
这个时候我们还想要恢复它原始的模样,那就需要将它反向的,堆叠到一起,再存储。
下面直接放代码,看效果,后面再详述过程,代码如下:
label_dict = {'Clear Label': '0', '肺结核': '1', '结节':'2'}
def draw_mask_label(label_path, file, mask_dir):
with open(label_path, 'r', encoding='utf8')as fp:
label_info = json.load(fp)
shapes = label_info['shapes']
mask_temp = np.zeros((512, 512))
for i in range(len(shapes)):
points = shapes[i]["points"]
label = shapes[i]["label"]
pixel_num = int(label_dict[label])
contour = np.array([points])
contour = np.trunc(contour).astype(int)
cv2.drawContours(mask_temp, [contour], 0, (pixel_num), cv2.FILLED)
cv2.imwrite(os.path.join(mask_dir, file.replace('.dcm', '.png')), mask_temp)
def genarete_zeromask_label(file, mask_dir):
mask_temp = np.zeros((512, 512))
mask_path = os.path.join(mask_dir, file.replace('.dcm', '.png'))
print('mask_path:', mask_path)
cv2.imwrite(mask_path, mask_temp)
def get_SequenceBool(src_dir, list_filename):
plain_sequence = False
slice0 = pydicom.read_file(os.path.join(src_dir, list_filename[0]))
slice1 = pydicom.read_file(os.path.join(src_dir, list_filename[1]))
if slice1.ImagePositionPatient[-1] > slice0.ImagePositionPatient[-1]:
plain_sequence = True
else:
pass
return plain_sequence
def rename_dcm(dcm_dir, json_dir):
for file in os.listdir(dcm_dir):
InstanceNumber = str(len(os.listdir(dcm_dir)) - int(file.split('_')[-1].split('.dcm')[0]) + 1)
InstanceNumber = (6 - len(InstanceNumber)) * '0' + InstanceNumber
new_Dcmname = file.split('_')[0] + '_' + file.split('_')[1] + '_' + InstanceNumber + '-0.dcm'
print(file, new_Dcmname)
os.rename(os.path.join(dcm_dir, file), os.path.join(dcm_dir, new_Dcmname))
json_path = os.path.join(json_dir, file.replace('.dcm', '.json'))
if os.path.exists(json_path):
new_Jsonname = file.split('_')[0] + '_' + file.split('_')[1] + '_' + InstanceNumber + '-0.json'
print(json_path, new_Jsonname)
os.rename(json_path, os.path.join(json_dir, new_Jsonname))
def main_json2nii():
data_dir = r'W:\tmp'
for patient in os.listdir(data_dir):
print(patient)
patient_dir = os.path.join(data_dir, patient)
dcm_dir = os.path.join(data_dir, patient, 'dcm')
json_dir = os.path.join(data_dir, patient, 'json')
mask_dir = os.path.join(data_dir, patient, 'mask')
list_filename = os.listdir(dcm_dir)
plain_sequence = get_SequenceBool(dcm_dir, list_filename)
if not plain_sequence:
rename_dcm(dcm_dir, json_dir)
if not os.path.exists(mask_dir):
os.makedirs(mask_dir)
for file in os.listdir(dcm_dir):
print(file)
json_path = os.path.join(json_dir, file.replace('.dcm', '.json'))
if os.path.exists(json_path):
draw_mask_label(json_path, file, mask_dir)
else:
genarete_zeromask_label(file, mask_dir)
allImg_mask = png2nii(mask_dir)
save_array_as_nii_volume(allImg_mask, os.path.join(patient_dir, patient + '.nii'))
print(np.shape(allImg_mask))
if __name__ == '__main__':
main_json2nii()显示内容:

这里解释下,上面一大串代码都干了啥:
- 有dcm文件和json标注文件
- 判断dcm文件沿Z轴方向,是否从小到大排列。这里主要是考虑itk-snap标注显示
- 如果反序,重新对dcm文件排序,json排序(json的排序对号是重点)
- 生成json文件对应的mask(这里未考虑序号大于255的情形,一般也没有那么多类)
- 最后生成nii文件,用itk-snap标注软件打开显示
代码引入头我就不写了,跟前面是差不多的,相信你们也知道怎么添加。关注持续更新中
七、dcm 文件读取
dicom 数据存储形式是一个存储信息比较丰富的文件格式,也是医学领域专业的数据格式。存储的不仅仅有图像数组信息,还有其他相关的信息。比如拍摄日期、拍摄条件、患者信息等等。
更多关于dicom文件的信息内容介绍,和为什么这样转图,可以参考这里详细学习:
这里做一个简单的案例,或许dicom文件内存储的患者ID、性别和年龄。如下所示:
def get_dicomInfo():
img_PATH = r"./test"
for path, dirs, files in os.walk(img_PATH):
for filename in files: # 遍历所有文件
file_path = os.path.join(path, filename)
ds = pydicom.dcmread(file_path, force=True) # 读取dcm
patient_id = str(ds.PatientID)
patient_sex = str(ds.PatientSex)
patient_age = str(ds.PatientAge)八、mhd+raw 文件转图
mhd 是医学影像的信息文件,要查看信息可以使用文本编辑器打开,如 notepad++,要查看图像可以使用 Fiji 打开。与raw文件放到一起存储:
- mhd: 存储影像信息,
- raw: 存储影像。
这里以 LUNA16 肺结节为例,展示如下:
import SimpleITK as sitk
def load_itk_image(filename):
"""Return img array and [z,y,x]-ordered origin and spacing
"""
itkimage = sitk.ReadImage(filename)
numpyImage = sitk.GetArrayFromImage(itkimage)
numpyOrigin = np.array(list(reversed(itkimage.GetOrigin())))
numpySpacing = np.array(list(reversed(itkimage.GetSpacing())))
return numpyImage, numpyOrigin, numpySpacing
def load_mhd():
numpyImage, numpyOrigin, numpySpacing = load_itk_image(r'./luna16/seg-lungs-LUNA16/1.3.6.1.4.1.14519.5.2.1.6279.6001.100225287222365663678666836860.mhd')
print(numpyOrigin, numpySpacing)
print(numpyImage.shape)
# print(numpyImage)
save_path = r"./tmp/res/lung_mask"
for i in range(numpyImage.shape[0]): # z, y, x
arr = numpyImage[i, :, :] # 获得第i张的单一数组
print(arr.shape)
print(arr[256])
plt.imsave(os.path.join(save_path, "{}_mask.png".format(i)), arr, cmap='gray')
print('photo {} finished'.format(i))图像保存结果如下所示:

取 z=150,y=256 的一行拿出来查看,数组如下所示:
[0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4
4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4
4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4
4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 4 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 5 5 5 5 5 5 5 5 5 5 5 5 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3
3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3
3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3
3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3 3
3 3 3 3 3 3 3 3 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0]这我才知道:
- 3-左肺、
- 4-右肺、
- 5-气管 。
九、dcm 转 mhd 和 raw 文件
前面 mhd+raw 可以转图,包含了影像和描述影像参数的信息。而dicom文件,恰好也是一个可以同时存储这两个信息的文件。
与此同时,我们知道dicom文件的存储是比较占空间的,那能不能通过将dicom文件,分别转储为mhd和raw文件,分开储存呢?
import cv2
import os
import pydicom
import numpy
import SimpleITK
def dcm2mhd_raw():
data_dir = r"Z:\tmp\raw/"
SaveRawDicom = r"Z:\tmp\mhd/" # 与python文件同一个目录下的文件夹,用来存储mhd文件和raw文件
for patient in os.listdir(data_dir):
# 路径和列表声明
PathDicom = os.path.join(data_dir, patient, 'dcm') # 与python文件同一个目录下的文件夹,存储dicom文件
if os.path.exists(PathDicom):
lstFilesDCM = []
# 将PathDicom文件夹下的dicom文件地址读取到lstFilesDCM中
for dirName, subdirList, fileList in os.walk(PathDicom):
for filename in fileList:
if ".dcm" in filename.lower(): # 判断文件是否为dicom文件
print(filename)
lstFilesDCM.append(os.path.join(dirName, filename)) # 加入到列表中
print(patient)
print('\n')
# 第一步:将第一张图片作为参考图片,并认为所有图片具有相同维度
RefDs = pydicom.read_file(lstFilesDCM[0]) # 读取第一张dicom图片
# 第二步:得到dicom图片所组成3D图片的维度
ConstPixelDims = (int(RefDs.Rows), int(RefDs.Columns), len(lstFilesDCM)) # ConstPixelDims是一个元组
# 第三步:得到x方向和y方向的Spacing并得到z方向的层厚
ConstPixelSpacing = (float(RefDs.PixelSpacing[0]), float(RefDs.PixelSpacing[1]), float(RefDs.SliceThickness))
# 第四步:得到图像的原点
Origin = RefDs.ImagePositionPatient
# 根据维度创建一个numpy的三维数组,并将元素类型设为:pixel_array.dtype
ArrayDicom = numpy.zeros(ConstPixelDims, dtype=RefDs.pixel_array.dtype) # array is a numpy array
# 第五步:遍历所有的dicom文件,读取图像数据,存放在numpy数组中
i = 0
for filenameDCM in lstFilesDCM:
ds = pydicom.read_file(filenameDCM)
ArrayDicom[:, :, lstFilesDCM.index(filenameDCM)] = ds.pixel_array
# cv2.imwrite("out_" + str(i) + ".png", ArrayDicom[:, :, lstFilesDCM.index(filenameDCM)])
i += 1
# 第六步:对numpy数组进行转置,即把坐标轴(x,y,z)变换为(z,y,x),这样是dicom存储文件的格式,即第一个维度为z轴便于图片堆叠
ArrayDicom = numpy.transpose(ArrayDicom, (2, 0, 1))
# 第七步:将现在的numpy数组通过SimpleITK转化为mhd和raw文件
sitk_img = SimpleITK.GetImageFromArray(ArrayDicom, isVector=False)
sitk_img.SetSpacing(ConstPixelSpacing)
sitk_img.SetOrigin(Origin)
SimpleITK.WriteImage(sitk_img, os.path.join(SaveRawDicom, patient + ".mhd"))十、nrrd文件转png图片
import nrrd
def load_nrrd():
mask, _ = nrrd.read(r'Z:\tmp\preprocessed_test\1\CT1893023-1_mask.nrrd')
print(type(mask), mask.shape)
for i in range(mask.shape[0]):
arr = mask[i, :, :] # 获得第i张的单一数组
save_path =r'Z:\tmp\preprocessed_test\1\mask'
if not os.path.exists(save_path):
os.makedirs(save_path)
plt.imsave(os.path.join(save_path, "{}.png".format(i)), arr, cmap='gray') # 定义命名规则,保存图片为彩色模式
print('photo {} finished'.format(i))十一、Dicom文件添加内容重写入
dicom文件是一个规范性的存储格式。那能不能通过操作,将一些附加的信息,添加到dicom文件内呢?简言之就是实现了对dicom文件的修改。
下面展示了如何对dicom文件的图像部分进行修改。当然,也可以对附加信息部分进行修改,比如改写studyDate。后半部分比较简单,这里不再赘述了。着重介绍前半部分修改影像吧。
# 获取dcm文件的pixel_array
filename_dcm = "a.dcm"
ds = pydicom.dcmread(os.path.join(dcm_Dir, filename_dcm)) # 读取dcm
img_dcm = np.array(ds.pixel_array)
# 采用OpenCV方式,对pixel_array进行划直线操作
cv2.line(img_dcm, (x1, line1_y), (raw_w-x1, line1_y), (0, 0, 255))
cv2.line(img_dcm, (x1, line2_y), (raw_w-x1, line2_y), (0, 0, 255))
# 将修改后的pixel_array,重新保存到dcm文件内,其他内容不发生改变
ds.PixelData = img_dcm.tobytes()
ds.Rows, ds.Columns = img_dcm.shape
ds.save_as('a_r.dcm')
print("File saved.")microDicom软件打开,前后的展示如下:

十二、普通图像文件(png)存储为Dicom文件
既然dicom文件可以另存为普通的png文件,那么,png文件是不是也可以存储为dicom文件呢?通过前面对raw和mhd文件分别存储信息的学习,知道肯定是可以的。
import os
import datetime
import numpy as np
from PIL import Image
import pydicom
from pydicom.dataset import Dataset, FileDataset
filename_little_endian ='bb.dcm'
filename_big_endian = 'testr.dcm'
def save_to_dcm(image_path):
img = np.asarray(Image.open(image_path).convert("L"), dtype=np.uint8)
print(img.shape)
print("Setting file meta information...")
# Populate required values for file meta information
file_meta = Dataset()
file_meta.MediaStorageSOPClassUID = '1.2.840.10008.5.1.4.1.1.2'
file_meta.MediaStorageSOPInstanceUID = "1.2.3"
file_meta.ImplementationClassUID = "1.2.3.4"
print("Setting dataset values...")
# Create the FileDataset instance (initially no data elements, but file_meta
# supplied)
ds = FileDataset(filename_little_endian, {},
file_meta=file_meta, preamble=b"\0" * 128)
# # Write as a different transfer syntax XXX shouldn't need this but pydicom
# # 0.9.5 bug not recognizing transfer syntax
ds.file_meta.TransferSyntaxUID = pydicom.uid.ImplicitVRLittleEndian
# Set creation date/time
dt = datetime.datetime.now()
ds.ContentDate = dt.strftime('%Y%m%d')
timeStr = dt.strftime('%H%M%S.%f') # long format with micro seconds
ds.ContentTime = timeStr
ds.Modality = "US"
ds.PatientName = "ZhangSan"
ds.patientAge = "105"
ds.SeriesInstanceUID = pydicom.uid.generate_uid()
ds.StudyInstanceUID = pydicom.uid.generate_uid()
ds.FrameOfReferenceUID = pydicom.uid.generate_uid()
ds.BitsStored = 8
ds.BitsAllocated = 8
ds.SamplesPerPixel = 3
ds.HighBit = 7
ds.PlanarConfiguration = 0
ds.InstanceNumber = 1
ds.ImagePositionPatient = r"0\0\1"
ds.ImageOrientationPatient = r"1\0\0\0\-1\0"
ds.ImagesInAcquisition = "1"
ds.Rows, ds.Columns = img.shape[:2]
ds.PixelSpacing = [1, 1]
ds.PixelRepresentation = 0
ds.PixelData = img.tobytes()
# import pdb;pdb.set_trace()
# ds.PhotometricInterpretation = 'RGB'
print("Writing file", 'test.dcm')
ds.save_as('test.dcm')
print("File saved.")
if __name__ == '__main__':
image_path = r"00d35ceb3c14c3318f4f3ec998879827.jpeg"
save_to_dcm(image_path)可以采用 microDicom 文件直接打开,且进行调窗。就是动态范围太少了,调整不太好。

自己写的目的,也是为了能够自己记录。在学习工作中,偶尔也会重复用到,想到之前做过,然后查起来也很方便。收到打赏,甚是开心,感谢感谢,继续努力。
如果您觉得本篇文章对你有帮助,欢迎点赞,让更多人看到,这是对我继续写下去的鼓励。如果能再点击下方的红包打赏,给博主来一杯咖啡,那就太好了。























 3754
3754











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










