实验一:实验平台及环境安装
课程:数据挖掘(Data Mining)
操作系统:Windows 11
1. 实验目的
- 掌握在 Windows 平台下安装与配置 Python 环境的方法。
- 掌握 Anaconda 的安装、环境管理与常用命令。
- 能够创建独立 Python 环境(本实验要求:创建名为 DM 的 Python 3.7 环境)。
- 熟练安装 PyCharm、Jupyter Notebook 等常用开发工具。
- 能够安装 PyTorch 2.2.2 及常见第三方库(numpy、pandas、tensorflow、h5py、mygene、matplotlib、seaborn、umap-learn)。
- 熟悉 pip 与 conda 的软件包管理方式。
2. 实验环境
- Windows 11
- Anaconda(Python 发行版)
- PyCharm(Community 或 Professional)
- Jupyter Notebook
- Python 3.7
- PyTorch 2.2.2(CPU/GPU 版本均可)
3. 实验内容与步骤
3.1 安装 Anaconda
- 从官网下载适用于 Windows 的 Anaconda 安装包。
- 双击安装程序,按默认设置安装
- 勾选 Add Anaconda to my PATH(可选)。
- 推荐使用 Anaconda Prompt 管理环境。
- 安装完成后,打开 Anaconda Prompt 验证:
conda --version
![![[Pasted image 20251128224049.png]]](https://i-blog.csdnimg.cn/direct/aa4ec57cbfa440deb3ced66675000db7.png)
由于之前已经安装过,所以版本较旧。
3.2 创建 Python 环境 DM(Python 3.7)
在 Anaconda Prompt 中输入:
conda create -n DM python=3.7 -y # 创建名为 DM 的 Python3.7 环境
conda activate DM # 激活环境
![![[Pasted image 20251128225214.png]]](https://i-blog.csdnimg.cn/direct/8ee68f0ddd3046829939e28f3be10ad2.png)
![![[Pasted image 20251128230849.png]]](https://i-blog.csdnimg.cn/direct/23e9601c4d004739bbe8a270e2c1dc7d.png)
查看环境是否创建成功:
conda env list
![![[Pasted image 20251128230954.png]]](https://i-blog.csdnimg.cn/direct/e9ce89d8ff41495bb3d8e85869dc4b1c.png)
可以看到在conda环境列表中已经有了DM,说明环境创建成功。
3.3 安装 Jupyter Notebook
Anaconda 自带 Jupyter,如缺失或希望重新安装,可执行:
conda install jupyter -y
启动 Jupyter:
jupyter notebook
浏览器正常打开 Jupyter 即成功。
![![[Pasted image 20251128233351.png]]](https://i-blog.csdnimg.cn/direct/c7bbabedbf0b453ca35296a8eb954555.png)
![![[Pasted image 20251128233519.png]]](https://i-blog.csdnimg.cn/direct/7e724656726b418393c1394df4de6df1.png)
3.4 安装 PyCharm
- 从 JetBrains 官网下载并安装 PyCharm。
- 安装完成后打开 PyCharm → File → Settings → Project → Python Interpreter
- 选择:
- Add Interpreter → Conda Environment → Existing Environment
- 选择路径:
<Anaconda路径>\Scripts\conda.exe
- 点击 OK,让项目使用 DM 环境的解释器。
![![[Pasted image 20251128235150.png]]](https://i-blog.csdnimg.cn/direct/b6a287b4424d492ebc267cbc09e94b32.png)
3.5 在 DM 环境安装 PyTorch 2.2.2
安装 GPU(CUDA)版本,例如 CUDA 12.1:
conda install pytorch==2.2.2 torchvision torchaudio cudatoolkit=11.8 -c pytorch -y
验证 PyTorch:
python -c "import torch; print(torch.__version__, torch.cuda.is_available())"
输出版本号说明安装成功。
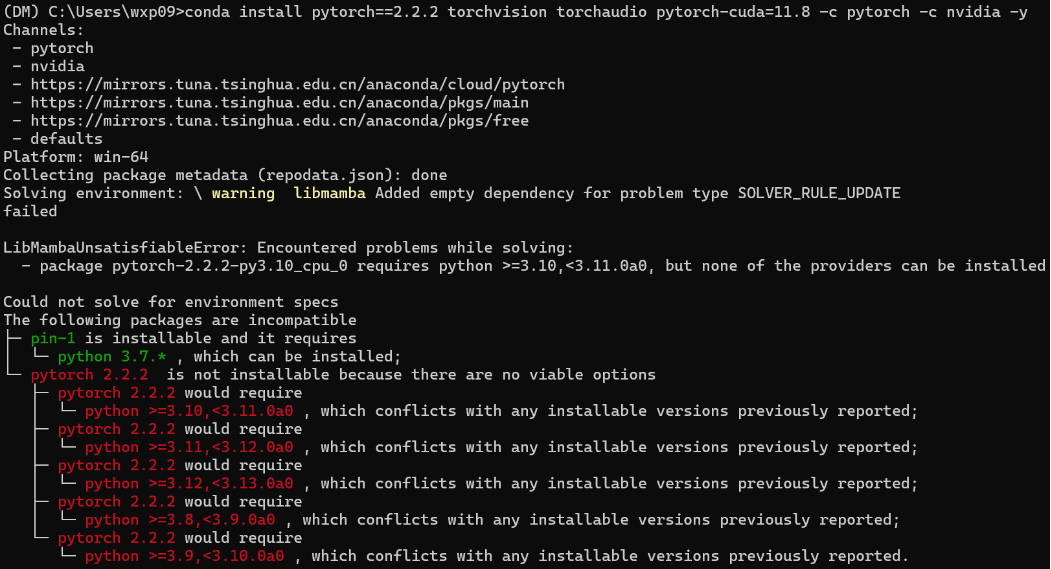
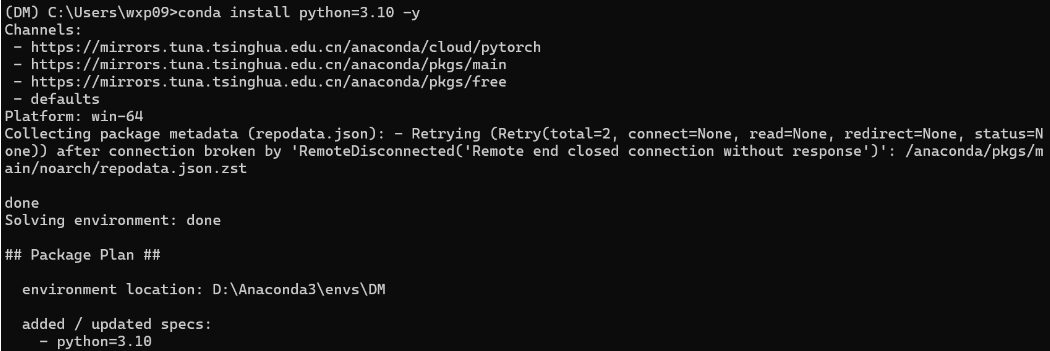

在安装 Pytorch 的时候,发现 Pytorch=2.2.2 要求 python 版本>=3.10,与我们的 python 3.7 冲突。于是只好将 python 升级成3.10.
3.6 安装常用数据科学库
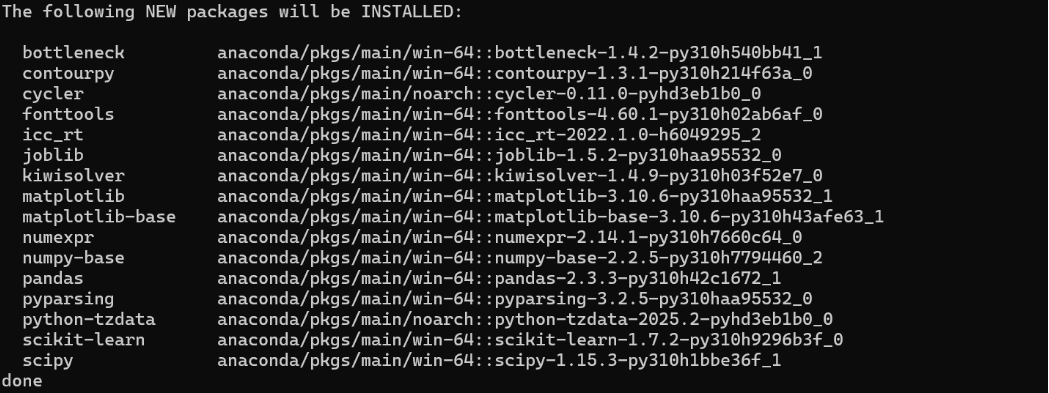
使用 conda 安装常见库
conda activate DM conda install numpy pandas matplotlib seaborn scikit-learn scipy -y
![![[Pasted image 20251129182346.png]]](https://i-blog.csdnimg.cn/direct/2af3fb9b45054012935d807fae7f0a38.png)
使用 conda-forge 安装 umap-learn
conda install -c conda-forge umap-learn -y
![![[Pasted image 20251129182748.png]]](https://i-blog.csdnimg.cn/direct/f94f38a56e4b4b038ec54620711ae66c.png)
其余库使用 pip 安装
pip install h5py mygene
![![[Pasted image 20251129184720.png]]](https://i-blog.csdnimg.cn/direct/c961e612617c4b76a397443c47b4f3b3.png)
3.7 pip 与 conda 常用命令总结
conda 常用命令
conda create -n envname python=3.X # 创建环境
conda activate envname # 激活环境
conda deactivate # 退出环境
conda install package # 安装包
conda list # 查看已安装包
conda remove -n envname --all # 删除环境
pip 常用命令
pip install package # 安装包
pip uninstall package # 卸载包
pip list # 查看已安装包
pip install -U package # 更新包
4. 实验心得
通过本次实验,我系统地完成了在 Windows 平台下搭建 Python 数据挖掘环境的全过程,包括 Anaconda 安装、虚拟环境管理、Jupyter Notebook 与 PyCharm 的使用,以及 PyTorch 等关键库的安装配置。在实践中我深刻感受到虚拟环境的重要性,它能够避免不同项目之间的依赖冲突,使得实验环境更加稳定可控。同时,通过手动安装多个常用库,我进一步理解了 conda 与 pip 的区别,以及在不同场景下如何选择更合适的包管理方式。
在配置 PyTorch 的过程中,我也认识到 CPU/GPU 版本的差异以及 CUDA 兼容性的重要性,这对后续深度学习相关实验非常关键。整体而言,本次实验不仅提高了我对 Python 科学计算环境的搭建能力,也让我对数据挖掘实验中常见工具链的结构有了更清晰的认识,为后续的建模、训练和分析提供了坚实基础。





















 1662
1662

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








