首先,需要安装nnUNet v2,注意2023年nnUnet更新了V2版本,做了许多改动。
1、数据集准备
下载百度飞桨的公共数据集 – Kits19肾脏肿瘤分割 - 飞桨AI Studio (baidu.com)
2、数据集结构化处理
在下载好数据以后,对数据进行处理。首先,先新建几个文件夹。
在nnUNet文件夹下,新建dataset。
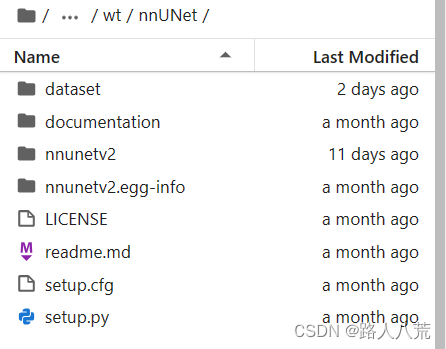
然后在dataset中,新建四个文件夹如下图所示,其中后三个用来存储nnunet数据和结果。dataset_conversion用来存储过程代码。

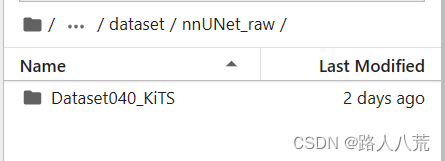
在nnUNet_raw文件中,新建文件夹Dataset040_KiTS。注意此处必须为Dataset,因为nnunetV1版本是task,v2改为了Dataset。然后在其中存放结构化处理后的数据。
然后在dataset_conversion文件中,新建一个py文件,用来生成对KiTS数据集做出描述的json文件dataset。(此处我还将其命名为Dataset040_KiTS)

代码内容如下:(此处参考博客nnUnet肾脏肿瘤分割实战(KiTS19)_宁远x的博客-CSDN博客,并做了一些改动)
import os
import json
import shutil
def save_json(obj, file, indent=4, sort_keys=True):
with open(file, 'w') as f:
json.dump(obj, f, sort_keys=sort_keys, indent=indent)
def maybe_mkdir_p(directory):
directory = os.path.abspath(directory)
splits = directory.split("/")[1:]
for i in range(0, len(splits)):
if not os.path.isdir(os.path.join("/", *splits[:i + 1])):
try:
os.mkdir(os.path.join("/", *splits[:i + 1]))
except FileExistsError:
# this can sometimes happen when two jobs try to create the same directory at the same time,
# especially on network drives.
print("WARNING: Folder %s already existed and does not need to be created" % directory)
def subdirs(folder, join=True, prefix=None, suffix=None, sort=True):
if join:
l = os.path.join
else:
l = lambda x, y: y
res = [l(folder, i) for i in os.listdir(folder) if os.path.isdir(os.path.join(folder, i))
and (prefix is None or i.startswith(prefix))
and (suffix is None or i.endswith(suffix))]
if sort:
res.sort()
return res
base = "/root/data/wt/data/KiTS19/origin" # 原始数据集路径
out = "/root/data/wt/nnUNet/dataset/nnUNet_raw/Dataset040_KiTS" # 结构化数据集目录
cases = subdirs(base, join=False)
maybe_mkdir_p(out)
maybe_mkdir_p(os.path.join(out, "imagesTr"))
maybe_mkdir_p(os.path.join(out, "imagesTs"))
maybe_mkdir_p(os.path.join(out, "labelsTr"))
for c in cases:
case_id = int(c.split("_")[-1])
if case_id < 210:
shutil.copy(os.path.join(base, c, "imaging.nii.gz"), os.path.join(out, "imagesTr", c + "_0000.nii.gz"))
shutil.copy(os.path.join(base, c, "segmentation.nii.gz"), os.path.join(out, "labelsTr", c + ".nii.gz"))
else:
shutil.copy(os.path.join(base, c, "imaging.nii.gz"), os.path.join(out, "imagesTs", c + "_0000.nii.gz"))
json_dict = {}
"""
name: 数据集名字
dexcription: 对数据集的描述
modality: 模态,0表示CT数据,1表示MR数据。nnU-Net会根据不同模态进行不同的预处理(nnunet-v2版本改为channel_names)
labels: label中,不同的数值代表的类别(v1版本和v2版本的键值对刚好是反过来的)
file_ending: nnunet v2新加的
numTraining: 训练集数量
numTest: 测试集数量
training: 训练集的image 和 label 地址对
test: 只包含测试集的image. 这里跟Training不一样
"""
json_dict['name'] = "KiTS"
json_dict['description'] = "kidney and kidney tumor segmentation"
json_dict['tensorImageSize'] = "4D"
json_dict['reference'] = "KiTS data for nnunet"
json_dict['licence'] = ""
json_dict['release'] = "0.0"
json_dict['channel_names'] = {
"0": "CT",
}
json_dict['labels'] = {
"background": "0",
"Kidney": "1",
"Tumor": "2"
}
json_dict['numTraining'] = len(cases) # 应该是210例
json_dict['file_ending'] = ".nii.gz"
json_dict['numTest'] = 0
json_dict['training'] = [{'image': "./imagesTr/%s.nii.gz" % i, "label": "./labelsTr/%s.nii.gz" % i} for i in cases]
#json_dict['test'] = []
save_json(json_dict, os.path.join(out, "dataset.json"))
结构化处理的结果是在nnUNet_raw/Dataset040_KiTS文件下,生成四个文件。

分别是,训练数据(应该是210条),测试数据(90),训练标签(210),数据说明。




3、设置数据路径
命令行,确保在nnunet激活环境下,输入vim ~/.bashrc,然后点击键盘insert开始插入,在bashrc文末,添加如下三行代码。然后按Esc键,输入:wq,就可以保存退出。
vim ~/.bashrc'''
说明,这里是路径是你自己的路径,就是上一步创建的三个文件夹的路径(这部分说明不需要写进去,只需要以下三行代码)
'''
export nnUNet_raw="/root/data/wt/nnUNet/dataset/nnUNet_raw"
export nnUNet_preprocessed="/root/data/wt/nnUNet/dataset/nnUNet_preprocessed"
export nnUNet_results="/root/data/wt/nnUNet/dataset/nnUNet_trained_models"
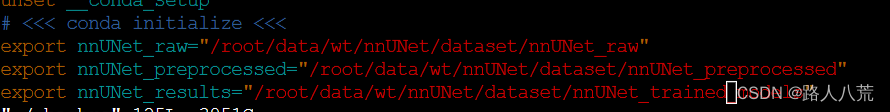
然后命令行输入source ~/.bashrc,确保激活路径。
source ~/.bashrc重点:然后分别键入三个echo $nnUNet_results,验证是否可以识别。不能识别,后续无法进行数据预处理。

最好三个都验证一下,避免后续出现问题。
4、数据集预处理
输入下面这行命令:
nnUNetv2_plan_and_preprocess -d 040 --verify_dataset_integrity
新旧版本不同,有的此处为t,有的为d,新版本应该用d,用t会发现显示没有此命令。
040表示KiTS分割任务在nnunet中,为第40个任务,代号040。
注意:可能会出现如下错误,不识别背景标签0。原因是v1和v2版本不同,dataset.json文件做出了改动。具体解决办法,见我的另一篇博客RuntimeError: Background label not declared (remeber that this should be label 0!)_路人八荒的博客-CSDN博客
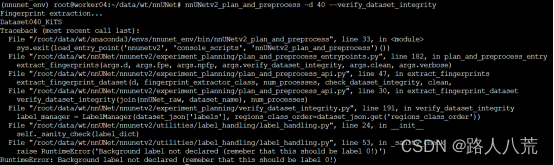
除了上述错误,可能由于V1和V2的区别,有很多错误,具体区别见我的博客:
nnUNet v2和v1版本在dataset.json的区别(KiTS19数据集)_路人八荒的博客-CSDN博客
预处理结果会在nnUNet_preprocessed文件夹下生成一个Dataset040_KiTS文件夹,里边会包含如下几个文件:
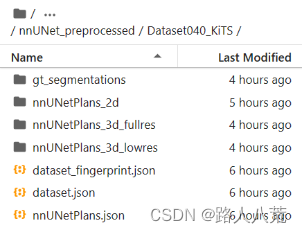
预处理结果分析:
gt_segmentations为标签
dataset.json就是上一步数据结构化处理生成的






















 3744
3744











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








