感谢阅读腾讯 AI Lab 微信号第 181 篇文章。本文介绍腾讯 AI Lab 3 篇蛋白质组论文相继入选国际顶级学术期刊,分别在蛋白质组的检测、分析以及探索发现方面提出全新的研究方案。
只有蛋白质组才能从根本上阐释生命。
3 月 20 日,腾讯 AI Lab 宣布 3 篇蛋白质组论文相继入选国际顶级学术期刊,论文分别在蛋白质组的检测、分析以及探索发现方面提出全新的研究方案,为人类从根本上阐释生命提供重要技术参考。
SPDB: a comprehensive resource and knowledgebase for proteomic data at the single-cell resolution
入选生物信息学领域数据库方面专业期刊《Nucleic Acids Research》
链接:https://academic.oup.com/nar/article/52/D1/D562/7416372
——————
A Versatile Deep Graph Contrastive Learning Framework for Single-cell Proteomics Embedding
入选《Nature》旗下方法学专业期刊《Nature Methods》
链接:https://www.nature.com/articles/s41592-024-02214-9
——————
Deep domain adversarial neural network for the deconvolution of cell type mixtures in tissue proteome profiling
入选《Nature》旗下机器学习专业期刊《Nature Machine Intelligence》
链接:https://www.nature.com/articles/s42256-023-00737-y
科学界曾经认为,只要绘制出人类基因组序列图,就能了解疾病的根源,但事实并非如此。相同的基因往往有不同的表达,比如,人体不同组织器官的基因组是一样的,但是各个组织器官的蛋白质组不完全一样。人和鼠的基因组的差别仅为 1%,但是其形态、性状差别非常大,这就是蛋白质组不一样的体现。
中国科学院院士贺福初有一个比喻:基因组和蛋白质组的关系就像词典与文章、元素表与化工厂。基因组学中微小的差异,在蛋白质组学中可以被千倍甚至近万倍地放大。因此,要真正阐释生命,必须从蛋白质组中寻找答案。
蛋白质组学是指大规模地对蛋白质的表达水平、翻译后修饰、蛋白质相互作用等进行研究。针对蛋白质组的研究不仅可以全景式地揭示生命活动的分子本质,还能阐明生命在生理或病理条件下的变化机制。蛋白组学测序技术发展迅速,相应的数据分析计算方法也方兴未艾。对此腾讯 AI Lab 从数据库、AI 建模和 AI 辅助临床分析三个角度,通过 AI 技术助力蛋白组学研究。
首次推出世界数据量最大
单细胞蛋白组数据库
目前针对蛋白质组学的研究已经深入到单细胞级别,虽然单细胞蛋白质组学能够直接在单细胞水平上测定蛋白质含量,提供了超越单纯从转录组分析所能推断的关于细胞表型的宝贵洞见。然而,缺乏足够的大规模集成数据库,阻碍了研究人员获取和探索单细胞蛋白质组数据,阻碍了这一领域的进步。
为了填补这一不足,腾讯 AI Lab 提供了一个综合性的数据库,即单细胞蛋白质组数据库 SPDB(https://scproteomicsdb.com),该数据库详细展示了单细胞蛋白质组数据,当前版本包括 133 个基于抗体的单细胞蛋白质组数据集,涉及超过 3 亿个细胞和超过 800 个标记/表面蛋白质,以及 10 个基于质谱的单细胞蛋白质组数据集,涉及超过 4000 个细胞和超过 7000 种蛋白质。
SPDB 目前已经成为世界上数据量最大,覆盖技术和数据集最为广泛的单细胞蛋白组知识库,标准化的数据处理和用户友好的网络界面,基于数据集级别和蛋白质级别的数据搜索和探索功能,为人类探索蛋白质组学的详细洞见,提供了重要的数据参考。
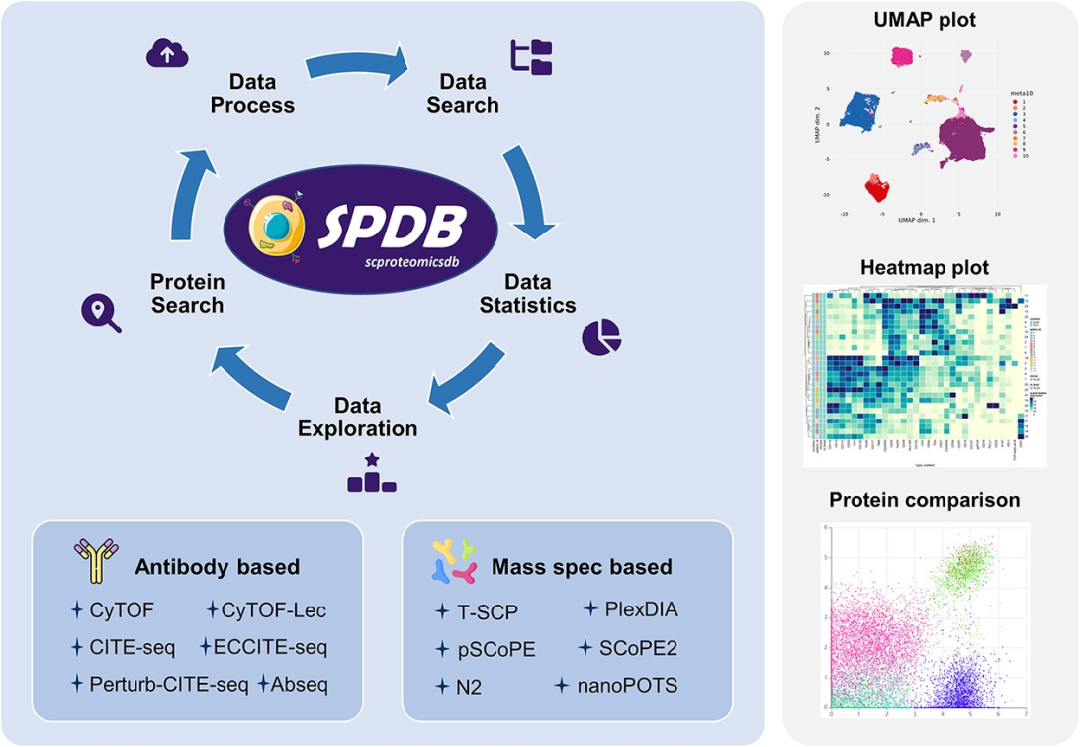
图片来源:https://academic.oup.com/nar/article/52/D1/D562/7416372
该技术可以供生命科学工作者和医生对世界上最全的单细胞蛋白组进行数据探索,也可以供生物信息和 AI 科学家对单细胞蛋白组提供更多的算法和工具,从而促进该领域的科学新发现和工具开发。本论文《SPDB: a comprehensive resource and knowledgebase for proteomic data at the single-cell resolution》也因此入选生物信息学领域数据库方面专业期刊《Nucleic Acids Research》。
全新的多功能算法
提升单细胞蛋白质组测序分析准确率
和基因检测类似,要研究蛋白质组,需要精准的检测技术。目前蛋白组学检测技术已经发展至单细胞分辨率。单细胞蛋白质组学测序技术,为揭示细胞中蛋白质-蛋白质相互作用、翻译后修饰和蛋白质形态动态的研究带来了新的启示。然而,肽段定量的不确定性、数据缺失、严重的批次效应和高噪声,阻碍了单细胞蛋白质组数据的准确分析和使用。
腾讯 AI Lab 研究人员提出了一种新颖的多功能算法 scPROTEIN,它由基于多任务异方差回归模型的肽段不确定性估计,以及基于图对比学习设计的单细胞蛋白质组数据分析的细胞嵌入学习组成。scPROTEIN 在一个统一的框架中估计了肽段定量的不确定性,去噪了蛋白质数据,消除了批次效应,并编码了单细胞蛋白质组特异性嵌入。该方法为首个针对单细胞蛋白组表征的框架,创造性解决了单细胞蛋白组学数据的特殊挑战。
随着单细胞蛋白组检测技术在生命科学和精准医疗领域的普及,该方法可以作为数据处理中重要的步骤,为基于单细胞蛋白组的肿瘤发生发展机制研究、药物靶点发现和肿瘤早筛和微环境研究提供重要的AI辅助作用。基于这项技术的《A Versatile Deep Graph Contrastive Learning Framework for Single-cell Proteomics Embedding》论文也入选《Nature》旗下方法学专业期刊《Nature Methods》。
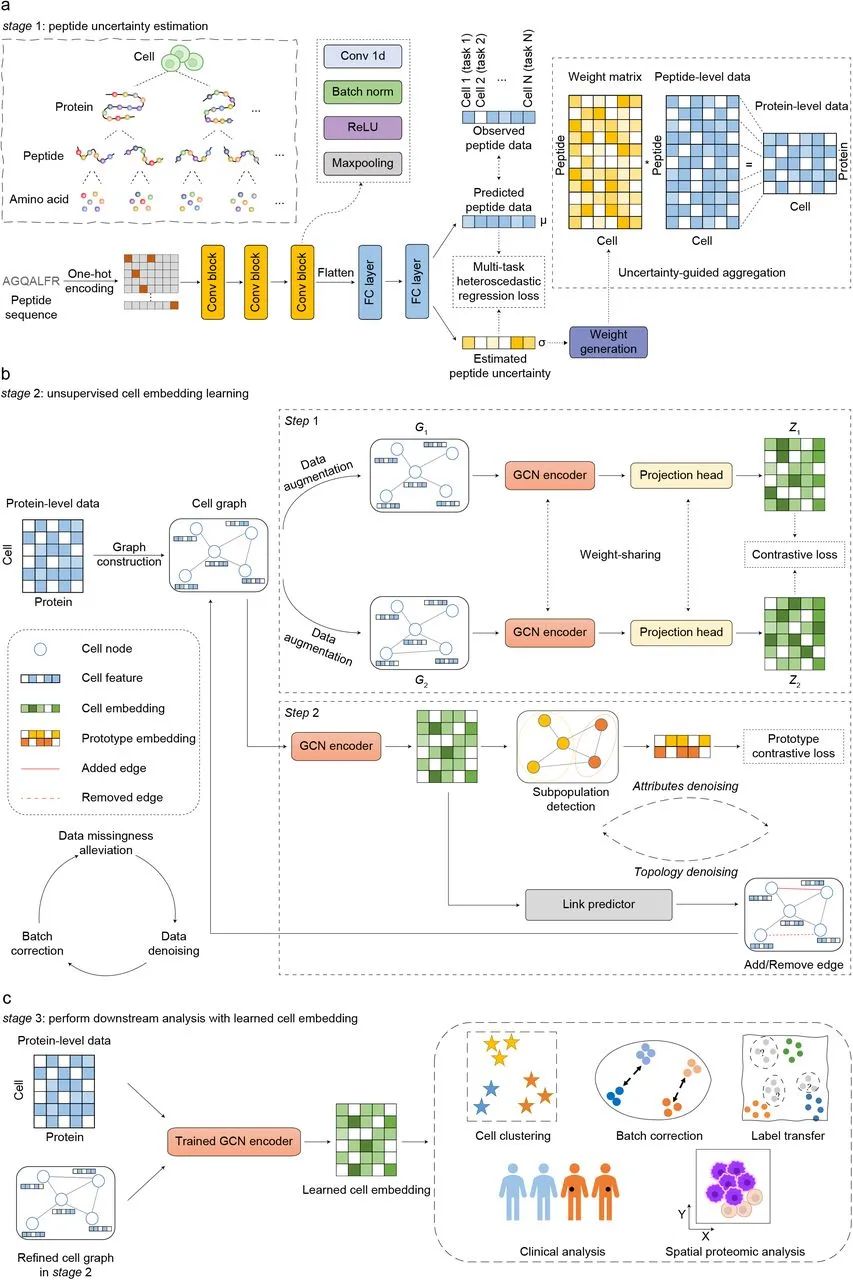
图片来源:https://www.nature.com/articles/s41592-024-02214-9
全新的反卷积方法
助力肿瘤辅诊和预后分析
同样入选《Nature》旗下专业期刊《Nature Machine Intelligence》的还有《Deep domain adversarial neural network for the deconvolution of cell type mixtures in tissue proteome profiling》,这篇论文从更为宏观的角度来分析特定组织中不同细胞类型的比例。例如,常用于分析肿瘤组织样本中不同细胞类型,该方法已经广泛适用于肿瘤微环境解释和临床诊断/分类等领域。
过去蛋白组学研究产生大量的数据,这些数据是组织中所有细胞的平均值,难以直接反映不同类型细胞的比例,而细胞比例对于肿瘤微环境分析较为重要。对此,腾讯 AI Lab 研究人员以单细胞蛋白组为参考,基于 AI 方法对现有的组织蛋白组数据进行反卷积(一种数学方法),从组织蛋白质组数据中挖掘出细胞类型比例这一新信息,应用大量现有的蛋白组数据,获取其中的肿瘤微环境信息,助力肿瘤辅诊和预后分析。
这种专为组织蛋白质组数据设计的基于深度学习的反卷积方法(scpDeconv),使用自动编码器利用整体蛋白质组数据的信息,从而提高单细胞蛋白质组数据的质量,并采用领域对抗模型来连接单细胞和整体数据分布,将标签从单细胞数据迁移到整体数据。大量实验验证了 scpDeconv 在反卷积来自不同物种/来源和不同蛋白质组技术产生的蛋白质组数据方面的性能。该方法是首个针对蛋白组进行反卷积的算法,解决了蛋白组独特的数据挑战。
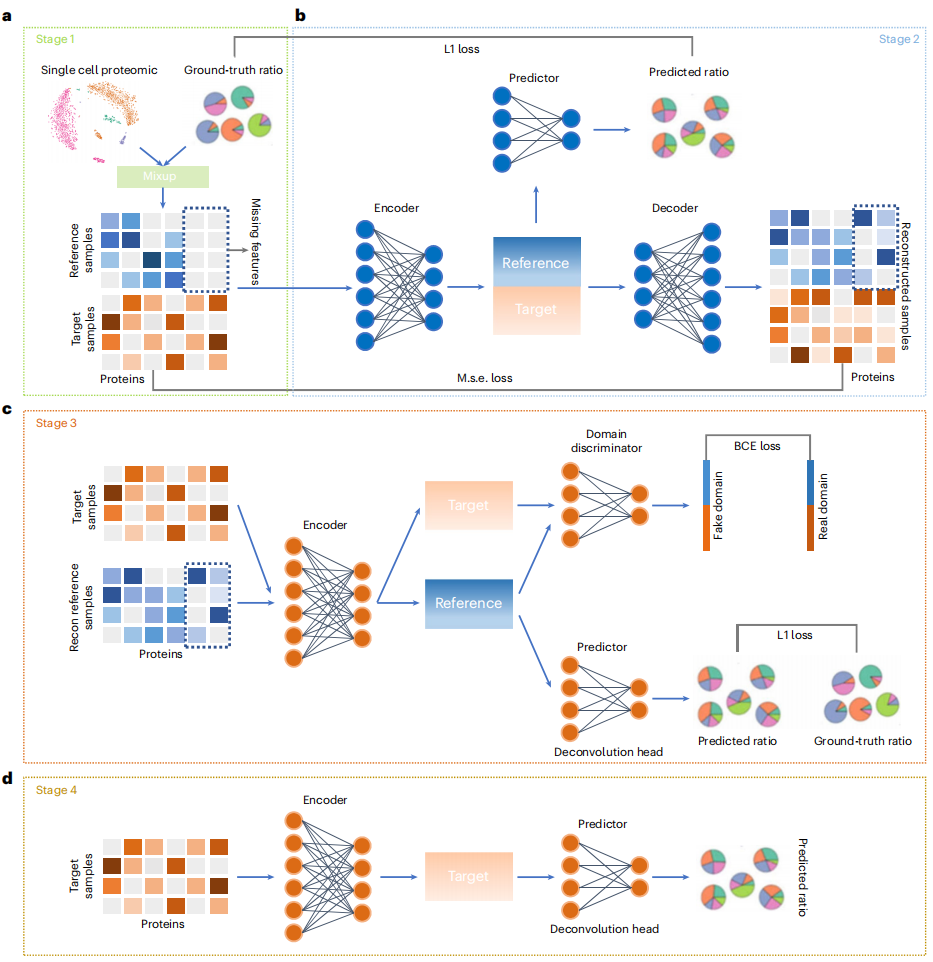
图片来源:https://www.nature.com/articles/s42256-023-00737-y
上述三篇论文分别展示了腾讯 AI Lab 为解决蛋白组学中数据库、AI 建模和AI辅助临床分析问题而进行的一系列探索,为高效精准分析蛋白组数据、理解肿瘤微环境和发现生物学新机制打下坚实基础。
腾讯 AI Lab 在 AI for Science 特别是生命科学领域深耕数年,具有丰富的知识和技术积累,研究领域包括单细胞多组学、蛋白质结构设计、蛋白质折叠、AI 制药、空间组学和免疫组库等,已发表 scBERT 和猕猴大脑图谱等研究成果。腾讯 AI Lab 成立于 2016 年,实验室强调研究与应用并重发展,以「学术有影响,工业有产出」为目标,基于扎实的学术研究,将 AI 能力运用在游戏、内容、虚拟人以及医疗、医药、基因计算等多个场景中。
参考文献:
[1] Wang F, Liu C, Li J, et al. SPDB: a comprehensive resource and knowledgebase for proteomic data at the single-cell resolution[J]. Nucleic Acids Research, 2024, 52(D1): D562-D571.
[2] Wang F, Yang F, Huang L, et al. Deep domain adversarial neural network for the deconvolution of cell type mixtures in tissue proteome profiling[J]. Nature Machine Intelligence, 2023, 5(11): 1236-1249.
[3] Li W, Yang F, Wang F, et al. A Versatile Deep Graph Contrastive Learning Framework for Single-cell Proteomics Embedding[J]. Nature Methods, 2024.

* 欢迎转载,请注明来自腾讯 AI Lab 微信(tencent_ailab)




















 44
44











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








