【准备工作】
准备软件——plink1.9,admixture32,tassel
准备源文件——vcf格式文件,表型数据文件
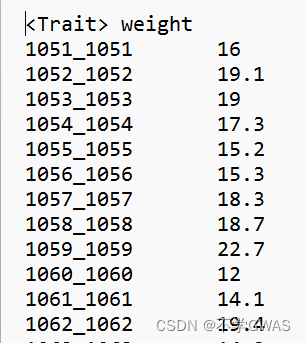
表型数据文件格式
【plink质控与筛选】
plink质控过滤
plink --vcf ABC.vcf -maf 0.05 --geno 0.1 --recode vcf-iid -const-fid --out ABC.filter --chr-set 24
其中“--chr-set ”为染色体条数多于23条时,自主设置染色体条数用,因为plink是为人类染色体设计,所以默认为23条,在未添加此参数时,识别23条以上染色体时会出错,这里我研究的样本是24条染色体的,故设置为24,下同。
plink LD筛选
plink --vcf G.filter.vcf --indep-pairwise 50 10 0.2 --out G2 --chr-set 24
plink 筛选结果提取
plink --vcf G.filter.vcf --const-fid --make-bed --extract G2.prune.in --chr-set 24 --out G3.in
plink 转换成admixture格式
plink --bfile G3.in --recode 12 --chr-set 24 --out G4.in
 得到map和ped文件,进入下一步。
得到map和ped文件,进入下一步。
【tassel计算混合线性模型】
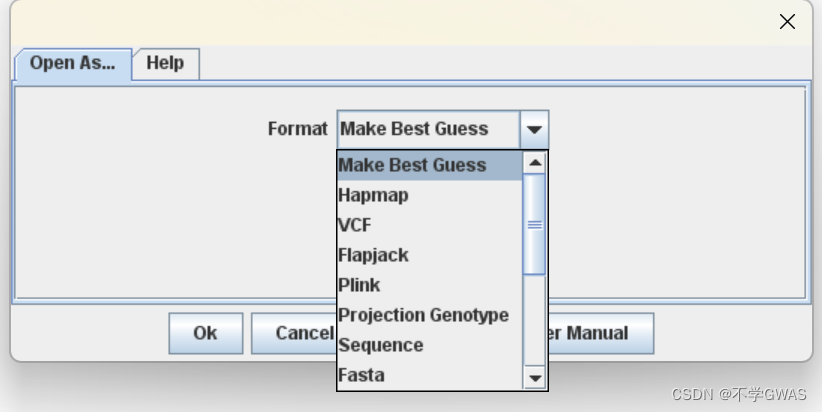
在Open As中选择“plink”格式,打开之前生成的map与ped文件,即经过质控过滤的基因型文件。
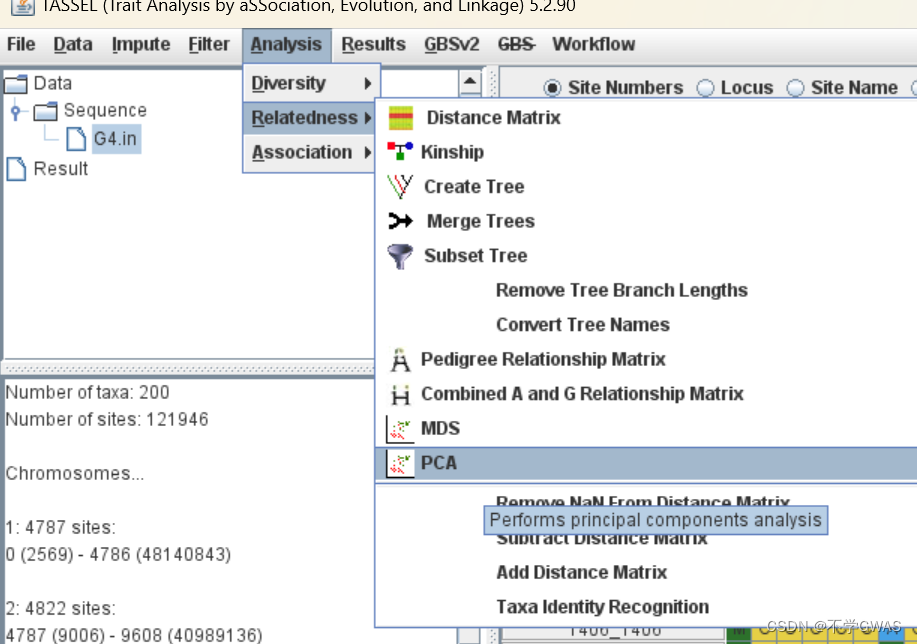 选择此项,进行pca分析。
选择此项,进行pca分析。
导入表型文件
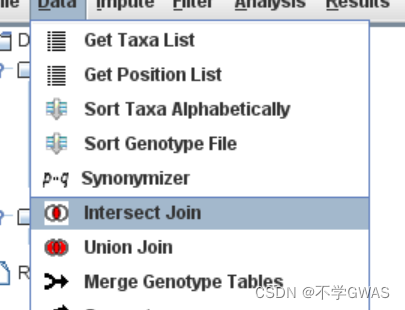
按ctrl选中表型文件,基因型文件,pca计算文件合并。
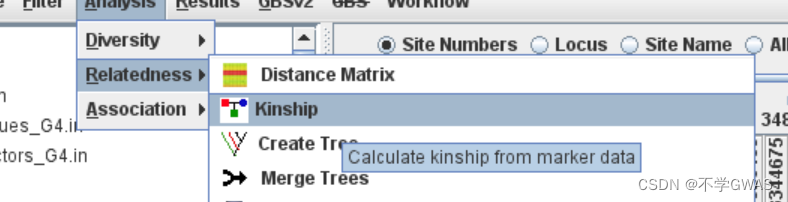
选择基因型文件,计算kinship。
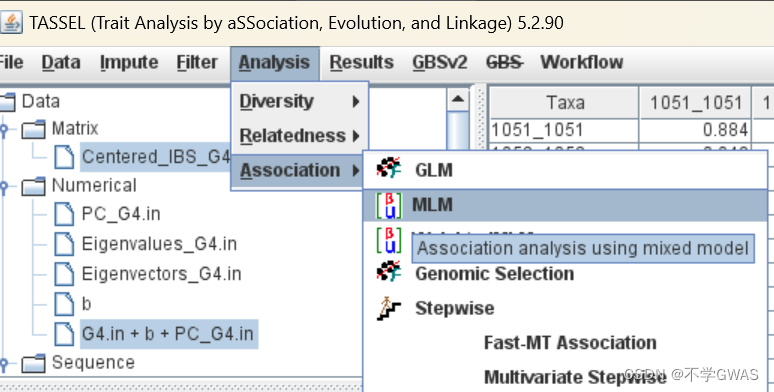 选择计算kinship得出的文件与刚才三合一的文件,计算MLM模型
选择计算kinship得出的文件与刚才三合一的文件,计算MLM模型
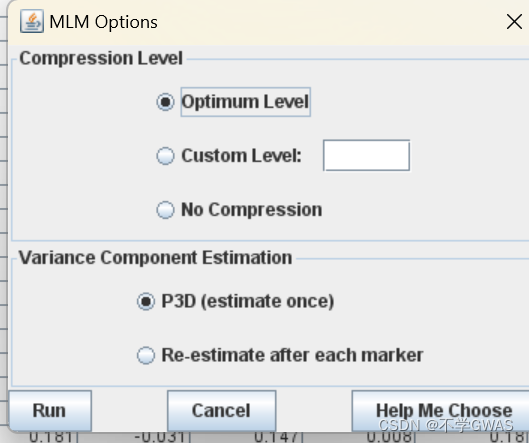
这里可以所选择的样本数据大小来选择压缩等级和不同的分析方法。
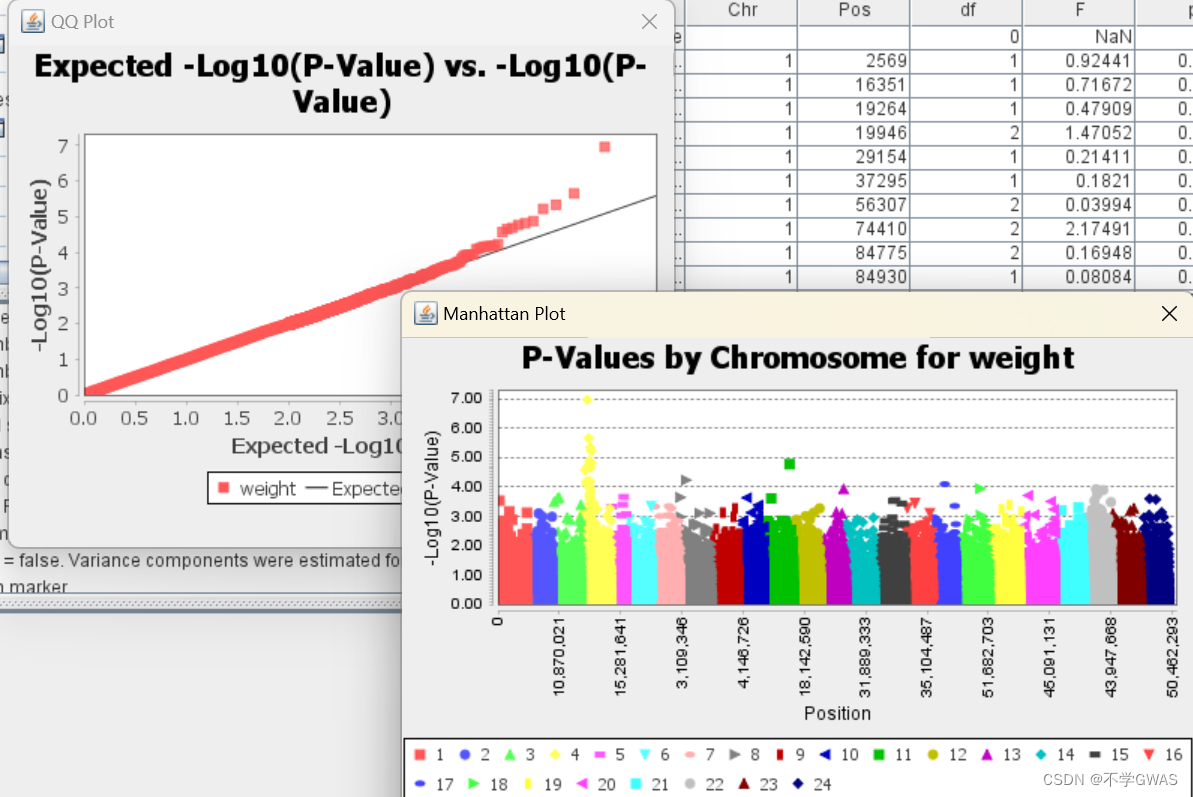
在计算完毕后,选择mlm混合线性模型,即可Results中选择生成不同的图片





















 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








