目录
1 概述
浪潮信息KOS是浪潮信息基于Linux Kernel、OpenAnolis等开源技术自主研发的一款服务器操作系统,支持x86、ARM等主流架构处理器,性能和稳定性居于行业领先地位,具备成熟的 CentOS 迁移和替换能力,可满足云计算、大数据、分布式存储、人工智能、边缘计算等应用场景需求。详细介绍见官网链接https://zh.ieisystem.com/product/kos/9453.html。
FASTA(Fast-All)是一套用于比对生物学序列的软件工具,最初由 William R. Pearson 和 David J. Lipman 在 1988 年首次发布。FASTA 旨在快速搜索序列数据库以寻找与给定查询序列相似的序列。FASTA 的核心原理是通过计算序列之间的局部相似性来找到匹配。它使用散列和滑动窗口技术来识别相似的、非连续的子序列。FASTA 被广泛应用于生物信息学领域,用于比对蛋白质序列和核酸序列。基本用法涉及提供一个查询序列,然后搜索一个或多个目标数据库,找到相似的序列。提供了多种参数和算法选项,允许用户根据实际需求进行调整。其输出通常包含相似性得分、匹配的位置以及其他统计信息。详细介绍见官网链接https://fasta.bioch.virginia.edu/。
2 安装环境
2.1 操作系统环境
版本信息:KeyarchOS 5.9
硬件平台:X86_64

2.2 工具版本
FASTA-36.3.8i
https://fasta.bioch.virginia.edu/wrpearson/fasta/fasta36/
3 安装与测试
3.1 安装FASTA
在https://fasta.bioch.virginia.edu/wrpearson/fasta/fasta36/
找到所需版本,下载安装包
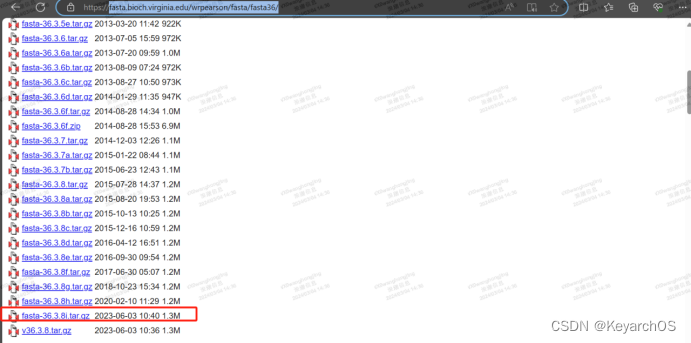
上传到服务器并解压
![]()
进入src文件夹
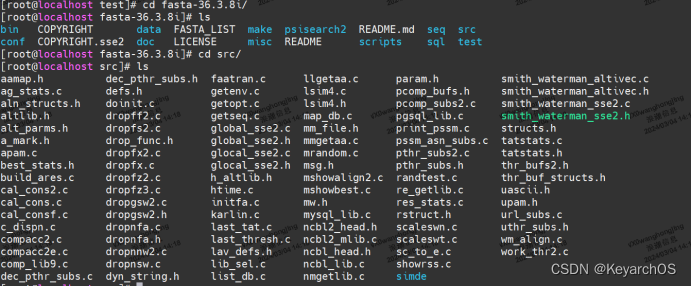
开始编译 make -f ../make/Makefile.linux_sse2 all

编译完成后进入../fasta-36.3.8i/bin文件夹后可以看到fasta36

将其添加到PATH中 export PATH=$PATH:/root/test/fasta-36.3.8i/bin
![]()
3.2 使用测试
查看fasta版本 fasta36 -v

使用seq中的测试文件进行验证fasta是否能够正常使用
在../fasta-36.3.8i路径下执行fasta36 ./seq/bovgh.seq ./seq/testdb.fa
这将使用 bovgh.seq 文件作为查询序列,testdb.fa 文件作为数据库序列进行比对
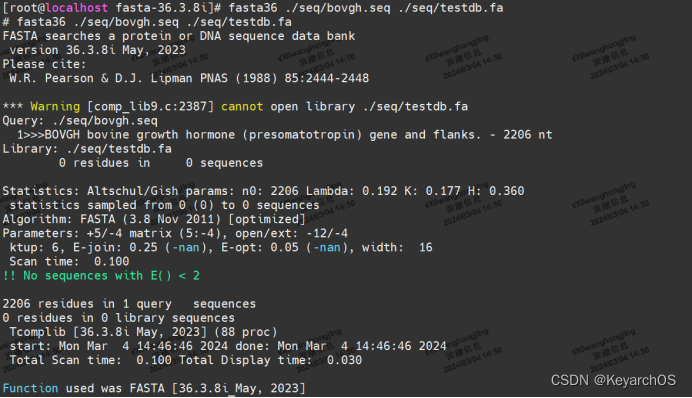
根据输出,程序成功读取了 bovgh.seq 查询序列文件。
执行fasta36 ./seq/bovgh.seq ./seq/bovprl.seq
这将使用 bovgh.seq 文件作为查询序列,bovprl.seq 文件作为数据库序列进行比对

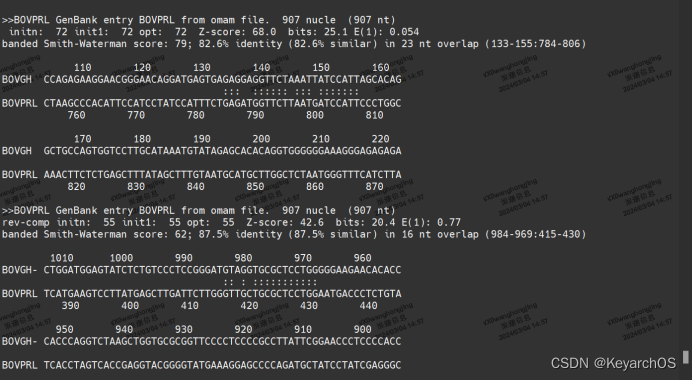
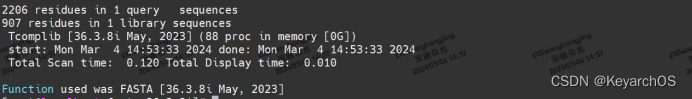
比对结果显示了相似区域,包括最佳得分、Z-score、比特分数等信息,以及相似性比对的可视化























 957
957











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








